| Главная страница | Обучение | Обо мне | Ссылки |
Семейства белковых доменов | |||
|
Практикум представлен в виде Jal-view проекта. Задание 1. Для белка антранилат-фосфорибозилтрансферазы (идентификатор в UniProt H6Q874) на сайте Pfam была построена доменная структура(см. Рис.1).
Для дальнейшей работы был выбран второй домен - a/b домен семейства гликозилтрансфераз, в которое входят антранилат-фосфорибозилтрансферазы и тимидинфосфорилазы.
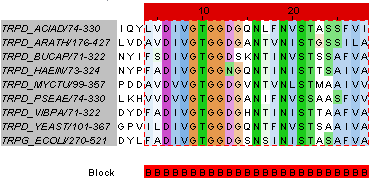
Скачанный seed семейства можно посмотреть во вкладке seed_align Jalview-проекта. Задание 2. Консенсусная последовательность всего белка сохранена в файле consensus.fasta. Для построения LOGO был найден вертикальный блок с 6 по 12 позиции (во вкладке seed_align отмечен буквой B в строке "Blocks") - см. рис. 2. Консенсус выбранного блока: consensus_block.fasta. LOGO построено с помощью сервиса http://weblogo.berkeley.edu/.
Задание 4. Сильный паттерн дает нам уверенность в том, что все последовательности, найденные по нему, являются гомологами. Слабый паттерн призван находить всех гомологов, однако помимо них возможны и ложные находки. Паттерны были построены для блока, использованного выше для создания LOGO, оно тоже было полезно для их создания. Для поиска гомологов по паттерну в БД SwissProt использовался сервис ScanProsite. Для построения сильного паттерна были удалены белки TYPH_HUMAN, TYPH_ECOLI, PDP_BACSU, так как у них в трех колонках (7-9) стоят аминокислоты, мешающие создать хороший сильный паттерн. Надо отметить, что в принципе по паттерну такого маленького блока, как выбранный, достаточно сложно найти достоверных гомологов без включения в результаты ложных находок. Сначала был произведен поиск в БД SwissProt паттерна D-[TISVA]-[AVC]-G-T-G-G, однако он оказался неудачным, так как в результаты попало много белков, очевидно не являющихся антранилат-фосфорибозилтрансферазами. Они попали в список находок по той причине, что их "блок" этого паттерна выглядел так: DTAGTGG. Среди антранилат-фосфорибозилтрансфераз тоже встречаются белки с таким участком. Это подтверждает то, что чем меньше паттерн, тем менее достоверны находки. Для того, чтобы в результате выводились только антранилат-фосфорибозилтрансферазы, паттерн был изменен на D-I-[VC]-G-T-G-G. Это помогло: были найдены антранилат-фосфорибозилтрансферазы и 2 белка, имеющих ровно такою же последовательность аминокислот в этом блоке, как и все найденные гомологи (из-за того, что выбран маленький блок, убрать их из результатов не получается). Посмотрев, какие действительно аминокислоты попадаются на этом участке у найденных антранилат-фосфорибозилтрансфераз, я убедилась, что нет необходимости включать в паттерн С в 3 позиции: все белки имели последовательность DIVGTGG. Для данного блока более сильного паттерна построить не удается. Но, удалив из seed'a все последовательности, у которых в 7-8 позиции стояло не IV и не VV, удалось сильно увеличить блок с 6 - 12 до 4 - 28 позиций (см. вкладку в проекте seed_new, отмечен буквами В, см. Рис.3).
Для него окончательный сильный паттерн выглядит так: [FLAVI]-[VLAS]-D-[IV]-V-G-T-G(2)-[DN]-[GS]-x-N-[LTIS]-[FVI]-N-[VIL]-[ST](2)-x-[SA](2)-[FI]-[VL]-[IAV]. Действительно, паттерн является сильным, так как находит только антранилат-фосфорибозилтрансферазы, а также уже появлявшиеся ранее 2 белка, имеющие последовательность, подходящую даже под этот более длинный паттерн. Вероятно, они тоже входят в рассматриваемое семейство. Слабый паттерн должен находить все антранилат-фосфорибозилтрансферазы, то есть в него как минимум должны быть включены все аминокислоты, попадающиеся в этих позициях у белков seed'a. Поэтому слабый паттерн (для исходного, маленького блока) будет выглядеть следующим образом: [DE]-x(2)-[GS]-T-G(2). В этом случае получаем 3159 находок по БД SwissProt, из которых 539 являются антранилат-фосфорибозилтрансферазами, а так же туда попали тимидинфосфорилазы, тоже входящие в данное семейство. Результат не очень хороший, однако улучшить его сложновато, так как даже в seed'e в колонках, которые в паттерне обозначены x, нет единообразия: встречаются и гидрофобные аминокислоты, и заряженные, и незаряженные полярные. Попробовав паттерн [DE]-[KITSNAV]-[HVCA]-[GS]-[TS]-G(2) (в позициях х(2) перечислив все аминокислоты, встречающиеся в seed'e), в результате было получено 882 последовательности, но среди них всего 522 антранилат-фосфорибозилтрансферазы, то есть не все семейство. Таким образом, окончательный слабый паттерн выглядит так: [DE]-x(2)-[GS]-T-G(2). | |||
| © Alexandra Boyko, 2014. Faculty of Bioengineering and Bioinformatics, MSU. | |||