| Главная страница | Обучение | Обо мне | Ссылки | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Выравнивание геномов | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
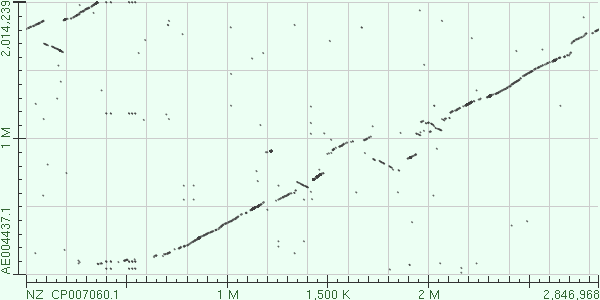
Задание 1. Для построение карты локального сходства были выбраны археи разных штаммов одного рода (вид не указан): Halobacterium sp. NRC-1 (AC AE004437.1) и Halobacterium sp. DL1 (АС NZ_CP007060.1).
Размер генома Halobacterium sp. DL1 - 2846968 bp, а Halobacterium sp. NRC-1 - 2014239 bp. Поэтому можно предположить, что на карте будут видны вставки или делеции. И действительно, на рис. 2 приведена та же карта локального сходства с обозначенными эволюционными событиями.
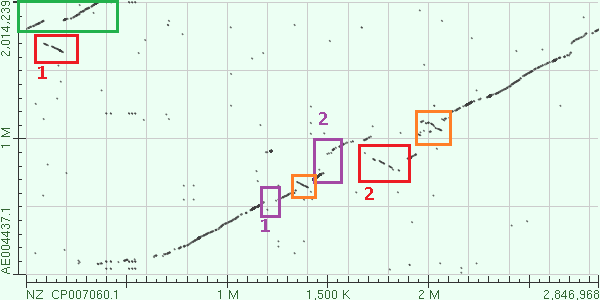
Описание карты локального сходства Прежде всего, нужно заметить, что основная часть выравнивания смещена относительно глаавной диагонали, которая была бы при выравнивании близкородственных организмов. Такое смещение объясняется тем, что последовательности имеют разные длины, относящиеся как 3:2 (DL1 : NRC). В зеленой рамке показана транслокация в геноме штамма NRC-1 относительно DL1 (примерно первые 0,4 Mb по геному DL1). Рядом с ней (ниже по геному DL1, 1.7 Mb - 1.6 Mb) расположен фрагмент выравнивания с инверсией (красная рамка). Предположительно, инверсия участка произошла после транслокации, так как участок сильно смещен к транслоцированному участку, описанному ранее. Похожая ситуация возникает на карте в районе 1,7 -1,9 Mb: на том участке произошла транслокация + инверсия. Оранжевым цветом обведены участки выравнивания, на которых произошла инверсия в геноме Halobacterium sp. NRC-1. Фиолетовым обведены участки, на которых произошла вставка или делеция. На первом участке (отмечен 1 на рис. 2) могла произойти либо делеция генома AE004437.1 (на уровне ~ 0,55 Mb) относительно генома NZ_CP007060.1, либо вставка в геноме NZ_CP007060.1 (в районе 1,2 - 1,25 Mb). На втором участке в фиолетовой рамке (отмечен 2 на рис. 2) ситуация аналогична. Установить однозначно, какое эволюционное событие произошло на этих участках (вставка или делеция) нельзя. Это можно было бы установить, посмотрев геномы предполагаемого предка, а также ближайших родственников данных штаммов. В целом про последовательности можно сказать, что они не близкородственны, однако некоторые участки родства у них есть.
Рис. 0. Золотистый стафилококк. Задание 2. Для описания геномов близкородственных бактерий были выбраны 4 штамма золотистого стафилококка (со статусом сборки "complete genome"). Самая важная информация о них приведена в Таблице 1. Золотистый стафилококк - это вид граммположительных шаровидных бактерий, вызывающий широкий спектр заболеваний, от легких инфекций до смертельно опасных. Это комменсал, живущий на коже и поверхности слизистых. Большинство штаммов устойчивы к пенициллиновым антибиотикам (благодаря наличию пенициллиназы), поэтому в борьбе с золотистым стафилококком стали применять химически модифицированный пенициллин - метициллин, однако уже сейчас есть штаммы, устойчивые и к этому виду антибиотика (например, MRSA252, Mu50, рассматриваемые в данном практикуме). Геном выбранной бактерии представлен одной кольцевой хромосомой (в некоторых случаях также присутствует плазмида). Таблица 1. Информация о выбранных штаммах Staphylococcus aureus
Сходства и различия в геномах выбранных штаммов были описаны с помощью программы NPGE. Для этого был создан файл с таблицей genomes.tsv. Параметры создания пангенома были изменены (изменение в MIN_IDENTITY = Decimal('0.377') ). Была получена информация о разных блоках генома. Информация о них приведена ниже. 1. g-блоки (global) - синтеничные блоки генома, то есть блоки с одинаковыми последовательностями опорных участков; при этом между опорными участками могут происходить совершенно любые изменения (инверсии, вставки и т.д., различающиеся у разных геномов).
G-блоки должны состоять из s-блоков, между которыми могут быть блоки остальных (любых) типов. В моей ситуации их оказалось 30 (+ 53 i-блока, которые так же упомянуты в файле blocks.gbi. Как я поняла, это тоже своего рода g-блоки, но встречающиеся не у всех 4 организмов, а только у 1-3 из них. Могу предположить, что буква i в названии блока означает "insertion" - вставку).
Выравнивание g-блоков представлено ниже на Рис. 1.
Анализируя данное выравнивание, в частности, можно заметить, что у первого штамма (ED133) есть 2 инвертированных блока (стрелочки, указывающие направление последовательности, обращены назад): g4x116 и g4x10909. Есть также 18 блоков, которые у всех 4 штаммов расположены в одном месте последовательностей. В штамме MRSA252 произошла транслокация начального g-блока g4x296469 и вставка фрагмента i1x128, а у штамма JH1 - транслокация конечного блока выравнивания g4x124 в самое начало. Хорошо прослеживаются и другие эволюционные изменения. 2. s-блоки (stem) - блоки, присутствующие во всех исследуемых геномах, так называемое "ядро геномов". Это наиболее стабильные блоки выравнивания последовательностей. Общая информация по таким стабильным блокам (взята из файла pangenome.info): 3. r-блоки (repeats) - блоки с повторяющимися последовательностями (условие - встречаются минимум 2 раза хотя бы в одном геноме). Информация по ним бралась из файла pangenome.bi, для удобства была переведена в таблицу blocks.xlsx. В принципе, в этой таблице есть информация по всем блоках пангенома (каждый тип блока описан на отдельном листе). Общая информация по r-блокам: Наиболее часто встречающийся r-блок - r46x118. Как видно из его идентификатора, он встречается 46 раз во всех 4 геномах. Еще один интересный блок повторов - наиболее длинный, r5x8590. Информация о них приведена ниже в Таблице 2.
Таблица 2. Информация о двух r-блоках из пангенома для 4 штаммов Staphylococcus aureus.
Можно заметить, что блок r5x8590 весьма длинный и при этом имеет высокий процент консервативности (идентичность 81,54%), а встречается он редко - 2 раза в геноме ED133 и по 1 разу в геномах других штаммов.
Поэтому, возможно, имеет смысл искать в нем какой-то кодирующий участок.
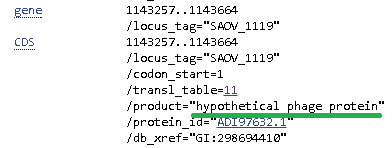
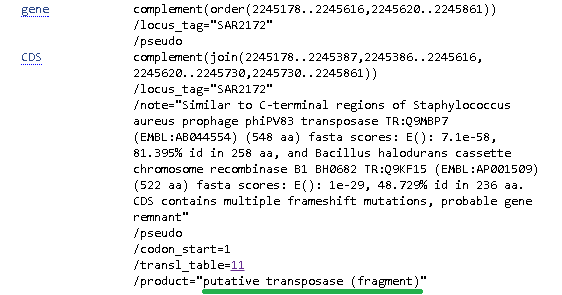
И действительно, в NPGE для штаммов MRSA и ED133 был найден ген белка бактериофага. Интересно, что в повторе в геноме штамма MRSA252 аннотирована транспозаза. Это подтверждается бластом. См. рис. 2.
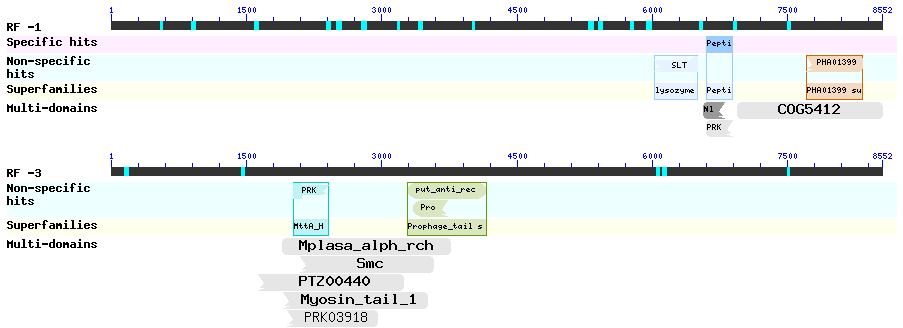
Рис. 2a. Аннотация генов внутри повтора r5x8590 в геноме Staphylococcus aureus subsp. aureus ED133. Рис. 2b. Аннотация генов внутри повтора r5x8590 в геноме Staphylococcus aureus subsp. aureus MRSA252, первый фрагмент. Рис. 2c. Аннотация генов внутри повтора r5x8590 в геноме Staphylococcus aureus subsp. aureus MRSA252, второй фрагмент. NCBI предоставил структуру белковых доменов для данного участка (рис. 3). Эта структура доказывает, что в данном блоке присутствуют белки фага. В остальных штаммах этот повтор содержит "гипотетический белок". Таким образом, с большой долей уверенности можно предположить, что в это место генома в какой-то момент эволюции встроился геном бактериофага.
Учитывая факт, что у штамме MRSA он аннотирован как транспозаза, а в геноме ED133 присутствует 2 таких r-блока, можно, вероятно, предположить, что этот фаг давным-давно стал транспозоном.
Рис. 3. Консервативные домены в r5x8590. Присутствуют домены, характерные для фаговых белков. 4. h-блоки (hemi) - "полустабильные" блоки, встречающиеся у части геномов из пангенома. На них были исследованы делеции.
Общая информация по h-блокам данного пангенома:
В Таблице 3 приведена информация по двум выбранным h-блокам.
Таблица 3. Информация о двух h-блоках пангенома
Ниже (для удобства, в виде Таблицы 4) представлена информация о том, какие белки кодируются в этих h-блоках.
Таблица 4. Информация о белках, закодированных в выбранных h-блоках пангенома
(Фактически, белки, описанные для одного блока у разных штаммов, являются одинаковыми - это прослеживалось по одинаковым длинам кодирующих их генов.)
Таким образом, можно заключить, что гены белков, описанных в Таблице 4, у штаммов, для которых стоит прочерк, были удалены из этих геномов.
5. u-блоки (unique) - блоки, встречающиеся в пангеноме лишь 1 раз. Являются, пожалуй, наиболее интересными для поиска различий в геномах разных штаммов одного вида бактерий, так как несут в себе последовательности, уникальные для каждого штамма.
Общая информация о u-блоках:
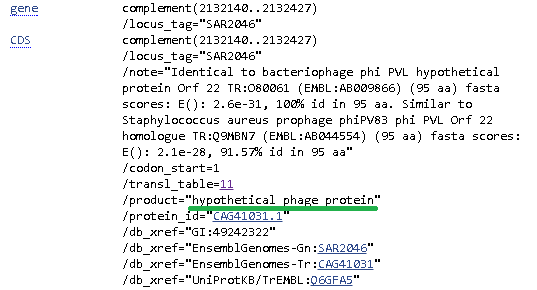
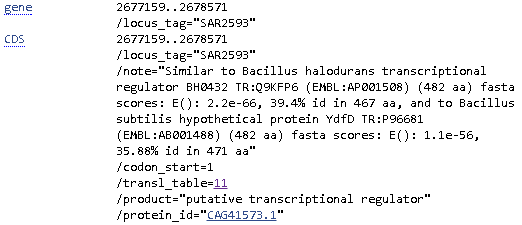
Для аннотирования был выбран блок u1x429. Он присутствует только у штамма MRSA252 и составляет 429 пн. В нем содержится ген, аннотированный для данного штамма как предполагаемый регулятор транскрипции (рис. 4).
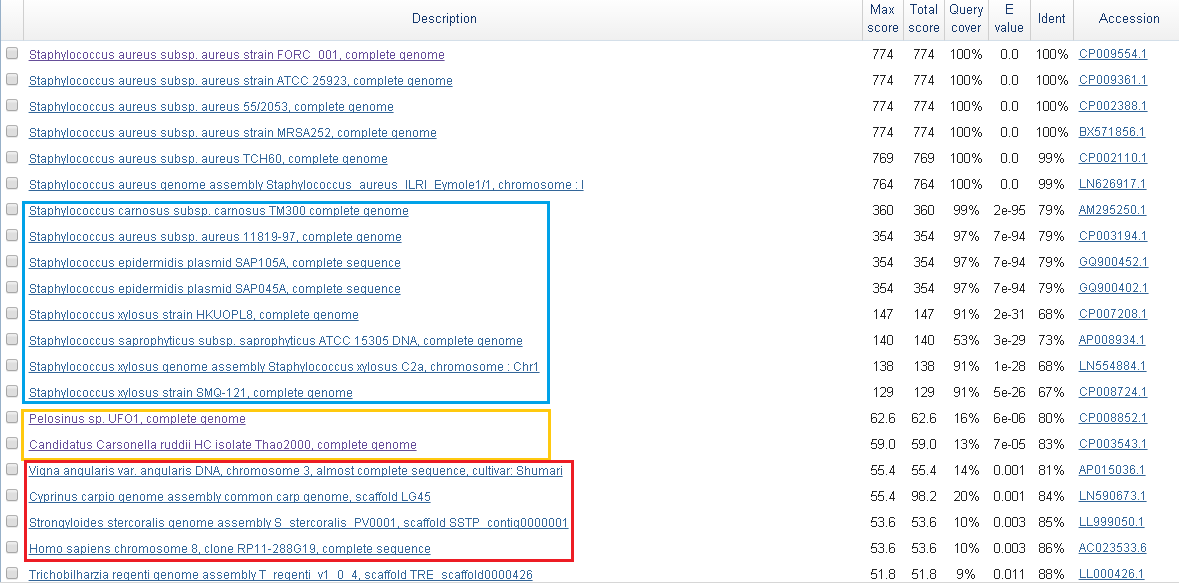
Рис. 4. Аннотация белка SAR2593 в геноме штамма MRSA252. Был запущен blastn для последовательности этого блока. Результаты показаны на рис. 5. Всего бласт выдал 895 находок, однако перед его запуском не был поставлен пороговый E-value, так что преимущественно в эти находки попали последовательности, имеющие очень низкое сходство с искомой.
Рис. 5. Результаты поиска с помощью blastn посл-ти u1x429 из генома штамма MRSA252. Показаны лучшие находки. Синим обведены находки из того же рода бактерий (Staphylocccus). Желтым обведены находки из бактерий, далеких по филогенетическому древу от изучаемого штамма. Красным обведены находки с низким качеством. Среди находок есть много бактерий других видов, в том числе протеобактерий (изучаемые стафилококки относятся к фирмикутам), так что можно предположить горизонтальный перенос генов. Так же есть находки, резко отличающиеся по query cover, e-value и другим показателям blast от предыдущих. Это находки из эукариотических организмов (карп, бобовое и даже хромосома человека). Очевидно, горизонтальный перенос генов от этих организмов весьма маловероятен.
Несоответствия в аннотации генов
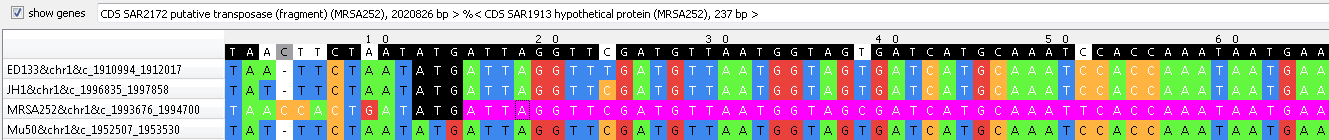
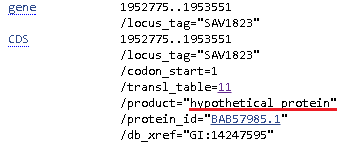
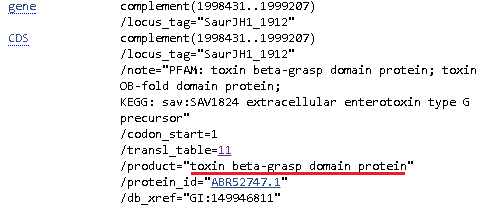
Рис. 6. Фрагмент выравнивания блока s4x1025. Видно, что ATG трех штаммов является стартовым кодоном для cds и не является таковым для Mu50 (не отмечен черным). Рис. 7. Фрагмент выравнивания блока s4x1025. Видно, что GTG является стартовым кодоном для генов штаммов Mu50 и JH1, при этом гены в данных штаммах обозначены по-разному. Рис. 8. Аннотация белка в геноме штамма Mu50 (слева) и JH1 (справа). Кстати, на рис. 7 можно также обратить внимание на интересную ситуацию: для первого штамма (ED133) стартовый кодон не GTG, как в остальных, а ATG, который смещен вправо. При этом после белок аннотирован как гипотетический (то есть, вероятно, только из-за присутствия ATG в посл-ти) и его ген имеет размер 756 пн (рис. 9). То есть если бы стартовый кодон не сместился, а был бы распознан как GTG вместе с такими же у других штаммов, то ген стал бы абсолютно аналогичен генам JH1 и Mu50.
Рис. 9. Фрагмент выравнивания блока s4x1025. Видно, что АTG был распознан как стартовый кодон для гена в штамме ED133, хотя в этом же месте такая посл-ть присутствует и во всех других штаммах, там стартовым кодоном является GTG. Подытоживая, можно сказать, что программа NPGE служит прекрасным визуализатором масштабных множественных выравниваний (таких больших, как выравнивания геномов) и помогает выделить нужную информацию. Это может активно применяться для аннотирования генов.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| © Alexandra Boyko, 2014. Faculty of Bioengineering and Bioinformatics, MSU. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||