| Главная страница | Обучение | Обо мне | Ссылки | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Предсказание генов прокариот | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Задание 1. Аннотация генома и сравнение с аннотацией генов в записи GenBank Для аннотации мною был выбран организм, принадлежащий домену Археи, Cenarchaeum symbiosum А (Рис. 1, 2). Эта архея интересна тем, что является эндосимбионтом морской губки Axinella mexicanа. Штамм был получен из губки этого вида, живущей у берегов г. Санта-Барбара (США). BioProject: PRJNA202. Страница сборки (Assembly): GCA_000200715.1. Таксономия организма: домен Archaea; тип Thaumarchaeota; Cenarchaeales; Cenarchaeaceae; Cenarchaeum; Cenarchaeum symbiosum. Как и любой представитель типа Thaumarchaeota, выбранная архея является хемолитотрофом и способна окислять аммиак. Соответственно, может играть существенную роль в биохимическом круговороте веществ. Также она - облигатный психрофил (криофил), то есть растет при температуре от -20 до +10 °C (оптимальная температура жизнедеятельности) и не размножается при температуре выше 20 °C.
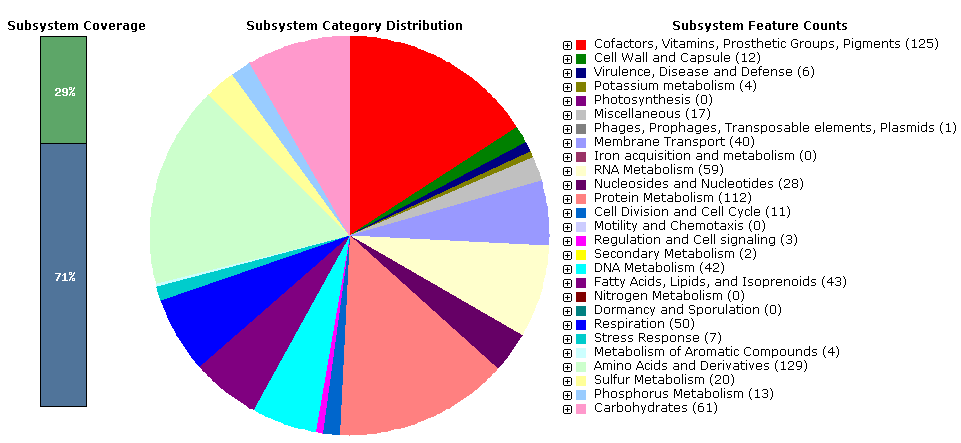
Сборка данного штамма представлена на уровне хромосом. Геном состоит из одной хромосомы (кольцевая ДНК длиной 2045086 пн). Ее запись в GenBank: DP000238. Файл с полной последовательностью генома: GCF_000200715_genomic.fna. Белковые последовательности: GCF_000200715_protein.faa. Всего в геноме закодировано (аннотировано) 2017 белков. Была скачана таблица аннотированных белок-кодирующих генов Protein_annot.xlsx из GenBank'a. Последовательность хромосомы была отправлена на аннотацию на сервер RAST с указанием ID организма. Выдача программы RAST содержит много различной информации. Таблица аннотированных белков была скачана в формате RAST_table.tcv и RAST_table.xls. В хромосоме Cenarchaeum symbiosum А RAST нашел 1897 кодирующих последовательностей белков и 42 РНК. Также этот сервер предоставляет информацию о распределении белков, закодированных на запрашиваемой хромосоме, по их функциям (к каким функциональным системам относится тот или иной белок).
Эти данные показаны в виде диаграммы на рис. 3.
Затем таблицы из GenBank'a и с сервера RAST были совмещены в одну таблицу аннотированных генов Table_annot.xlsx. (Для ее создания были удалены некоторые столбцы из таблицы RAST_table.tcv и Protein_annot.xlsx, введен столбец "Source", строки из таблицы RAST окрашены зеленым для удобства.) Задание 2. Аннотация генов во фрагменте геномной ДНК бактерии Была взята бактерия Acidicaldus organivorans (штамм DX-1), ее таксономия: домен Bacteria; тип Proteobacteria; класс Alphaproteobacteria; порядок Rhodospirillales; семейство Acetobacteraceae; Acidicaldus. Принадлежит семейству уксуснокислых бактерий, получающих энергию из окисления этанола до уксусной кислоты. Грамотрицательные аэробные палочковидные бактерии. Acidicaldus organivorans - ацидофильный термофил, а значит, оптимальный рН для нее варьируется в интервале 5,4—6,3. Сборка осуществлена на уровне скэффолдов: GCA_000759655.1. Скэффолдов собрано 376 штук, контигов - 1 595. Для скэффолдов N50 - 12 041, L50 - 71. Для контигов N50 - 3 731, L50 - 241. BioProject: PRJNA257903. WGS Project: JPYW01. По этой ссылке можно получить Список контигов . На удачу, многие контиги этой сборки не аннотированы, однако в основном контиги были короче 10 Кбаз. В итоге для последующей аннотации был выбран контиг 89 (JPYW01000089) длиной 12 896 бп. Его fasta-последовательность: contig_89.fasta. Последовательность была загружена на сайт GeneMark и были выбраны следующие параметры: таблица генетического кода 11, предсказание на обеих цепях, эвристические параметры из статьи 2010 года (я решила, что со временем алгоритмы должны оптимизироваться, а значит, 2010 версия будет, возможно, точнее, чем 1999). Также для выдачи были запрошены белковые последовательности, нуклеотидные последовательности генов и график кодирующего потенциала в формате PDF. Вот что было получено после запуска этой онлайн-программы для contig_89:
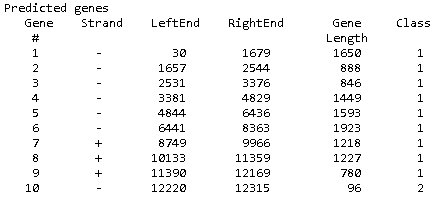
Для удобства представлю основную информацию из файла о координатах предсказанных генов в виде таблицы на рис. 5.
При взгляде на эту табличку сразу бросается в глаза столбец "Class", в котором для всех генов, кроме последнего, проставлены "1" (а напротив 10-го гена стоит "2"). Последний ген отличается от остальных своей длиной - она существенно меньше, всего 96 нуклеотидов, то есть 31 аминокислота в составе 10-го белка. Возможно, продукт этого гена является каким-то особым белком. Затем нужно было найти гомологов каждого гена. Я выбрала поиск с помощью алгоритма blastx, так как он позволяет находить белки-гомологи для заданной нуклеотидной последовательности. Но помимо этого, поскольку GeneMark предлагает на выходе не только нуклеотидные последовательности генов, но и аминокислотные последовательности белков (я, однако, не очень понимаю, как именно устанавливается эта последовательность, ведь генетический код избыточен и одному кодону соответствует не одна аминокислота), я провела поиск гомологов белков по белковой посл-ти продукта того или иного гена (с помощью blastp). Использовался банк SwissProt, бактериальная таблица ген. кода (11) и выдача 20 000 результатов (чтобы не потерять возможных гомологов). Приведу результаты поиска по BLAST в Таблице 1. Таблица результатов аннотации генов, содержащихся в contig_89, с помощью BLAST
* Эти параметры приведены для лучших находок.
** Ситуация поиска неоднозначна, при дополнительном изучении данного варианта не удалось установить четкое соответствие какого-то из двух белков данному гену. Мое предположение состоит в том, что оба варианта несут достаточно близкую функцию (хотя катализируют разные реакции!), а значит, несут очень много похожих доменов и субдоменов (так как взаимодействуют с практически с одними и теми же веществами). Поэтому BLAST'y не удалось разделить эти две находки.
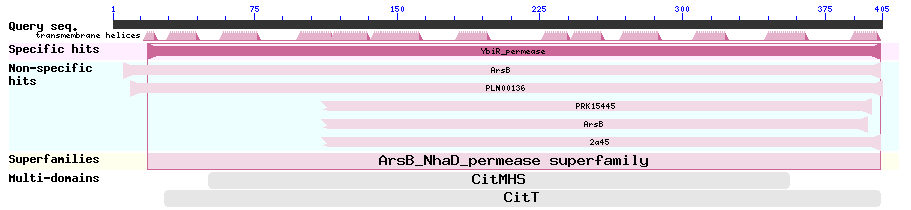
*** Сложный случай, так как найденные белки имеют плохие выравнивания с query, хотя малое количество находок blastx помогает отсечь лишние, найденные алгоритмом blastp. В качестве лучшего (с параметрами, приведенными в таблице) был выбран предполагаемый белок ArsB. Подтвердить такой вариант аннотации можно доменной структурой, анализ которой предоставляет сервер NCBI (рис. 6).
По рис. 6. видно, что почти вся последовательность подходит под домен суперсемейства ArsB_NhaD пермеаз, то есть белков, осуществляющих транспорт органических и неорганических ионов (арсенат, сульфат и др.) через биологические мембраны как за счет осмоса, так и формируя канал АТФ-зависимого анионного насоса.
Затем я ограничила поиск BLAST так, чтобы он осуществлялся только среди бактерий, и результаты изменились: лучшей находкой стал белок Uncharacterized transporter MT2759. Он также содержит домены, которые присутствуют и у суперсемейства ArsB_NhaD пермеаз (см. Рис. 7), однако его функции еще окончательно неохарактеризованы, впрочем, скорее всего, он тоже входит в состав этого суперсемейства и является трансмембранным транспортером.
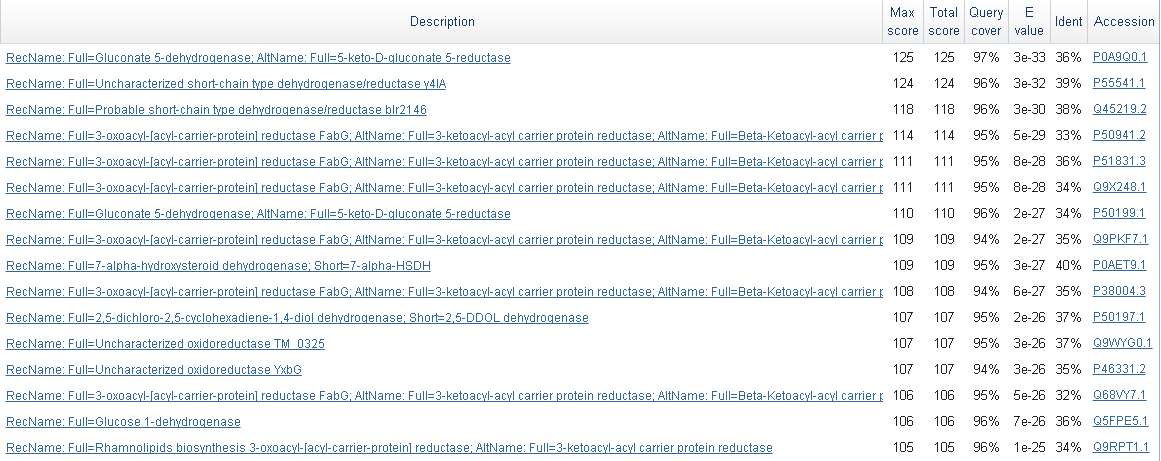
**** Результаты поиска последовательности гена и его продукта в BLAST (в данном случае оба варианта поиска совпадают по результатам) не позволяют однозначно определить белок, закодированный в gene_9. На рис. 8 показан фрагмент выдачи blastx (лучшие находки).
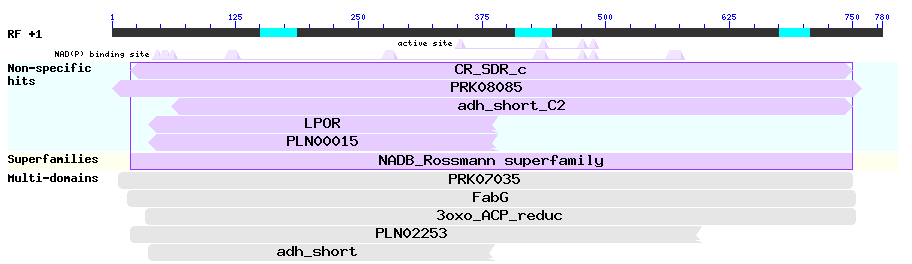
Как и в предыдущих случаях, я посмотрела доменную структуру, предлагаемую NCBI для искомого белка (рис. 9).
Из рис. 9. видно, что данный белок принадлежит NADB_Rossmann суперсемейству. В него входят многие оксидредуктазы (в частности, дегидрогеназы, одна из которых - глюконат-5-дегидрогеназа - встречается и в результатах этого поиска), которые содержат в своей структуре так называемую укладку Россмана - фрагмент β–α–β–α–β, связывающий один нуклеотид. Таким образом, в это суперсемейство входят такие оксидредуктазы, которые связывают НАД(Ф)Н или НАД(Ф)+ в качестве кофактора. Скорее всего, девятый ген кодирует дегидрогеназы/3-оксоацил-ACPредуктазу, но какую именно - однозначно определить нельзя. ***** К сожалению, gene_10 аннотировать не удалось, так как у него получается очень короткий продукт. Я предположила, что этот ген кодирует не белок, а какую-либо РНК и провела поиск по blastn (somewhat similar sequences) (база данных nr/nt). Однако лучшего результата это не принесло - во-первых, возникли находки из эукариотических геномов, а во-вторых, они были по своим параметрам не очень достоверны.
Впрочем, было несколько относительно неплохих находок: с conserved hypothetical protein partial mRNA из организма Theileria orientalis strain Shintoku (рис.10) и с участком генома организма Synechococcus phage S-CBP2 (рис. 10). Тем не менее, эти находки не помогают сделать каких-либо выводов о десятом гене. Вообще, его продукт (если это белок) очень короток и даже белком не является, а, скорее, пептидом. Наиболее вероятны две ситуации - либо этот участок кодирует РНК, либо этот участок вообще некодирующий.
Использованные источники:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| © Alexandra Boyko, 2014. Faculty of Bioengineering and Bioinformatics, MSU. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||