| Главная страница | Обучение | Обо мне | Ссылки | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
A-, B- и Z-формы ДНК. Структура РНК | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Задание 1. С помощью программы fiber пакета X3DNA были построены дуплексы ДНК в A-, B- и Z-формах. А-форма и В-форма состоят из последовательности нуклеотидов gatc, повторенной 5 раз. Z-форма состоит из пар GC, повторенных 10 раз.
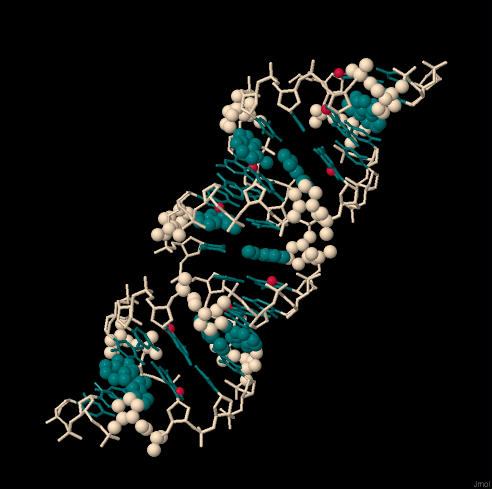
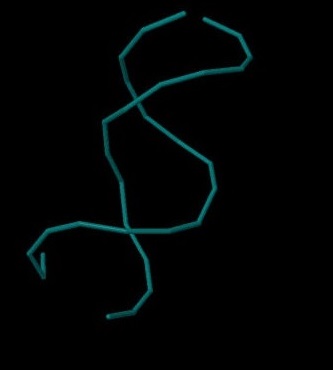
Соответствующие PDB-файлы, созданные программой fiber: A-форма, В-форма, Z-форма. Задание 2. Упражнение 1. Результаты покраски различных фрагментов ДНК см. на рис. 2. ДНК целиком состоит из нуклеотидов, поэтому можно сказать, что нуклеотиды отображены на этом рисунке в проволочной модели.
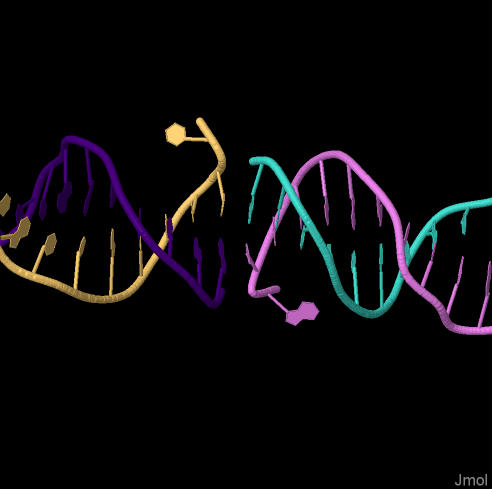
Упражнение 2. В этом части практикума были скачаны 2 структуры. Первая - ДНК-белковый комплекс с pdb-кодом 1LQ1, biological assembly 1. Белок, ассоциированный с ДНК, является ключевым фактором транскрипции в спорулирующей (образующей споры) бактерии. С помощью команды restrict dna были показаны только цепочки ДНК (2 субъединицы белка были скрыты), а команда color chain наглядно показала, что в ДНК есть разрыв, обусловленный тем, в файле находится 2 ДНК-дуплекса (см. Рис.3).
Для того, чтобы обеспечить структуру без разрывов, были выбраны цепи G и Н.
В выбранном фрагменте промежутков нет, это показано на рис.4. Вторая структура - комплекс тРНК и аспартил-тРНК-синтетазы. Последняя играет роль транспортера аспарагиновой кислоты к рибосоме.
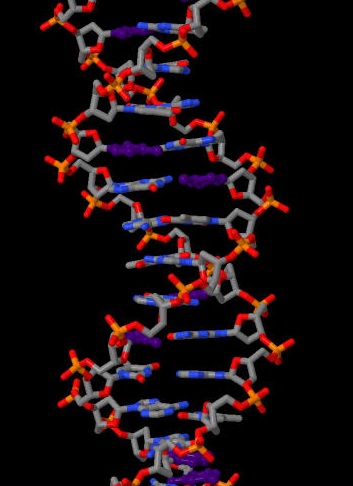
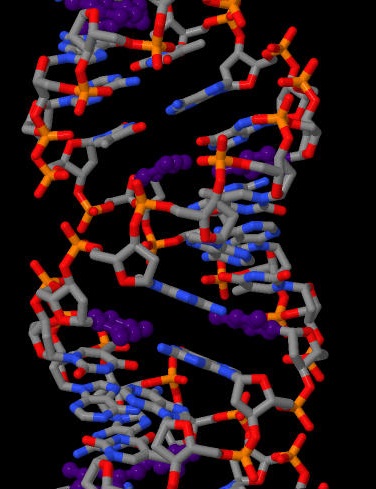
На рисунках хорошо видно, что обе структуры неразрывны. Они (без сопутствующих белков) были сохранены в отдельный файлы, ДНК (цепи E и F) в файл 1LQ1.pdb, а цепь тРНК - в файл 1C0A.pdb. Задание 3. Упражнение 1. В В-форме дуплекса ДНК визуально довольно легко выделить большую и малую бороздки. В этом задании мне нужно было понять, какие атомы цитозина обращены в сторону большой бороздки, а какие - в сторону малой. Для большей наглядности было сделано изображение В-формы ДНК (рис.5), где цитозины выделены отдельно.
Был выбран цитозин, входящий в состав 32-го нуклеотида цепи В. Он также приведен на рис. 5, атомы раскрашены в разные цвета в зависимости от того, куда смотрит тот или иной атом. Также было изучено расположение атомов цитозина в А-форме молекулы ДНК (изображена на рис. 5). Ниже приведены результаты. Резюме:
Z-форма отличается по строению от А и В. Она строится на многократном повторении пар GC и CG, поэтому ее структура визуально непохожа на предыдущие 2 формы ДНК. В частности, в ней можно выделить только минорную бороздку. Упражнение 2. Сравнение основных спиральных параметров различных форм ДНК: параметры приведены в Таблице 1. Таблица 1. Основные спиральные параметры ДНК
Упражнение 3. В данном упражнении необходимо сравнить торсионные углы dCMP (нуклеотиде, содержащем цитозин) в А- и В-формах ДНК. Был выбран С28 (см. Рис. 6). Значения торсионных углов, полученные вручную, сравнивались с данными в презентации. Сравнение представлено в виде Таблицы 2.
Таблица 2. Торсионные углы А- и В-форм ДНК
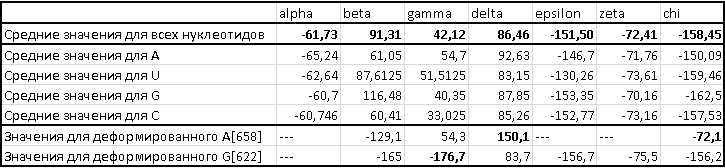
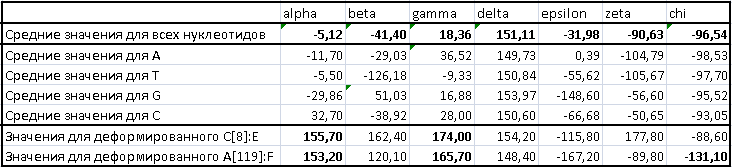
Задание 4. В этом задании структуры трех форм ДНК и структура тРНК были переведены в старый формат с помощью программы remediator для работы с ними в пакете 3DNA. Затем были выполнены команды find_pair -t XXXX_old.pdb stdout | analyze, результатом которых являются файлы с информацией о параметрах структур ДНК и тРНК. Упражнение 1. Для тРНК были получены значения торсионных углов (рис.7). С помощью Excel были найдены средние значения для каждого угла для всех нуклеотидов и для каждого нуклеотида по отдельности. Полученные данные приведены в Таблице 3. В этой же таблице приведены значения углов для "деформированных" нуклеотидов( т.е. таких, у которых значения одного или нескольких торсионных углов сильно отличаются от средних значений для данной тРНК).
Аналогичная работа была проделана для структуры ДНК, взятой из файла 1LQ1.pdb. Значения углов приведены в Таблице 4.
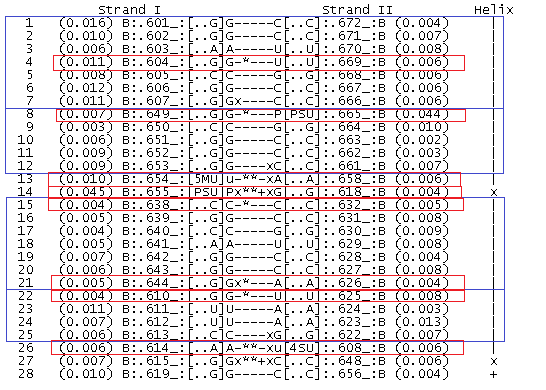
Таким образом, "деформированными" оказались нуклеотиды А[658], G[622] для тРНК и C[8]:E, A[119]:F для ДНК. Упражнение 2. Была изучена вторичная структура тРНК. С помощью команды analyze была получена таблица водородных связей (рис.8), образующихся между нуклеотидами тРНК. Эта информация позволяет найти стебли, образующиеся в структуре. В этой же таблице указываются неканонические пары оснований (то есть не А-U, не G-C и пары, включающие модифицированные основания).
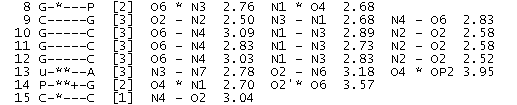
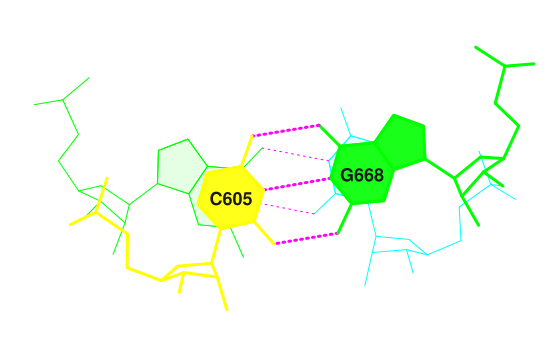
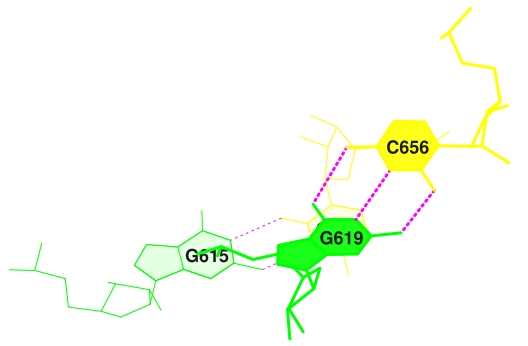
На рис. 8 звездочками помечены изолированные пары оснований, а "x" - те места, на которых происходила смена цепи. Стабилизирующие структуру водородные связи, не входящие в стебли, в данном случае представлены в местах перекрещивания цепей между модифицированными основаниями и обычными (рис. 9). Интересная ситуация возникает в 14 паре: там есть водородная связь между кислородом рибозы псевдоуридина и кислородом гуанина (впрочем, надо заметить, что она самая длинная по сравнению с остальными, а значит, не очень прочная).
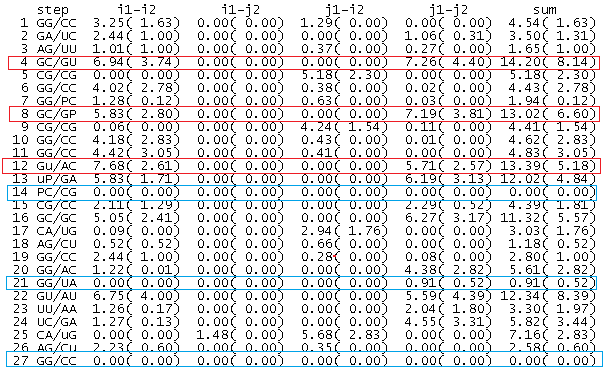
Упражнение 3. Также было рассмотрено стэкинг-взаимодействие оснований тРНК. на рис.10 приведен фрагмент файла, получаемого командой analyze. На этом скриншоте в правой столбце показаны площади перекрывания пар нуклеотидов.
Можно заметить, что есть пары, которые вообще не перекрываются (номера 14, 27) - в этих местах цепи резко поворачивают. С помощью программ пакета X3DNA были получены изображения (рис. 11) стэкинг-взаимодействий для последовательных пар оснований с наименьшей и наибольшей площадью перекрывания.
Так же с помощью команды pdb2img -bc 1C0A_old.pdb 1C0A.ps было получено изображение тРНК с нуклеотидами, представленными в виде блоков (рис. 12).
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| © Alexandra Boyko, 2014. Faculty of Bioengineering and Bioinformatics, MSU. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||