| Главная страница | Обучение | Обо мне | Ссылки | ||||||||||||||||||||||||||||||||||||||||||||
Комплексы ДНК с белком | |||||||||||||||||||||||||||||||||||||||||||||||
|
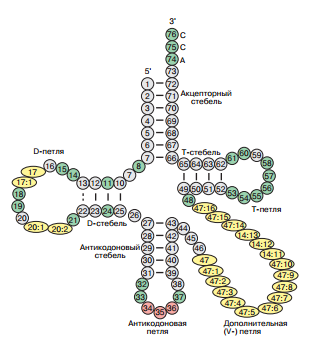
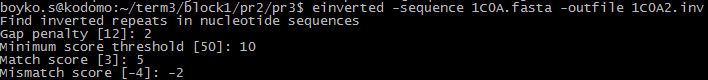
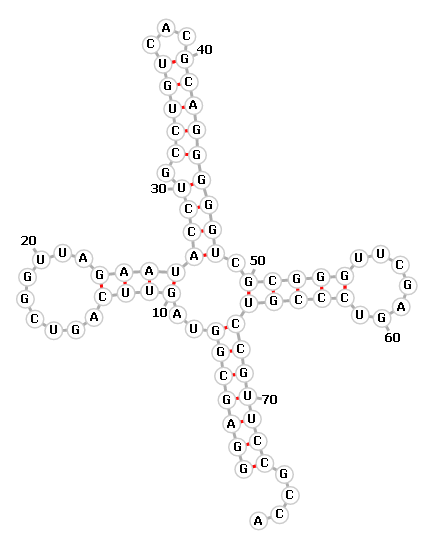
Рис. 1. Вторичная структура молекулы тРНК (взята из статьи О.О. Фаворовой). Задание 1. Предсказание вторичной структуры заданной тРНК Известно, что тРНК обладает фиксированной вторичной структурой, включающей в себя акцепторный стебель, D-стебель, D-петлю, антикодоновую весть (антикодоновый стебель, антикодоновая петля), Т-петлю и Т-стебель, а также иногда дополнительную V-петлю. (структура приведена на рис.1). Необходимо было сравнить предсказания вторичной структуры аспартил-тРНК 1C0A.pdb, полученные с помощью команды einverted пакета EMBOSS и с помощью алгоритма Зукера. Упражнение 1. Поиск вторичной структуры тРНК с помощью einverted. Данная программа ищет инвертированные участки в нуклеотидных последовательностях. Запрос со стандартными параметрами не дал никакого результата, поэтому параметры были изменены на те, которые представлены на рис. 2. Видно, что пришлось установить довольно низкий порог score, маленький штраф за гэпы, увеличить балл за совпадение и уменьшить штрафной балл за несовпадение. В результате была получена схема комплементарных взаимодействий (представлена на рис.3)
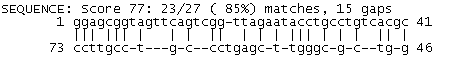
Рис. 2. Команда einverted с использованными параметрами для тРНК, чья последовательность лежит в файле 1C0A.fasta. Рис. 3. Схема инвертированных повторов, предоставленная программой einverted. Очевидно, при таких недостоверных параметрах программа находит далеко не все стебли; отчетливо виден лишь акцепторный (1-7:67-73). Сравнение полученных данных с данными, полученными в прошлом практикуме с помощью программы find_pair, приведены в Таблице 1. Упражнение 2. Поиск вторичной структуры тРНК с помощью алгоритма Зукера. Для построения структуры тРНК была использована программа RNAfold online со стандартными параметрами.
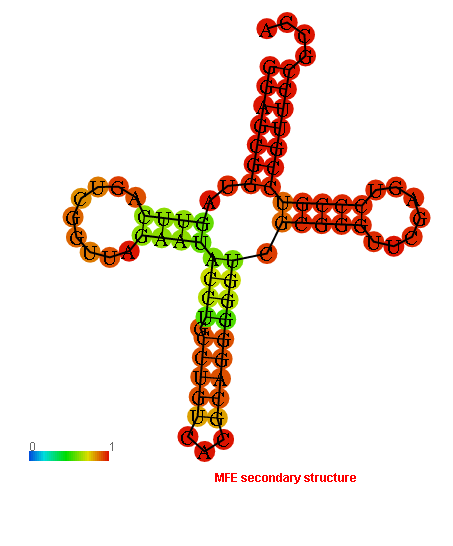
Программа определила минимальную свободную энергию структуры как -28.10 kcal/mol, а по термодинамическому предсказанию -29.03 kcal/mol. Результат в виде картинки приведен на рис. 4.
Рис. 4. Вторичная структура аспартил-тРНК, сформированная программой RNAfold по алгоритму Зукера. Цвет обозначает вероятность пары оснований, шкала приведена на рисунке. Сравнение результатов двух программ по предсказанию вторичной структуры тРНК с реальной структурой, полученной find_pair, приведено ниже в Таблице 1. Таблица 1. Сравнение трех вариантов структур аспартил-тРНК, найденных find_pair, einverted и RNAfold.
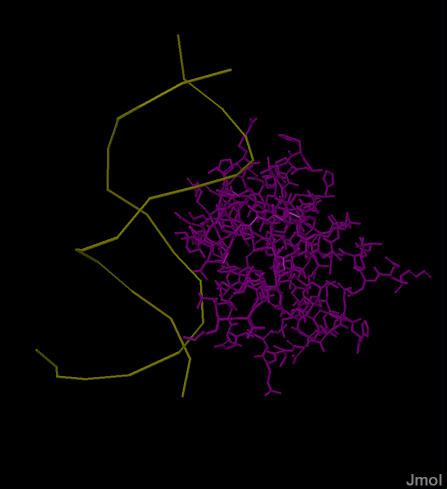
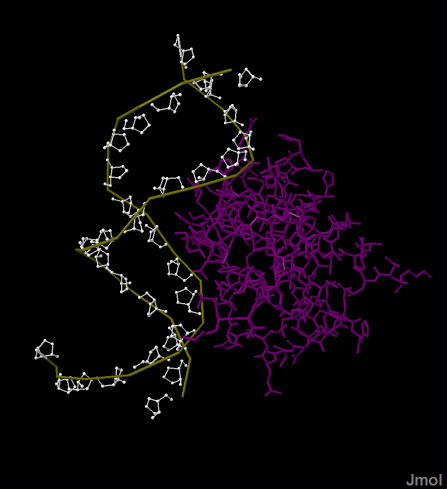
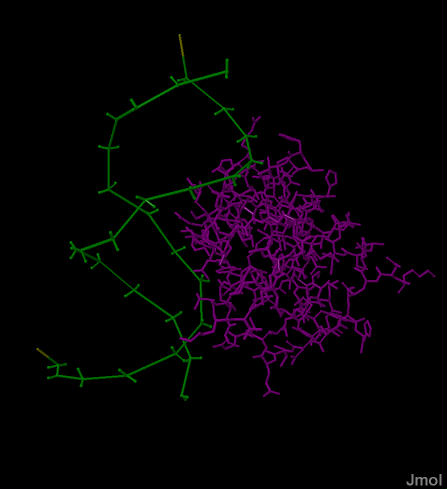
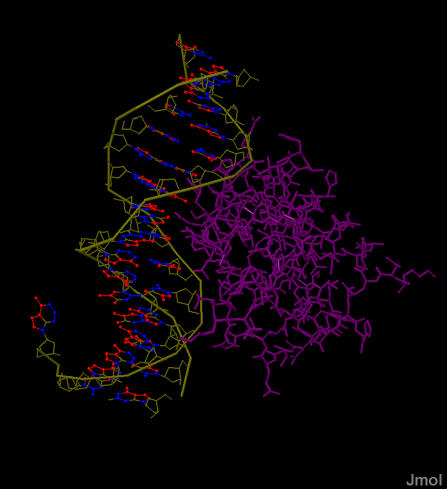
Заметно, что программа einverted заметно проигрывает в достоверности предсказания вторичной структуры тРНК алгоритму Зукера. Вероятно, этому способствовало кардинальное изменение параметров программы, однако при более жестких условиях участки структуры находились хуже. При таких параметрах, по крайней мере, был найден акцепторный стебель. Задание 2. Поиск ДНК-белковых контактов в заданной структуре Была дана структура ДНК-белкового комплекса c PDB-ID 1LQ1, которая представляет собой комплекс ДНК с ключевым фактором транскрипции, инициирующим начало спорообразования бактерий. Из белка была взята цепь D, а из ДНК цепи E и F, так как с двумя другими цепочками ДНК (G и H) белковая цепь D не взаимодействует. Далее использовался файл 1LQ1_c.pdb. Определим кислород и азот как полярные атомы, а углерод, серу и фосфор - как неполярные. Полярным контактом будем считать ситуацию, когда медлу полярным атомом белка и полярным атомом ДНК меньше 3,5 Å. Неполярным контактом будем считать пару неполярных атомов на расстоянии не более 4,5 Å. Были рассмотрены контакты белка с различными группами атомов ДНК: с атомами дезоксирибозы (set1), атомами фосфатов (set2), атомами азотистых оснований, смотрящих в большую бороздку (set3) и малую бороздку (set4). Эти группы изображены на рис. 5.
Рис. 5. Разные группы атомов ДНК, с которыми может взаимодействовать белок. Наверху слева: общая структура. ДНК показана в виде остова оливкового цвета, белок в виде wireframe фиолетового цвета. Наверху справа: Белым цветом покрашены и показаны в wireframe и cpk 2'-дезоксирибозы (set1).
Внизу слева: в виде wireframe и cpk показаны фосфатные группы (set2), они же покрашены в зеленый. Внизу справа: в виде wireframe и cpk показаны азотистые основания: красным покрашены атомы, смотрящие в стороны большой бороздки (set3), синим - в сторону малой (set4). Полярные и неполярные контакты различных функциональных групп ДНК и белка показаны в Jmol-апплете ниже (Скрипт). После того, как структура загрузится, нужно выбрать сценарий: 1. Запустить скрипт: 2. Продолжить исполнение скрипта:
По показанным с помощью скрипта атомам была создана Таблица 2, в которой описано кол-во полярных и неполярных взаимодействий ДНК (цепи E, F) и белка.
Из таблицы видно, что больше всего белок взаимодействует с фосфатами ДНК, а также немного меньше - с азотистыми основаниями в большой бороздке. Упражнение 3. Была построена популярная схема контактов ДНК с белком с помощью программы nucplot (предварительно pdb-фалй был переведен в старый формат программой remediator).
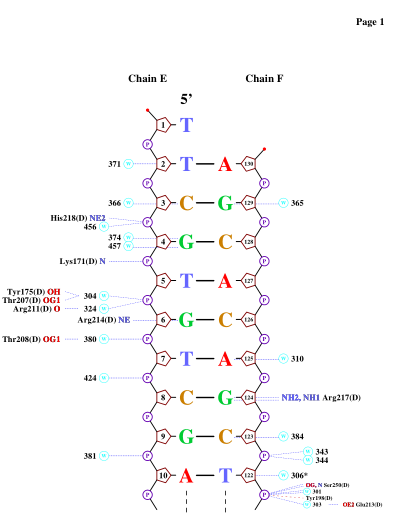
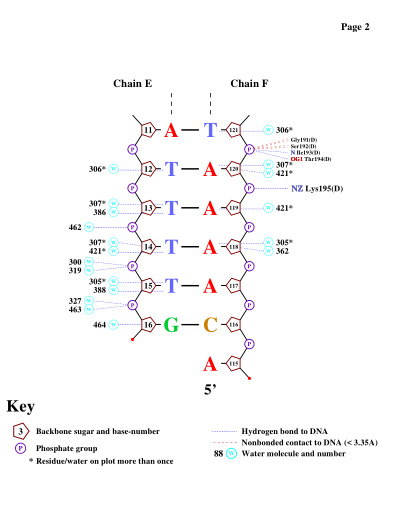
Результат приведен ниже на рис. 6.
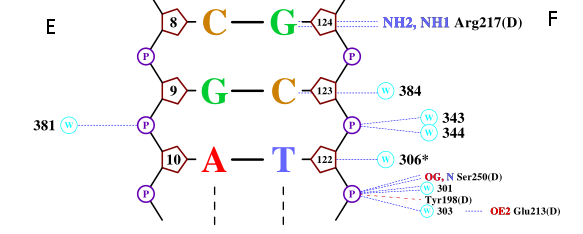
Рис. 6. Популярная схема контактов ДНК и белка, полученная программой nucplot. Судя по схеме, предоставленной nucplot, наибольшее число контактов с ДНК (а именно 2) имеют 2 остатка цепи белка D: Arg(217) (связи через NH2 и NH1) и Ser(250) (связи через OG, N). Они более крупно изображены на рис. 8.
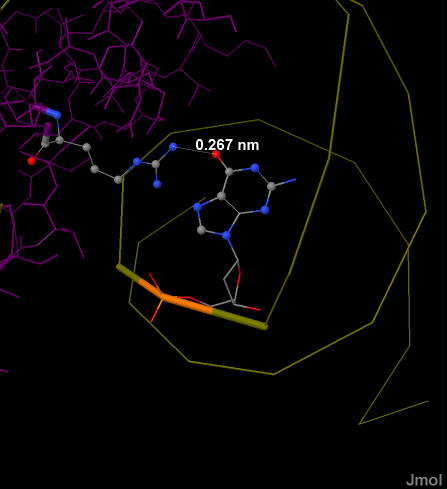
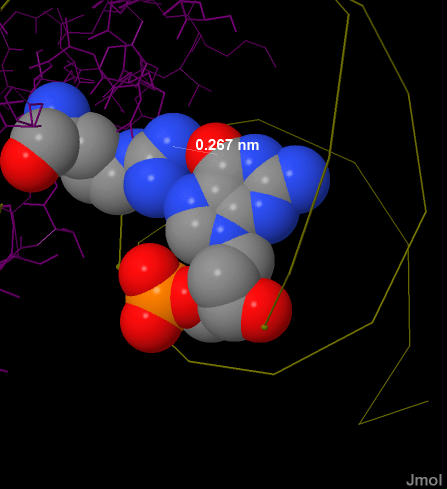
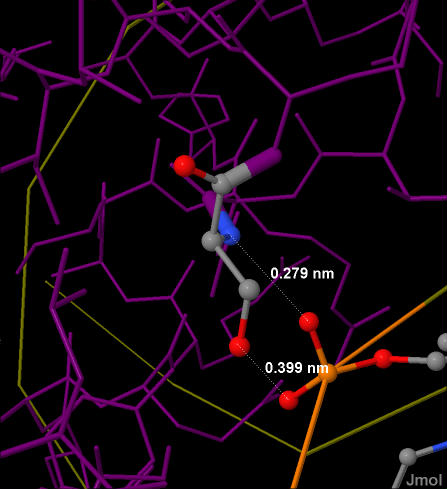
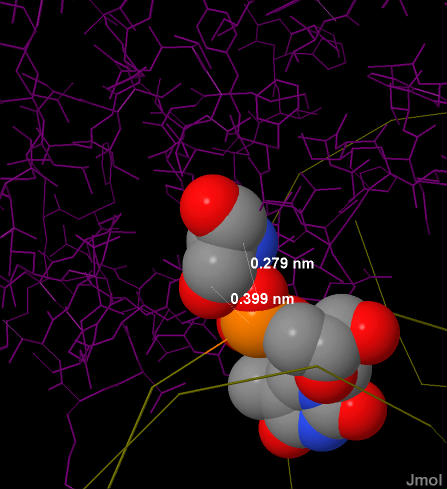
Рис. 8. Фрагмент популярной схемы ДНК-белковых контактов. Показаны кусочки цепей E и F ДНК и контактирующие с ними части белковой цепи D. Видно, что аргинин (Arg217)имеет 2 общие водородные связи с азотистым основанием (гуанином) ДНК, тогда как серин (Ser250) связан с фосфатным остатком нуклеотида. Это может означать, что 217-й аргинин играет более важную роль в распознавании ДНК, так как связывается специфично с азотистым основанием, которое находится "глубже" в структуре ДНК, чем фосфат, с которым связывается 250-ый серин. Иллюстрации контактов этих двух аминокислотных остатков с ДНК приведены на рис. 9. На схеме контактов ДНК с белком также можно заметить скопление аминокислотных остатков около фосфатного остатка между 120 А и 121 Т цепи ДНК F. Однако те взаимодействия не рассматривались, так как 2 из 4 "связей" связями как таковыми не являются, атомы просто сильно сближены друг с другом, а 2 другие связи относятся к разным аминокислотам.
Рис. 9. Сверху слева изображены Arg217 и G124 в виде атомов, покрашены по типу атомов. Видно, что расстояние между ними очень маленькое. Сверху справа картинка, подтверждающая это: атомы этих остатоков показаны с вандерваальсовыми радиусами. Снизу слева изображены 121-ый фосфат и Ser250, покрашены по типу атомов. Снизу справа аналогично подтверждается маленькое расстояние между атомами ДНК и белка. | |||||||||||||||||||||||||||||||||||||||||||||||
| © Alexandra Boyko, 2014. Faculty of Bioengineering and Bioinformatics, MSU. | |||||||||||||||||||||||||||||||||||||||||||||||