| Главная страница | Обучение | Обо мне | Ссылки |
Чтение последовательности ДНК по Сэнгеру | |||
|
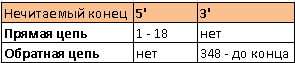
Задание 1. Исходные файлы хроматограмм: WS2980_H3_F_A03 (прямая цепь), WS2980_H3_R_A04 (обратная цепь). Нечитаемые концы хроматограмм приведены в таблице справа. Следует отметить, что после удаления соответствующих концов нумерация нуклеотидов автоматически изменилась. В таблице приведены цифры для исходных последовательностей. Для обратной цепи указаны числа по нумерации после применения к посл-ти reverse+complement. При выравнивании хроматограмм получилось так, что 1-ый (читаемый) нуклеотид прямой цепи приходился на 49-ый нуклеотид обратного комплемента, а нечитаемых нуклеотидов в прямой цепи еще 18, они были достроены по обратной цепочке, а 31 начальный нуклеотид обратной цепи был удален. Характеристика качества хроматограмм:
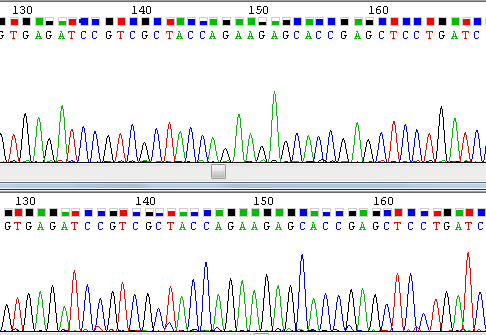
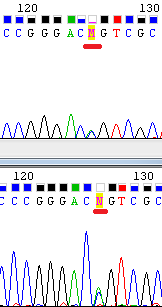
Хроматограмма прямой цепи Хроматограмма обратной цепи Редактирование последовательности Последовательности прямой и обратной комплементарной цепи (FASTA-файл прямой цепи, FASTA-файл обратной цепи) были введены в программу Jalview, где были выровнены, затем некоторые позиции были отредактированы. Результат выравнивания приведен в отдельном окне Jalview-проекта. Измененные нуклеотиды прописывались строчными буквами, а также отмечались "х" в строке Changes. Комментарии по редактированию: На Рис.1. показан фрагмент хроматограмм, который читается без каких-либо проблем.
При редактировании были введены следующие обозначения:
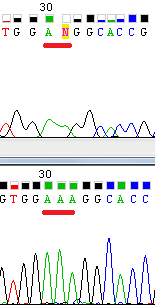
Ниже описаны некоторые проблемные ситуации, возникшие в ходе работы. 1. Пропуск нуклеотида и нераспознанный нуклеотид в прямой цепи. См. рис.2, 1). В прямой цепи в позициях 32-33 пропущено место и стоит N, что портит выравнивание двух цепей. Это можно исправить, посмотрев на обратную комплементарную цепь - там программа однозначно распознает 3 А подряд.
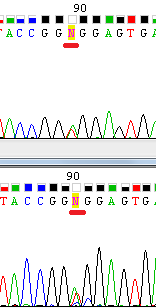
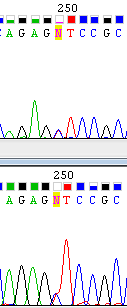
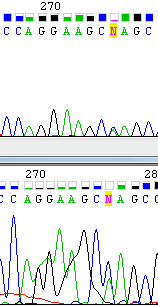
2. Неоднозначная трактовка нуклеотида. См.Рис.2, 2). В позиции 90 Chromos поставил N, но в прямой последовательности в принципе можно увидеть, что там стоит либо А, либо Т, поэтому я поставила W. 3. Пример полиморфизма. См. рис.2, 3). В позиции 126 прямой посл-ти программа поставила букву М (означающую полиморфизм, А или С), в обратной - N. Пики практически совпадают друг с другом и по высоте примерно в 2 раза ниже окружающих, а шум достаточно низкий, поэтому я решила, что там скорее всего либо А, либо С, и в обоих посл-тях поставила М. Аналогично было сделано в 184-ом нуклеотиде (там либо Т, либо С, поэтому была вписана буква Y). 4. Наложение пиков. См. рис.2, 4). В прямой цепи пик гуанина накладывается на пик аденина, в обратном комплементе ситуация не лучше. Скорее всего, в этой позиции должен стоять либо A, либо G, как видно в прямой цепи, поэтому я заменила N на R. 5.Наложение и размытость пиков в обратной цепи. См. Рис. 2, 5).
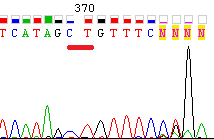
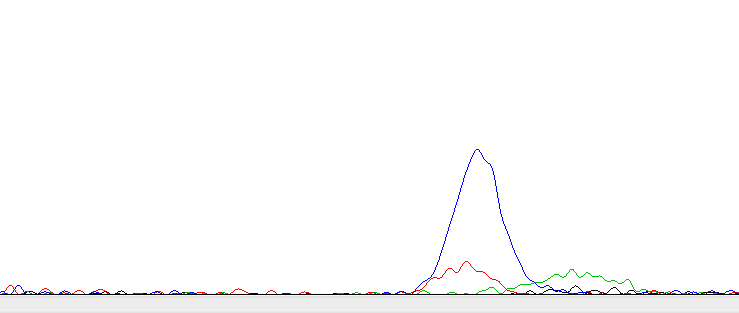
В обратной цепи на показанном фрагменте пики довольно размыты и перекрываются с соседними, однако программа сопоставила их с правильными нуклеотидами (их правильность можно проверить по прямой последовательности, где сигналы хорошо читаемы). В конце прямой последовательности сомнительные нуклеотиды подтвердить нечем, так как читаемый кусок обратной цепи заканчивается раньше. Поэтому я оставила в последовательности те нуклеотиды, которые программа определила верно,но отрезала кусок после 370-го нуклеотида (см. рис. 3.), так как там после С, может быть, стоит еще один С, а может быть и ничего не стоит. Проверить по обратной цепи возможности нет. Итоговая (после редактирования) последовательность прямой цепи ДНК: WS2980_H3_F_A03_final. Последовательность обратного комплемента: WS2980_H3_R_A04_final. Финальная последовательнсть считываемого фрагмента последовательности: final_seq.fasta Задание 2. В качестве примера нечитаемой последовательности был взял файл WSV23_COI_F_A01. Фрагмент нечитаемой хроматограммы, полученной в результате секвенирования по Сэнгеру, приведен на Рис.4.
В такой хроматограмме не указана даже верхняя шкала с буквами N, сигнала будто бы вообще нет, а большой широкий пик, возможно, является просто кляксой. По-видимому, секвенирование было проведено неудачно и не были получены последовательности, оканчивающиеся на терминирующие нуклеотиды, которые затем считываются хроматографом. | |||
| © Alexandra Boyko, 2014. Faculty of Bioengineering and Bioinformatics, MSU. | |||