| Главная страница | Обучение | Обо мне | Ссылки |
Мембранные белки | |||
|
Задание 1. Анализ множественного выравнивания трансмембранных белков Для анализа был выбран белок MgtE (транспортер марганца с PDB-кодом 4WIB, цепь А). Был проведен blast на сайте EMBL-EBI по БД UniRef50, на выходе было получено 200 хитов, худший из которых имел E-value = 2.9E-28. Затем было построено множественное выравнивание полученных находок с помощью алгоритма Muscle. Fasta-файл с множественным выравниванием. К сожалению, выравнивание получилось очень плохим. В первую очередь потому, что все находки оказались заметно длиннее, чем подававшаяся на вход (исходная - 178 остатков, а хиты в среднем около 400). К тому же, в выборку белки попались довольно разные, и сложно было сделать какой-то более строгий порог по E-value, например (а не по количеству хитов), так как он менялся плавно от лучшей находки до худшей (не считая оригинала). Из-за разнообразия длин, а также в принципе аминокислотного состава образовалось много длинных гэповых участков, и в консервативные (хорошо выровненные) блоки попали в основном гидрофобные аминокислоты вроде лейцина, валина, изолейцина, фенилаланина, хотя в тех же блоках встречаются и заряженные аминокислоты, вероятно, несущие функцию, связанную с деятельностью мембранных белков. Из первоначальной выборки было удалено довольно большое количество белков - в первую очередь, коротких, содержащих много гэпов даже в консервативных позициях. Так, было удалено большинство белков длиной менее 300 амк. В проекте Jal-view можно посмотреть исходное множественное выравнивание (вкладка Original) и "почищенное" выравнивание, состоящее из 167 последовательностей (включая исходную), которое затем использовалось для предсказания трансмембранных спиралей. Надо сказать, что итоговое выравнивание (Fasta-файл с итоговым выравниванием) все равно содержит часть (процентов 40) плохо выровненных белков, которые не входят в блоки консервативности. Между консервативными позициями (трансмембранными частями) ситуация сильно разнится - как по длине, так и по составу, похоже, какие-то белки являются практически полностью мембранными, а какие-то еще имеют цитоплазматические домены (или надмембранные). Разметка трансмембранных доменов была сделана по цепи А MgtE с помощью изображения исходного белка в Jmol, которое показано на рис. 1. Изображение получено по координатам, взятым с сервиса OPM для того, чтобы отметить p и n стороны. Результаты приведены в Jal-view проекте: вкладка Original содержит исходное множественное выравнивание, checked - немного улучшенное выравнивание (удалены плохие последовательности) + разметка TM_REAL и TM_PREDICTED. В строке TM_REAL буквы М отображают трансмембранные домены исходного белка MgtE. .jpg) Красными точками отмечена наружная мембрана p(+), синими точками - внутренняя, n(-). Покраска была получена в программе Jmol, так как в Jal-view не удавалось задать User defined. Также были предсказаны трансмембранные участки последовательности гомолога A0A0W0WQE3_9GAMM (MgtE из организма Legionella israelensis) на сервере TMHMM с помощью скрытых марковских моделей. Результат предсказания (график) приведен на рис. 2. 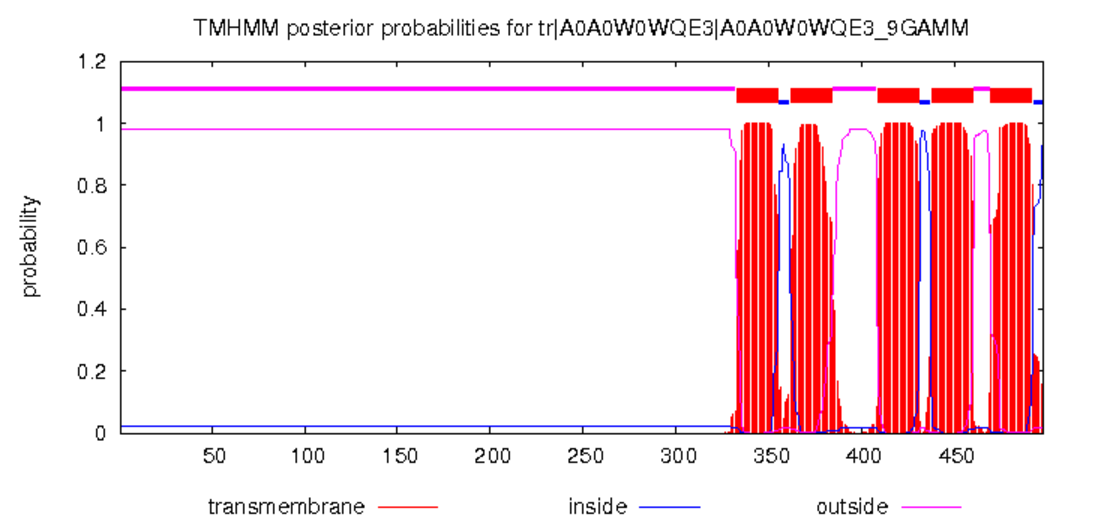 Предсказанные трансмембранные участки для белка-гомолога приведены в Jal-view проекте в строке TM_PREDICTED и обозначены буквой Р. В целом нельзя сказать, что сервер ТМНММ совершил где-то ошибку. Его предсказания примерно совпали с трансмембранными участками, обозначенными мной по Jmol-рисунку. По-видимому, трансмембранная часть выбранных белков содержится в конце последовательностей, там хорошее выравнивание и в консервативных позициях стоят в основном гидрофобные остатки (лейцин, изолейцин, валин и пр.), также в некоторых местах попадается аспарагиновая кислота (по предсказанным участкам она встречается всего один раз, а по моей разметке - 4 вместе с глутаматом в одной позиции), что может быть важно функционально для связывания положительно заряженного двухвалентного иона магния (или марганца и кальция, которые тоже транспортируются белками этого семейства). Начальный участок (первая половина) последовательностей очень не консервативен, для исходного белка он (короткий кусок последовательности) является цитоплазматической частью, что противоречит предсказанию ТМНММ: сервер почему-то обозначает начальный участок как надмембранный. Надо заметить, что у большинства остальных белков в части до предсказанных трансмембранных спиралей присутствует немалое количество аспартатов и глутаматов, что подтверждает отрицательный заряд (и соответственно нахождение "под" мембраной). Репрезентативность полученной выборки белков Проверялось, достаточно ли репрезентативная выборка белков была взята для практикума. Для этого с помощью BLAST было получено 935 хитов. Для них был построен график зависимости величины отрицательного lg(e-value) от номера найденной последовательности в выборке. Он приведен на рис. 3. 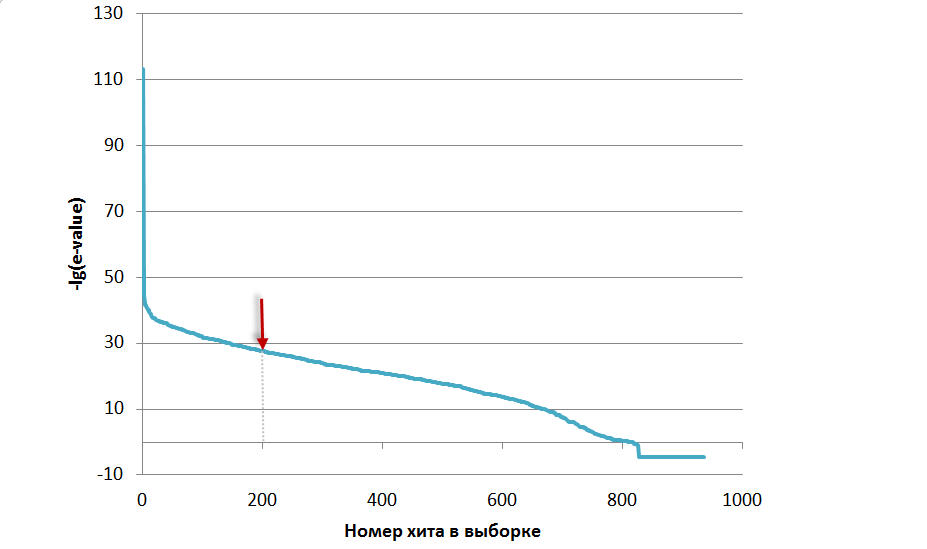 Стрелкой отмечено место (200 хитов), где обрывается выборка, которая была взята для работы. По графику видно, что e-value менялся довольно постепенно, рос плавно, то есть, вообще говоря, нет оснований отсекать именно первые 200 находок для дальнейшего изучения. Однако ясно, что обрабатывать все ~ 900 находок было бы тоже довольно затруднительно. Эти 700 сверх взятых добавили бы разнообразия в цитоплазматические участки и фрагменты между трансмембранными спиралями и может быть усилили бы "подтвержденность" консервативных позиций.
А может и нет, так как даже в выборке из первых 200 хитов есть процентов 40, не попавших в кластер с предсказанными трансмембранными спиралями.
На страницу четвертого семестра
| |||
| © Alexandra Boyko, 2014. Faculty of Bioengineering and Bioinformatics, MSU. | |||