| Главная страница | Обучение | Обо мне | Ссылки |
Электронная плотность в PyMol | |||
|
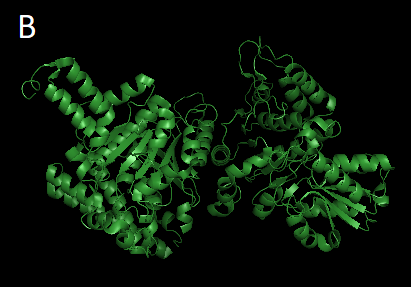
Для работы с кристаллической структурой, полученной методом рентгено-структурного анализа (РСА), был выбран белок 2-оксоглутаратдекарбоксилаза из организма Mycobacterium smegmatis (по PDB-коду 2YIC можно найти информацию об этой структуре на сайте EMBL). Этот фермент катализирует реакцию декарбоксилирования 2-оксоглутарата (ЕС номер 4.1.1.71) с образованием СО2 и сукцинил-СоА. В качестве кофактора белку необходим тиаминпирофосфат. В структуре SucA под кодом 2YIC отсутствуют первые 360 N-концевых остатков, которые в полноценном белке (A0R2B1) входят в состав ацил-трансферазного домена. Таким образом, белок SucA (то есть KGD без 1-360 аминокислотного остатка) не использует ацетил-СоА в ходе реакции и не образует сукцинил-СоА. Тем не менее, у него присутствует дегидрогеназная активность. Белок занимает важнейшее место в метаболизме Mycobacterium smegmatis, аналогичная роль отводится ему и в других бактериях (например, гомолог домена SucA в E.coli (Frank et al., 2007) и в Corinebacterium glutamicum (Hoffelder et al., 2010). Данная структура была получена совместно французскими и английскими учеными и опубликована в работе Wagner et al., 2011 (PMID: 21867916). В ней подробно изучены различные структурные аспекты KGD, а также исследована его активность и регуляция. Это белок длиной 868 аминокислотных остатков и приблизительной молекулярной массой 97 кДа. Для структуры указано хорошее разрешение - 1.96 Å. Итак, для кристалла 2YIC была скачана собственно структура 2yic.ent и файл с электронной плотностью 2yic.ccp4. Изображение полной структуры KGD с электронной плотностью приведено на Рис. 1. Выбранный белок состоит из четырех одинаковых цепей, поэтому в дальнейшей работе использовалась только цепь А.
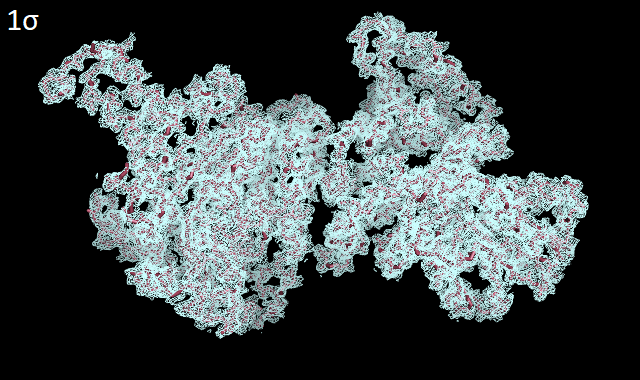
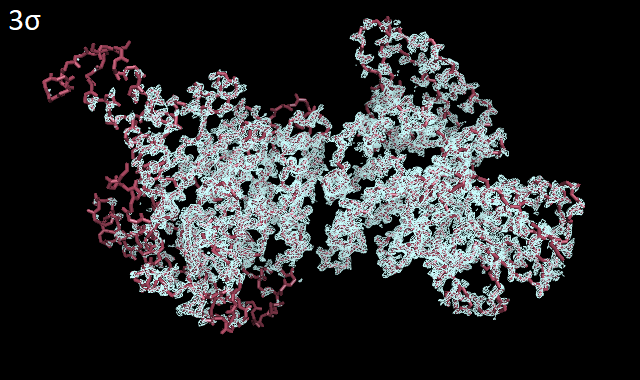
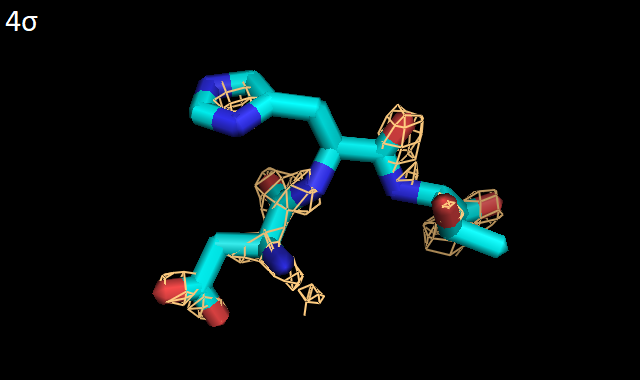
Ниже представлены изображения структуры SucA с электронной плотностью на разных уровнях значений электронной плотность (уровнях подрезки) - от 0.5 до 3σ.
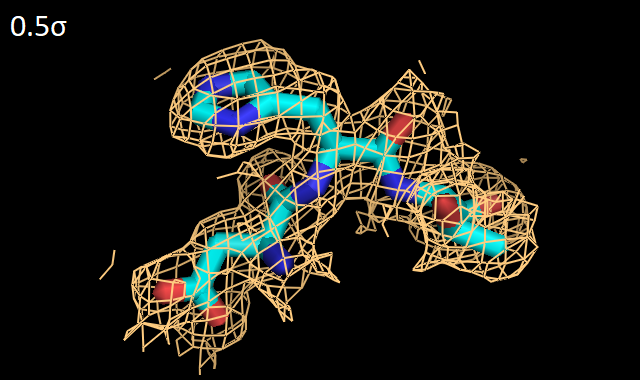
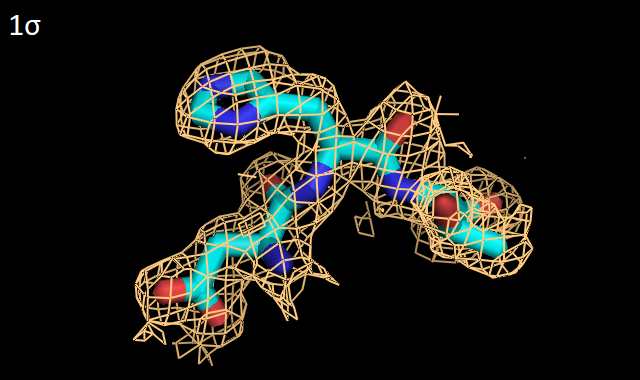
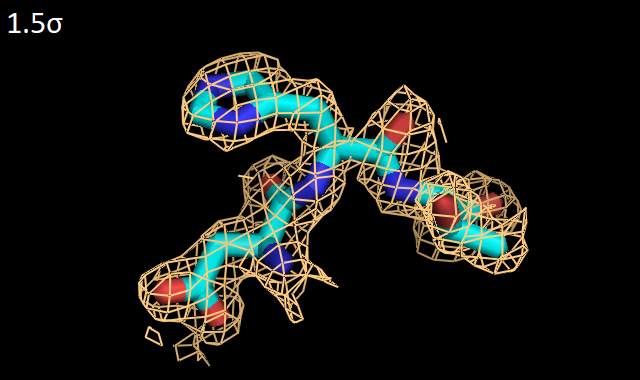
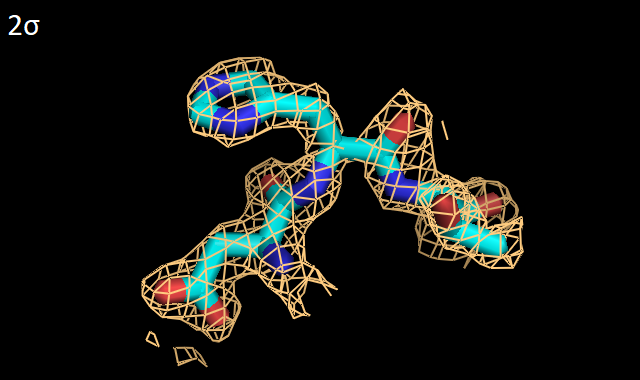
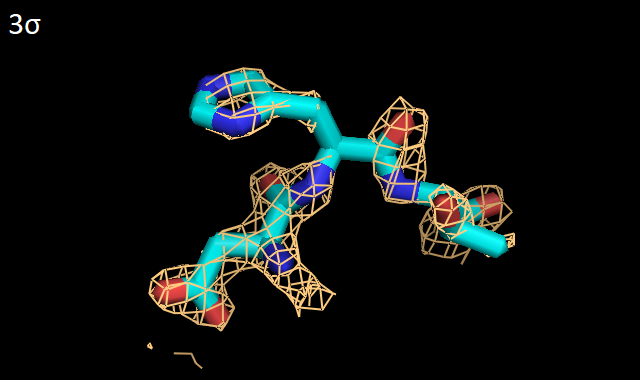
Можно заметить, что на низких уровнях подрезки - от 0.5σ до 1.5σ - электронная плотность хорошо обтягивает полипептидную цепь, с высокой точностью отражая местонахождение ее атомов, тогда как при увеличении уровня подрезки постепенно некоторые части пептидного остова оказываются не покрыты электронной плотностью. По-видимому, это связано с различной подвижностью (и, следственно, качеством кристаллизации) боковых цепей белка по сравнению со внутренностью глобулы. Далее были выбраны 3 аминокислотных остатка из цепи А рассматриваемого белка: Asp1019, His1020 и Thr1021. ДЛя них также были построены изображения электронной плотности с разными уровнями подрезки - от 0.5σ до 4σ (Рис. 3).
По Рис. 3 видно, что все три остатка хорошо покрываются электронной плотностью вплоть до уровня 2σ. При уровне 4σ заметно, что электронная плотность скапливается вокруг атомов, создающих полярность - кислородов аспартата (Asp1019) и OH-группы треонина (Thr1021). Кроме этого, внутри ароматической системы гистидина (His1020) также присутствует электронная плотность, иллюстрируя ее равномерное распределение в кольце. Таким образом, для выбранного белка KGD 3D-модель построена с довольно высоким качеством, и координаты атомов были точно вписаны в полученную электронную плотность. | |||
|
1. Wagner, T., Bellinzoni, M., Wehenkel, A., O'Hare, H. M. & Alzari, P. M. (2011) Functional plasticity and allosteric regulation of alpha-ketoglutarate decarboxylase in central mycobacterial metabolism, Chemistry & biology. 18, 1011-20. 2. Hoffelder, M., Raasch, K., van Ooyen, J. & Eggeling, L. (2010) The E2 domain of OdhA of Corynebacterium glutamicum has succinyltransferase activity dependent on lipoyl residues of the acetyltransferase AceF, Journal of bacteriology. 192, 5203-11. 3. Frank, R. A., Price, A. J., Northrop, F. D., Perham, R. N. & Luisi, B. F. (2007) Crystal structure of the E1 component of the Escherichia coli 2-oxoglutarate dehydrogenase multienzyme complex, Journal of molecular biology. 368, 639-51. 4. ExPASy: 2-oxoglutarate decarboxylase 4.1.1.71 | |||
| © Alexandra Boyko, 2014. Faculty of Bioengineering and Bioinformatics, MSU. | |||