Хемоинформатика
В этом практикуме мы будем искать аналоги ибупрофена.
from rdkit import Chem
from rdkit.Chem import AllChem
from rdkit import RDConfig
from rdkit.Chem.Draw import IPythonConsole
from rdkit.Chem import Draw
import numpy as np
from IPython.display import display,Image
ibu=Chem.MolFromSmiles('CC(C)CC1=CC=C(C=C1)C(C)C(=O)O')
AllChem.Compute2DCoords(ibu)
display(ibu)
Так выглядит ибупрофен. Посчитаем параметры для правила Липински:
import rdkit.Chem.Lipinski as Lipinksy
print Lipinksy.NumHDonors(ibu)
print Lipinksy.NumHAcceptors(ibu)
print Lipinksy.rdMolDescriptors.CalcExactMolWt(ibu)
print Lipinksy.rdMolDescriptors.CalcCrippenDescriptors(ibu)[0]
Ибупрофен удовлетворяет правилу пяти Липински. Сделаем функцию для проверки этого правила для будущих аналогов ибупрофена:
def checkLip(molecule):
return bool((Lipinksy.NumHDonors(molecule) <= 5) and (Lipinksy.NumHAcceptors(molecule) <= 10) \
and (Lipinksy.rdMolDescriptors.CalcExactMolWt(molecule) < 500) \
and(Lipinksy.rdMolDescriptors.CalcCrippenDescriptors(molecule)[0]) <= 5)
checkLip(ibu)
Мы будем искать аналоги, имитируя реакцию Click-химии. В ней изначально участвуют 2 вещества: одно с терминальной алкильной группой, другое - с терминальным азидом. А продуктом является вещество с двойной связью и азидом в цикле. Мы не можем провести реакцию in silico, но можем сымитировать ее, подставив азид в цикл ибупрофена, а потом заменим эту азидную часть на азид + довесок веществ, найденных в PubChem, и таким образом сделаем что-то похожее на ибупрофен.
Сначала модифицируем ибупрофен. Выберем место для модицикации:
ibu2=Chem.MolFromSmiles('C(C#C)C(C)CC1=CC=C(C=C1)C(C)C(=O)O')
AllChem.Compute2DCoords(ibu2)
display(ibu2)
ibu3=Chem.MolFromSmiles('CC(C)CC1=CC=C(C=C1)C(C(C#C))C(=O)O')
AllChem.Compute2DCoords(ibu3)
display(ibu3)
ibu4=Chem.MolFromSmiles('CC(C#C)(C)CC1=CC=C(C=C1)C(C)C(=O)O')
AllChem.Compute2DCoords(ibu4)
display(ibu4)
ibu5=Chem.MolFromSmiles('CC(C)C(C#C)C1=CC=C(C=C1)C(C)C(=O)O')
AllChem.Compute2DCoords(ibu5)
display(ibu5)
Мне нравится последний вариант, будем модифицировать его.
ibu_mod = Chem.MolFromSmiles('N1N=NC=C1C(C(C)C)c2ccc(C(C)C(=O)O)cc2')
AllChem.Compute2DCoords(ibu_mod)
display(ibu_mod)
checkLip(ibu_mod)
Дальше был проведен поиск по PubChem структур веществ, у которых есть терминальная азидная группировка. Нашлось 143075 соединений, они были сохранены в файл 3893474530663651931.txt.
! wget ftp://ftp-private.ncbi.nlm.nih.gov/pubchem/.fetch/88/3893474530663651931.txt
strings = np.genfromtxt('3893474530663651931.txt', dtype=np.str)
smiles = []
for line in strings:
if (len(line[1]) < 30) and (len(line[1]) > 10) and not ( '.' in line[1]):
smiles.append(line[1])
print len(smiles)
Теперь сымитируем клик-реакцию:
good_analogs = []
ibu_mod = 'N1N=NC=C1C(C(C)C)c2ccc(C(C)C(=O)O)cc2'
# Новую молекулу лучше создавать в try из-за битых Smiles:
for smi in smiles:
try:
if not "N=[N+]=[N-]" in smi:
continue
newsmi = smi.replace("N=[N+]=[N-]", ibu_mod)
if checkLip(Chem.MolFromSmiles(newsmi)):
good_analogs.append(newsmi)
except Exception:
pass
len(good_analogs)
Ура, нашлось целых 8259 соединений-аналогов ибупрофена, удовлетворяющих правилу пяти Липински. Посмотрим несколько из них:
from random import sample
type(good_analogs[1])
sample_mols = [Chem.MolFromSmiles(item) for item in sample(good_analogs, 20)]
len(sample_mols)
for mol in sample_mols:
AllChem.Compute2DCoords(mol)
display(mol)
Я очень пыталась заставить работать Draw.MolsToGridImage, но он постоянно ругался на несостыковку типов, хотя там по идее все было правильно. Поэтому мне пришлось использовать для отображения аналогов ибупрофена не эту прекрасную функцию, а обычный display.
Дальше построим Similarity map для 5-го соединения из изображенных выше.
from rdkit.Chem.Draw import SimilarityMaps
from IPython.display import Image
ibu = Chem.MolFromSmiles('CC(C)CC1=CC=C(C=C1)C(C)C(=O)O')
from rdkit.Chem.Draw import SimilarityMaps
fp = SimilarityMaps.GetMorganFingerprint(sample_mols[4], fpType='bv')
fig, maxweight = SimilarityMaps.GetSimilarityMapForFingerprint(ibu, \
sample_mols[4], SimilarityMaps.GetMorganFingerprint)
fig
maxweight
Понятия не имею, откуда берется эта серая линия через все изображение, но как я ни пыталась что-то сделать, она не исчезла. Ну и ладно, similarity map от нее несильно страдает.
m3d=Chem.AddHs(sample_mols[4])
Chem.AllChem.EmbedMolecule(m3d)
AllChem.MMFFOptimizeMolecule(m3d,maxIters=500,nonBondedThresh=200 )
m3d
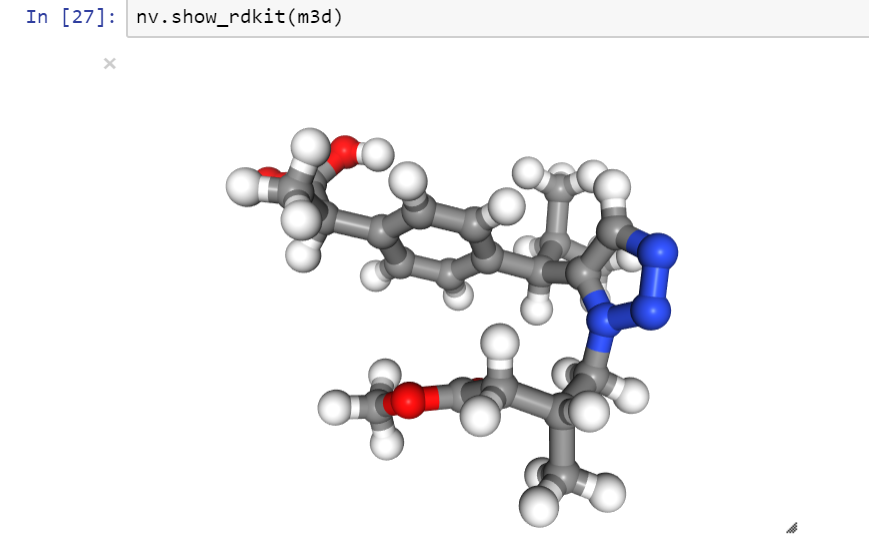
Теперь посмотрим это в 3D:
import nglview as nv
import ipywidgets
nv.show_rdkit(m3d)
Вот такая вот молекула получилась (Рис. 1). (В html она не видна, но на скриншоте видно, что она честно была запущена прям в ноутбуке.)