Задание 1
С помощью программы fiber, пакета 3DNA были получены файлы формата PDB содержащие информацию о структурах трёх молекул ДНК в различных формах:
A-DNA , B DNA, Z DNA
Задание 2
Как легко заметить, сгенерированная программой структура является более конденсированной и симметричной нежели структура полученная экспериментально,
вероятно силовое поле использованное для создания модели плохо отражает действительность.
У обеих форм наблюдается чётко выраженный переход между большой и малой бороздками. (см. Рис.1)
Ширины бороздок соответствуют расстоянию между атомами фосфора остатков DG-14 и DC-4 (большая бороздка) и DC-4 DG-5 (малая бороздка). (см. Рис.2)

синий - сгенерированная
розовый - эксперементально полученная

Пространственное расположение атомов азотистого основания (тимин DT7) относительно бороздок ДНК.
В сторону большой бороздки обращены атомы: c7n1, c7c2, c7o2.
В сторону малой бороздки обращены атомы: c7c4, c7c5, c7c7, c7o4.
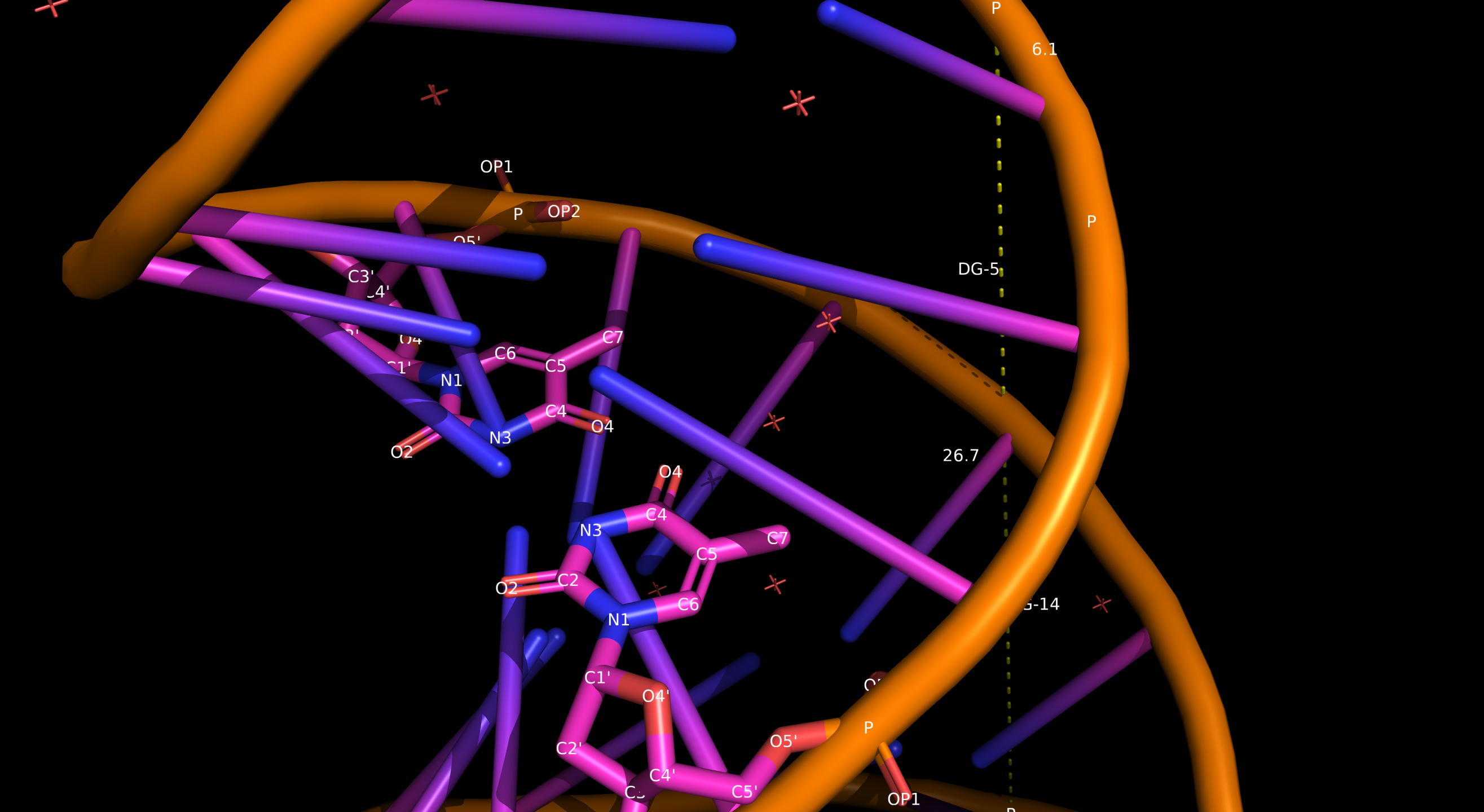
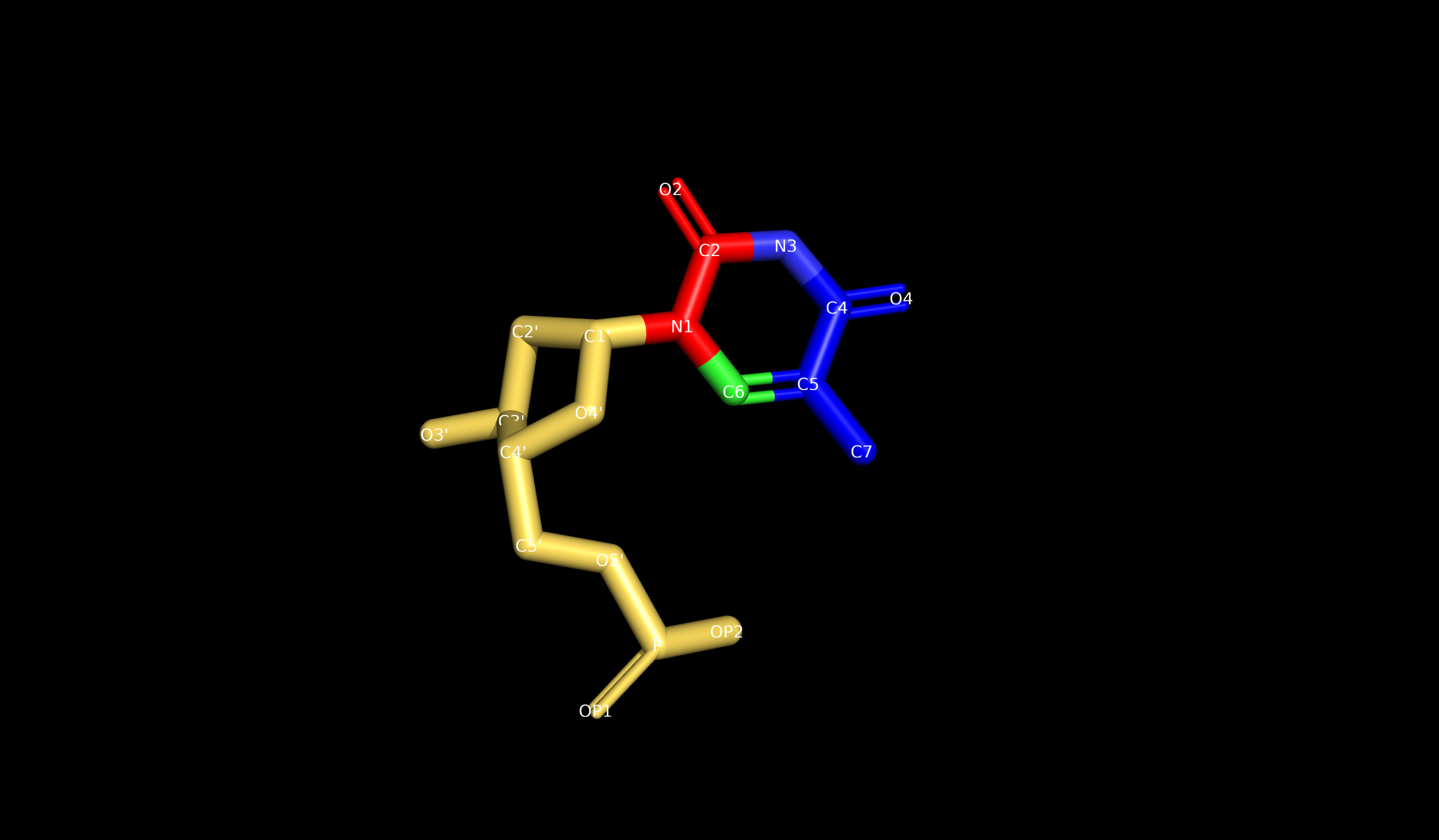
синим - в сторону малой
В таблице 1 приведены числовые характеристики раличных форм ДНК, полученных с помощью программы fiber (поскольку в задании не уточнено, были выбраны предсказанные структуры как более удобные). В Z-ДНК большая бороздка слабо различима, малая бороздка узкая и глубокая.
Таблица 1. Сравнение структур форм ДНК
|
Форма ДНК |
А-форма ДНК |
В-форма ДНК |
Z-форма ДНК |
|
Тип спирали |
правая |
правая |
левая |
|
Шаг спирали |
25.9 |
30.9 |
42.3 |
|
Число оснований на виток |
11 |
10 |
12 |
|
Ширина большой бороздки |
16.8 (A/DC'8/P - B/DT'35/P) |
17.2 (A/DT'11/P - B/DT'27/P) |
- |
|
Ширина малой бороздки |
8.0 (A/DT'11/P - B/DC'24/P) |
11.7 (A/DT'11/P - B/DA'34/P) |
7.2 (A/DG'7/P - B/DG'37/P) |
Задание 3
Таблица 2. Торсионные углы
|
Молекула |
alpha |
beta |
gamma |
delta |
epsilon |
zeta |
chi |
|
tRNA 1o0b |
-16.45 |
33.04 |
44.06 |
83.68 |
-124.32 |
-74.62 |
-137.8 |
|
A-DNA |
62 |
173 |
52 |
88.3 |
178 |
-50 |
-160 |
|
B-DNA |
63 |
172 |
54 |
123.13 |
155 |
-90 |
-117 |
Как мы можем видеть торсионные углы нуклеотидов в тРНК разительно отличаются от таковых в обоих формах ДНК из-за чего определить то, в какой форме находится РНК представляется сложной задачей.
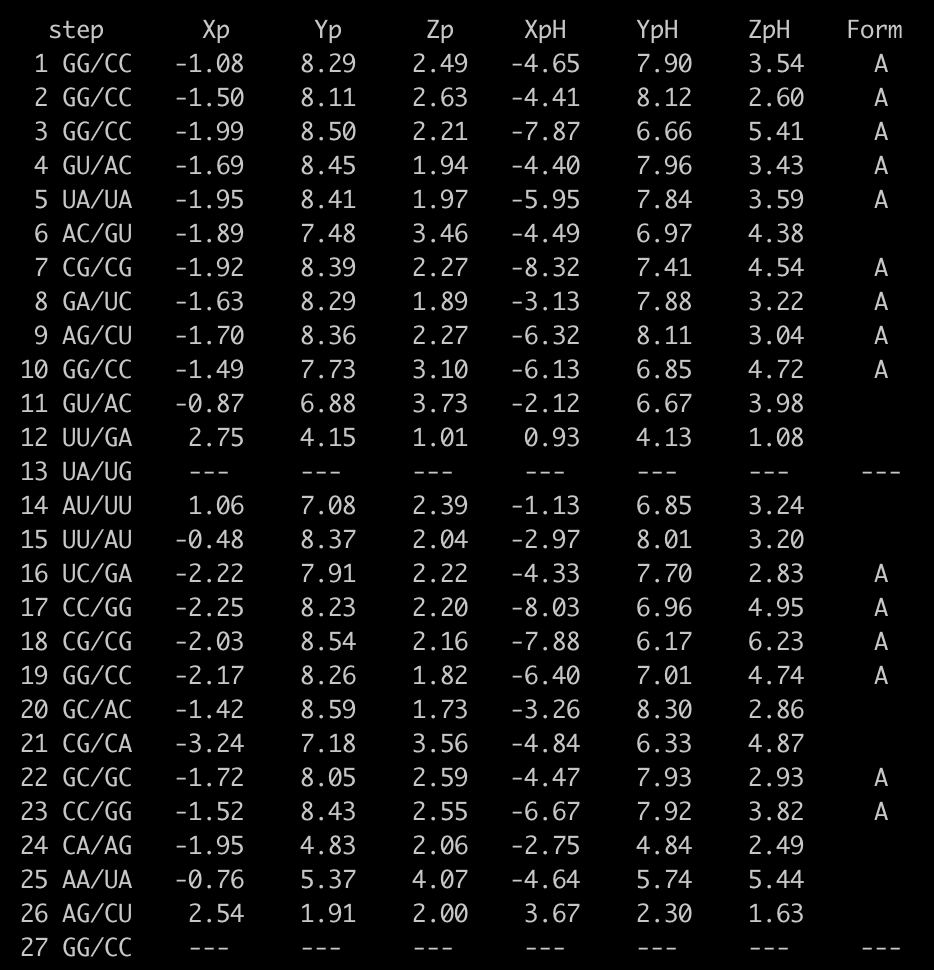
Тем не менее если обратится к команде analyze пакета 3DNA, можно обнаружить что алгоритм распознает форму тРНК как А-форму в большинстве случаев, в остальных программа просто не даёт ответа, в связи с чем можно предположить, что состояние в котором находится тРНК приближено к A-форме ДНК заметно сильнее, нежели к другим формам.
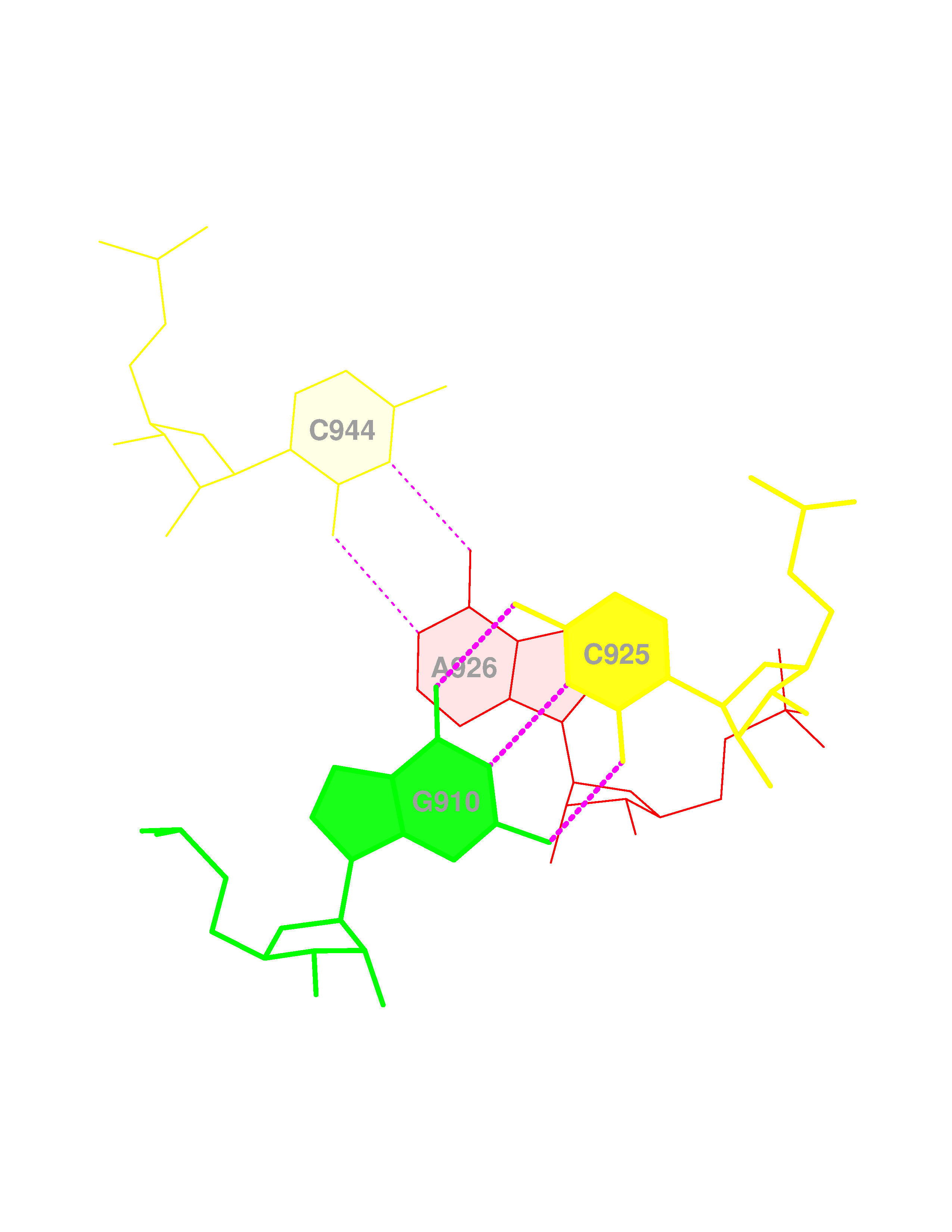
Водородные связи и стебли

Стебли образованы следущими парами нуклеотидов:
Дополнительные водородные связи:
Неканонические пары нуклеотидов:
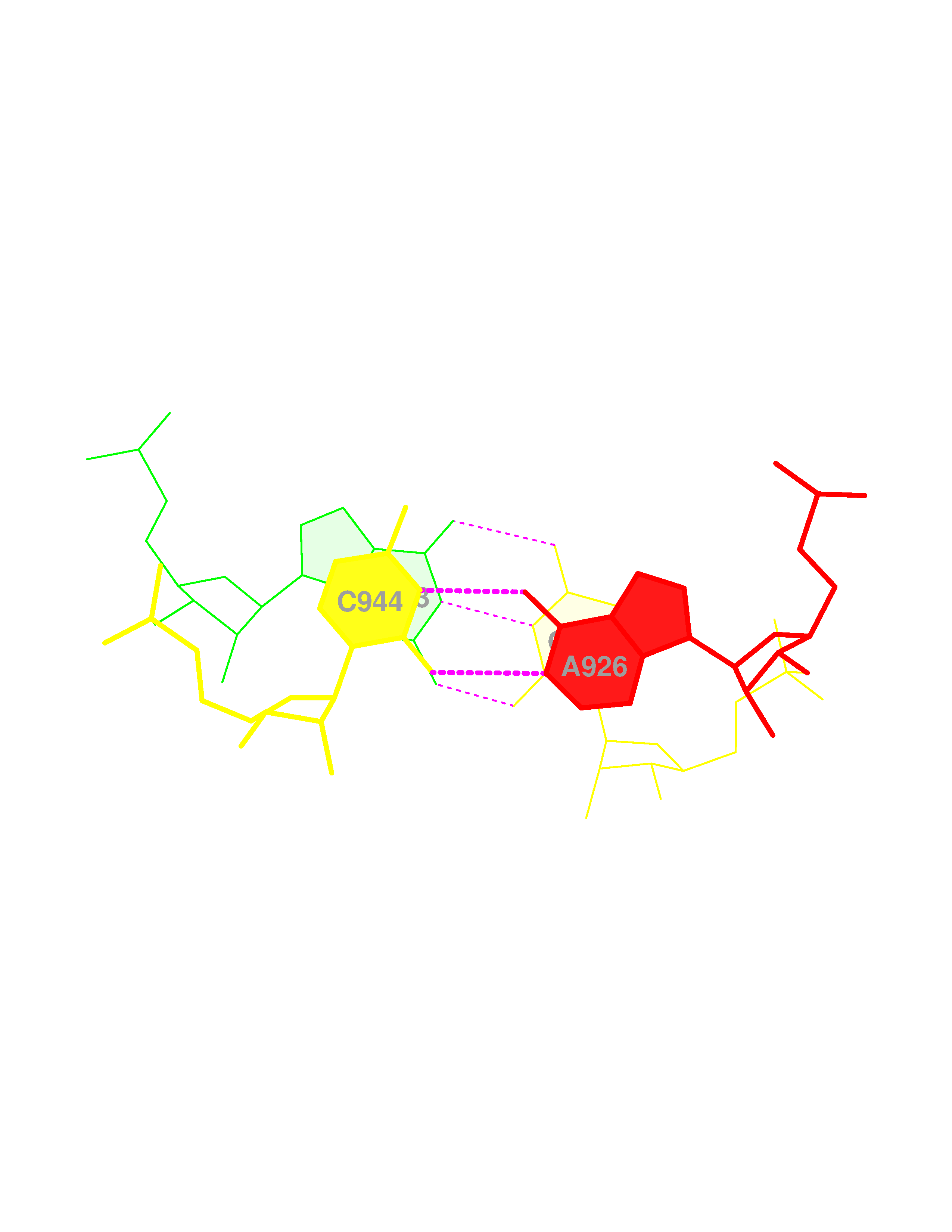
Cтекинг-взаимодействия