Нуклеотидные банки данных
-
Описание сборки
Для дальнейшей работы был выбран геном пекарских дрожжей (Saccharomyces cerevisiae) .
Это известный модельный организм из отдела сумчатых грибов (Ascomycota). На примере данного организма можно изучать многие процессы свойственные эукариотам. S. cerevisiae являются факультативными анаэробами, что позволяет исследовать на этом организме дисфункцию митохондрий. Примечательно, что пекарские дрожжи — первый эукариотический организм, чей геном был полностью секвенирован. Геном S. cerevisiae представлен 16 хромосомами и мтДНК.
По запросу "Saccharomyces cerevisiae (baker's yeast)" было найдено 1688 различных сборок, из них 29 являются полногеномными (filter -> assenmbly level -> complete). В соответствии с информацией из файла README_assembly_summary.txt это означает, что все хромосомы без промежутков и не имеют ни одной серии из 10 или более неоднозначных оснований (N), нет неразмещенных или нелокализованных скафолдов и все ожидаемые хромосомы присутствуют (т. е. сборка не отмечена как имеющая частичное представление генома). Плазмиды и ДНК органелл могут быть или не быть включенным в сборку, но если присутствуют, то последовательности не имеют пропусков. Выбираю сборку R64 - она является референсной, т. е. отобранной вручную высококачественной сборкой генома, которую определелили в качестве стандарта, с которыми сравниваются другие данные.
-
Характеристики выбранной сборки
Таблица 1.
Название сборки
R64
AC (Genbank)
GCA_000146045.2
AC (RefSeq)
GCF_000146045.2
Уровень сборки
Complete Genome
Общий размер генома
12.1 Mb
Число скэффолдов
16
Скэффолд N50
924.4 kb
Скэффолд L50
6
Число контигов
16
Контиг N50
924.4 kb
Контиг L50
6
Параметр N50 равен длине контига (скэффолда), для которого половина (50%) всех нуклеотидов сборки содержится в контигах (скэффолдах) такой и большей длины. Параметр L50 равен наименьшему числу контигов (скэффолдов), в которых содержится половина (50%) всех нуклеотидов сборки.
-
Скачивание файлов
-
3.1 Обязательное задание
Из базы данных RefSeq были скачаны необходимые файлы:
- protein.faa - последоватльности белков в формате fasta
- genomic.fna - последовательность ДНК генома
- genomic.gbff - аннотированный геном
- 3.2 (7) Дополнительноe задание
- assembly_report.txt - информация о сборке (организм, геном, сведения о последовательностях)
- assembly_stats.txt - статистика сборки (длина, длина без гэпов, количество контигов и скэффолдов, contig-N50, scaffold-L50, scaffold-N50, scaffold-N75, и scaffold-N90)
- cds_from_genomic.fna - кодирующие последовательности, предсказанные на основе генома
- feature_count.txt - статистика особенностей генома на основе GCF_000146045.2_feature_table.txt
- feature_table.txt - таблица особенностей генома
- genomic.gff - аннотации последовательностей генома, показывают расположение и характеристики генетических элементов в геноме.
- genomic.gtf - аннотации последовательностей генома, который показывает структуру генов и транскриптов в геноме
- protein.gpff - белки с ссылками на источники аннотации
- rna.fna - последовательности РНК
- rna.gbff - РНК с ссылками на источники аннотации
- rna_from_genomic.fna - РНК, предсказанные на основе генома
- translated_cds.faa - автоматическая трансляция белков из GCF_000146045.2_cds_from_genomic.fna
-
3.1 Обязательное задание
- Поисковые системы
-
4.1 База данных NCBI
Цитохром c — это небольшой гем-содержащий белок, расположенный в межмембранном пространстве митохондрий. Он является ключевым компонентом дыхательной цепи переноса электронов. У дрожжей не один, а два гена, кодирующих цитохром c - CYC1, CYC7. Однако у млекопитающих это лишь CYCS.
Все последовательности цитохрома c:
- "cytochrome c"[All Fields] AND "Saccharomyces cerevisiae"[Organism] - 1260
- "cytochrome c"[All Fields] AND "Saccharomyces cerevisiae"[Organism] AND genbank[filter] - 1177
- "cytochrome c"[All Fields] AND "Saccharomyces cerevisiae"[Organism] AND refseq[filter] - 68
-
4.1 База данных NCBI
-
Только в мРНК:
- "cytochrome c"[All Fields] AND "Saccharomyces cerevisiae"[Organism] AND "biomol mrna"[Properties] - 59
- "cytochrome c"[All Fields] AND "Saccharomyces cerevisiae"[Organism] AND "biomol mrna"[Properties] AND genbank[filter] - 7
- "cytochrome c"[All Fields] AND "Saccharomyces cerevisiae"[Organism] AND "biomol mrna"[Properties] AND refseq[filter] - 52
-
Только геномные последовательности:
- "cytochrome c"[All Fields] AND "Saccharomyces cerevisiae"[Organism] AND "biomol genomic"[Properties] - 1201
- "cytochrome c"[All Fields] AND "Saccharomyces cerevisiae"[Organism] AND "biomol genomic"[Properties] AND genbank[filter] - 1035
- "cytochrome c"[All Fields] AND "Saccharomyces cerevisiae"[Organism] AND "biomol genomic"[Properties] AND refseq[filter] - 16
-
Для человека:
- "cytochrome c"[All Fields] AND "Homo sapiens"[Organism] - 10630
- "cytochrome c"[All Fields] AND "Homo sapiens"[Organism] AND "biomol mrna"[Properties] AND genBank[filter] - 7779
- "cytochrome c"[All Fields] AND "Homo sapiens"[Organism] AND "biomol mrna"[Properties] AND refseq[filter] - 376
- "cytochrome c"[All Fields] AND "Homo sapiens"[Organism] AND "biomol genomic"[Properties] AND genBank[filter] - 1804
- "cytochrome c"[All Fields] AND "Homo sapiens"[Organism] AND "biomol genomic"[Properties] AND refseq[filter] - 208
-
Для человекa, поиск по конкретным генам:
- "CYCS"[Gene Name] AND "Homo sapiens"[Organism] - 37
- ("CYCS"[Gene Name] OR "CYCL1"[Gene Name] OR "CYCL2"[Gene Name]) AND "Homo sapiens"[Organism] - 47
- "cytochrome c"[All Fields] AND "Homo sapiens"[Organism] AND "complete cds"[All Fields] - 196
- "cytochrome c"[All Fields] AND "Homo sapiens"[Organism] NOT "partial"[All Fields] - 9332
- 4.2 Поиск в ENA
-
Цитохром c в мРНК у дрожжей:
- tax_tree(559292) AND description="cytochrome c" AND mol_type="mrna" - 0
- tax_tree(4932) AND description="cytochrome c" AND mol_type="mrna" - 0
-
Цитохром c у человека:
- tax_tree(9606) AND description="cytochrome c" - 10923
- tax_tree(9606) AND description="cytochrome c" AND mol_type="mrna" - 9584
- >tax_tree(9606) AND description="cytochrome c" AND mol_type="genomic dna"- 1335
-
4.3 Поиск в DDBJ -
Цитохром c у человека:
- (MolecularType:(mRNA)) AND (Definition:(cytochrome c)) AND (Organism:(Homo sapines)) - 30
- (MolecularType:(mRNA)) AND (Definition:(cytochrome c)) AND (Organism:(Homo sapines)) - 30 *совпадения числа находок меня конечно смутило, но они в каждом из случаев разные.
- (MolecularType:(mRNA)) AND (Definition:(cytochrome c)) AND (Organism:(Saccharomyces cerevisiae)) - 2
- Геномы органелл
На основе информации из файла assembly_report.txt получена информация о наличии в сборке генома митохондрий. Митохондриальная ДНК отнесена к отдельной единице сборки (Assembly-Unit) с названием "non-nuclear" (внеядерная). Ей соответствует запись GCF_000189485.1. В самой таблице, где перечислены все хромосомы, митохондрия указана под именем MT. Поиск информации происходил по NC_001224.1 - конкретному идентификатору митохондриальной последовательности. Также важно добавить, что основная сборка имеет идентификатор GCF_000146045.2.
Таблица 2.
Идентификатор записи (ACCESSION)
GCF_000189485.1
Тип органеллы
митохондрия
AC (RefSeq)
GCF_000146045.2
CDS
19
Число генов рРНК
2
Число генов тРНК
24
Число генов других нкРНК
1
Число псевдогенов
0
Некодирующая РНК RPM1 является частью комплекса митохондриальной РНКазы P, отвечает за процессинг 5’-конца тРНК.[1]
-
Построение графика фрагмента генома
На основе информации из файла assembly_report.txt построены графики ссылка на код, показывающие убывание длины каждого фрагмента генома, а также статистика генома На графике отмечена точка, отвечающая контигу, на который указываются величины N50 и L50.
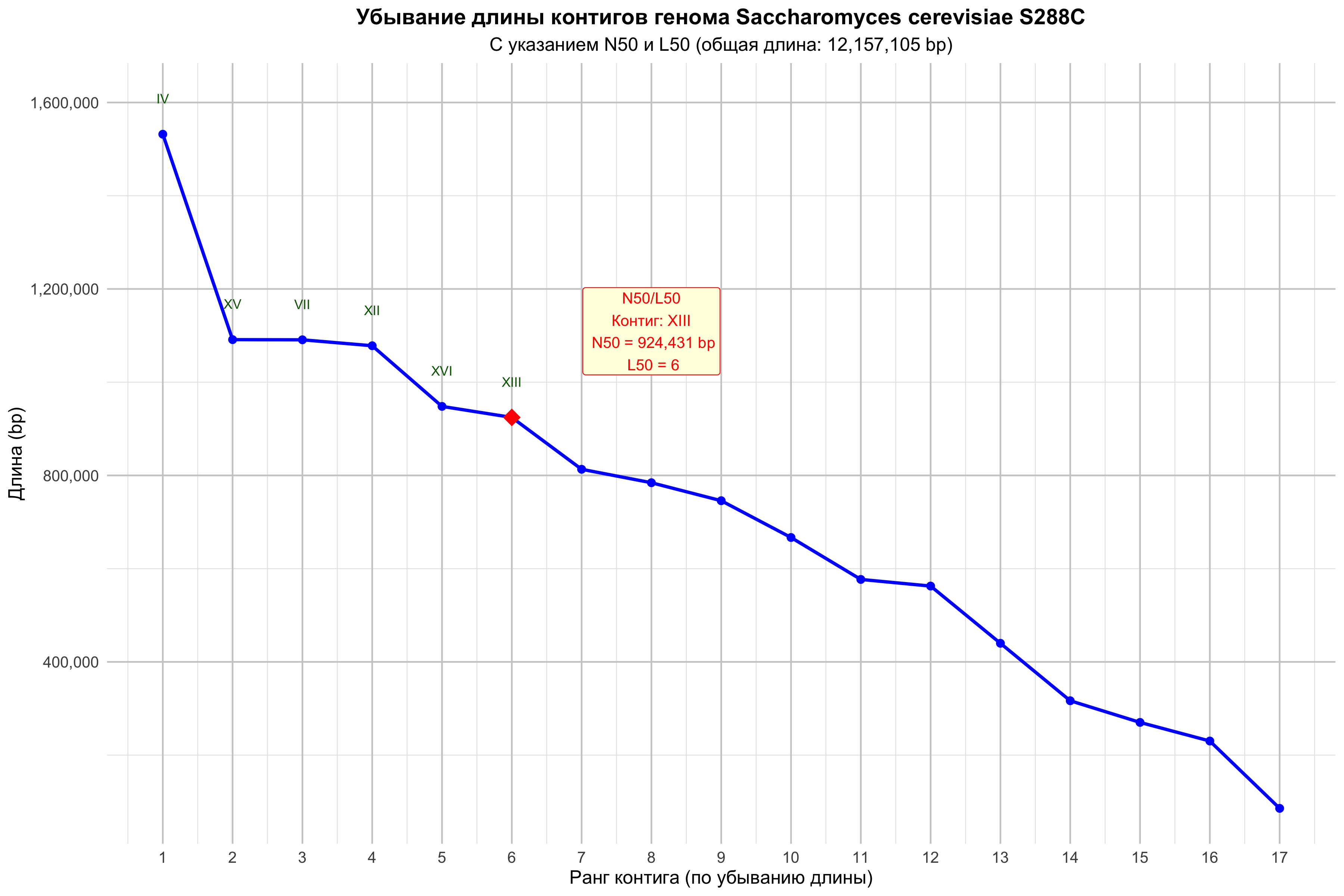 Рис.1 график убывания длин
Рис.1 график убывания длин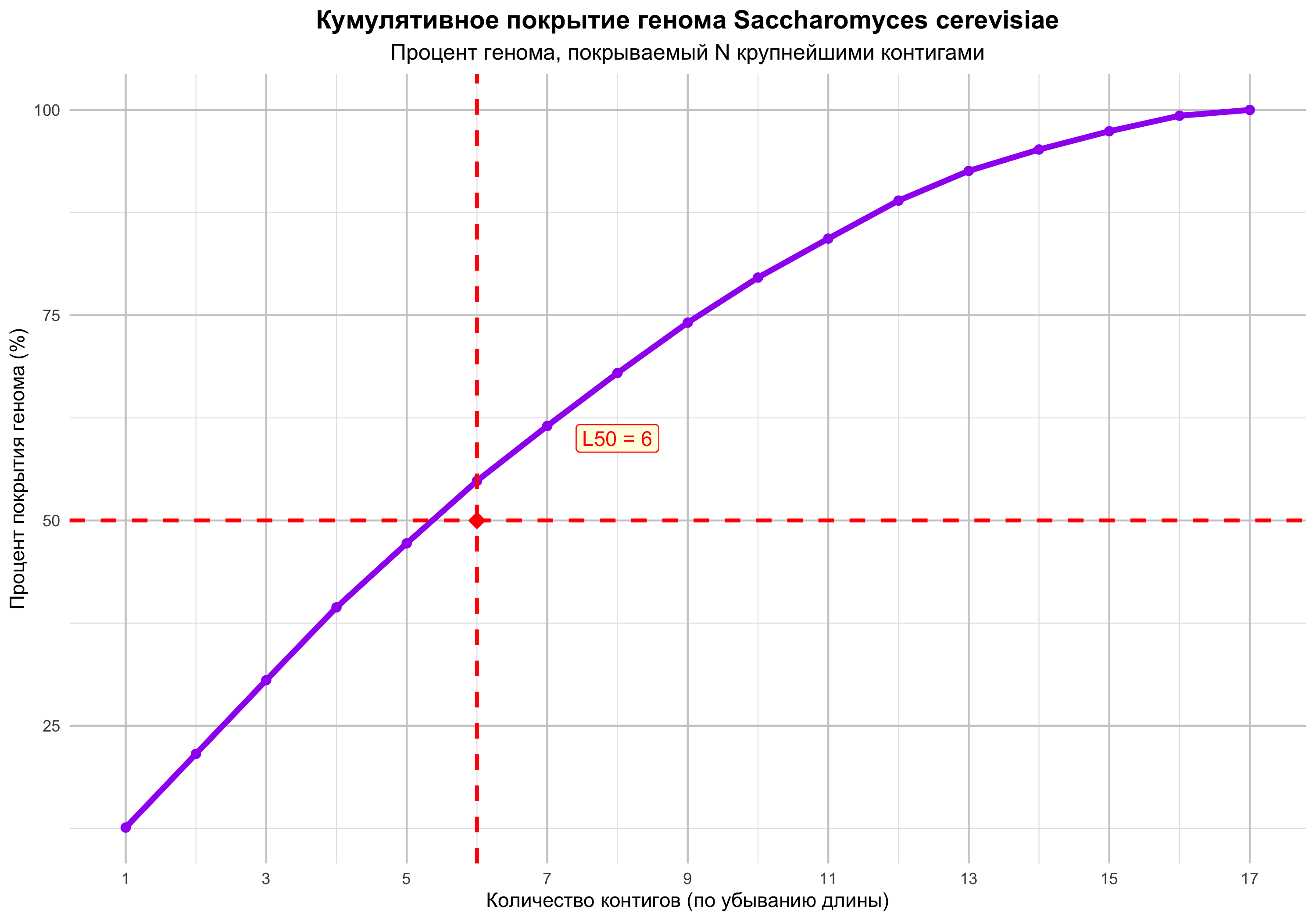 Рис.2 кумулятивное покрытие
Рис.2 кумулятивное покрытие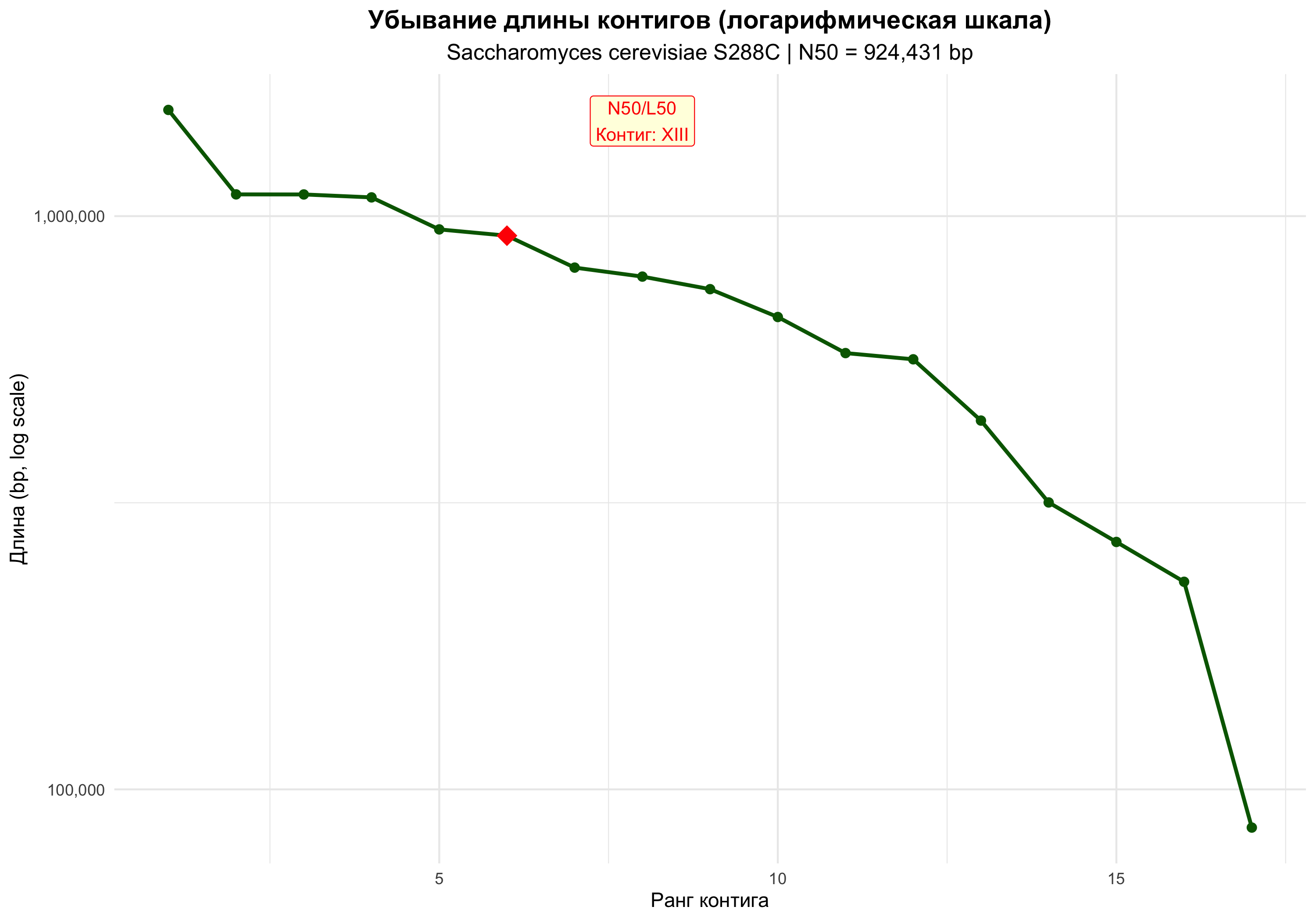 Рис.3 логарифмическая шкала
Рис.3 логарифмическая шкалаПо графику 1 из непрерывности можно судить об отличном качестве сборки. Крупнейшие хромосомы: IV (1.53 Mb), XV (1.09 Mb), XII (1.08 Mb). Средние: VII (1.09 Mb), XIII (924 kb), II (813 kb). Мелкие: I (230 kb), VI (270 kb), MT (86 kb). Первые 3 контига значительно крупнее остальных. После 7-го контига спад становится более плавным.
На графике 2 можно увидеть эффективность покрытия генома, точки перегиба - резкое замедление роста после L50 показывает переход от крупных к средним контигам. Крутизна кривой = качество сборки. Крутая начальная часть - несколько крупных контигов быстро покрывают большую часть генома. Плавный хвост - много мелких контигов добавляют немного к покрытию.
График 3 показывает, что размеры хромосом уменьшаются по закону, близкому к экспоненциальному. Все хромосомы теперь равномерно распределены по графику, а не "скучены" в левом нижнем углу. N50 находится ближе к началу распределения. Это подчеркивает, что немногие крупные хромосомы доминируют в геноме.
-
Список литературы
- RPM1 [Saccharomyces cerevisiae S288C] - Gene - NCBI
Цитохром c в мРНК дрожжей::
