Выравнивание последовательностей
Задание 1
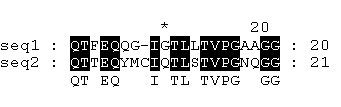
Процент идентичности = 61,90%
Процент сходства = 61,90%
Проценты идентичности и сходства данных последовательностей получились
одинаковые, потому что в матрице BLOSUM62 не оказалось ни одного положительного
значения для пар данного выравнивания, состоящих из разных
аминокислотных остатков.
alignment.msf
Задание 2
Короткая последовательность seq1 из предыдущего задания является фрагментом последовательности белка PURR_BACSU.
Чтобы узнать координаты seq1 в последовательности целого белка, я использовала программу bl2seq и получила
следующие результаты:
начало seq1 - 49-й аминокислотный остаток, конец - 68-й.
Задание 3
Сравнение двух белков: PURR_BACSU из Bacillus subtilis
и XPT_PSEMY из Pseudomonas mendocina (strain ymp).
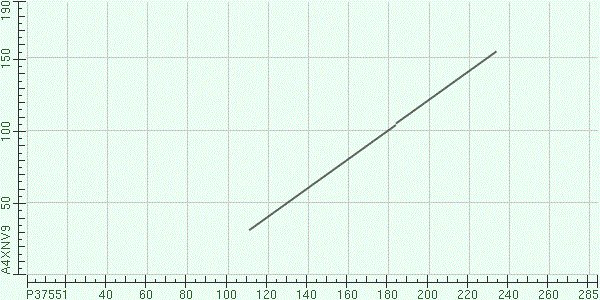
Карта локального сходства (Dot matrix view)
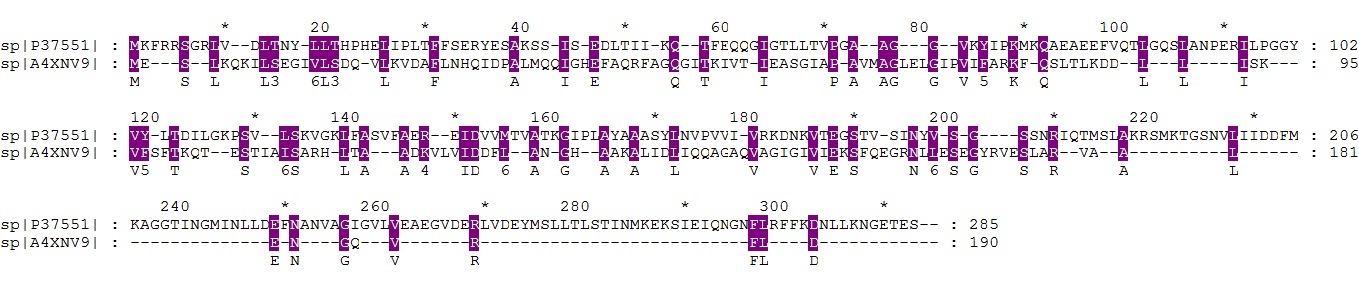
Процент идентичности (Identities) = 38/124 (31%), Процент сходства (Positives) = 69/124 (56%),
Число гэпов (Gaps) = 1/124 (0%)
Координаты выровненных участков:
| PURR_BACSU | XPT_PSEMY |
| 112 - 234 | 32 - 155 |
E-mail: yan1303@yandex.ru
Официальный сайт ФББ
Ваши предложения :)))
Проекты
Главная страница