Банк EMBL
Задание 1. Знакомство со структурой банка EMBL посредством поисковой системы SRS.
а)
Дата последнего проиндексированного в системе релиза EMBL: 8 сентября 2011 года. Число записей в нем - 71833235.
b)Список классов банка EMBL:
| CON | Constructed sequence | не проиндексирован |
| EST | Expressed Sequence Tag | не проиндексирован |
| GRV | Genome Reviews | не проиндексирован |
| GSS | Genome Survey Sequence | 32578894 |
| HTC | High Throughput cDNA sequencing | 533398 |
| HTG | High Throughput Genome sequencing | 145701 |
| MGA | Mass Genome Annotation | не проиндексирован |
| PAT | Patent | 22346593 |
| SET | Project set (EMBL WGS Masters only) | не проиндексирован |
| STD | Standard | 11737741 |
| STS | Sequence Tagged Site | 1322285 |
| TSA | Transcriptome Shotgun Assembly | 3168623 |
| WGS | Whole Genome Shotgun | не проиндексирован |
c)Cписок разделов ("Divisions") банка EMBL
| ENV | Environmental Samples | Образцы из окружающей среды | 6637887 |
| FUN | Fungi | Грибы | 2219095 |
| HUM | Human | Человек | 10687844 |
| INV | Invertebrates | Беспозвоночные | 5906093 |
| MAM | Other Mammals | Другие млекопитающие(кроме человека и мыши) | 5765536 |
| MUS | Mus musculus | Мышь домовая | 4779883 |
| PHG | Bacteriophage | Бактериофаги | 8082 |
| PLN | Plants | Растения | 17132180 |
| PRO | Prokaryotes | Прокариоты | 1467025 |
| ROD | Rodents | Грызуны | 1265394 |
| SYN | Synthetic | Искуственные | 3632878 |
| TGN | Transgenic | Трансгенные | 265545 |
| UNC | Unclassified | Неклассифицированные организмы | 7500493 |
| VRL | Viruses | Вирусы | 1177936 |
| VRT | Other Vertebrates | Другие позвоночные | 3387364 |
d*-дополнительное
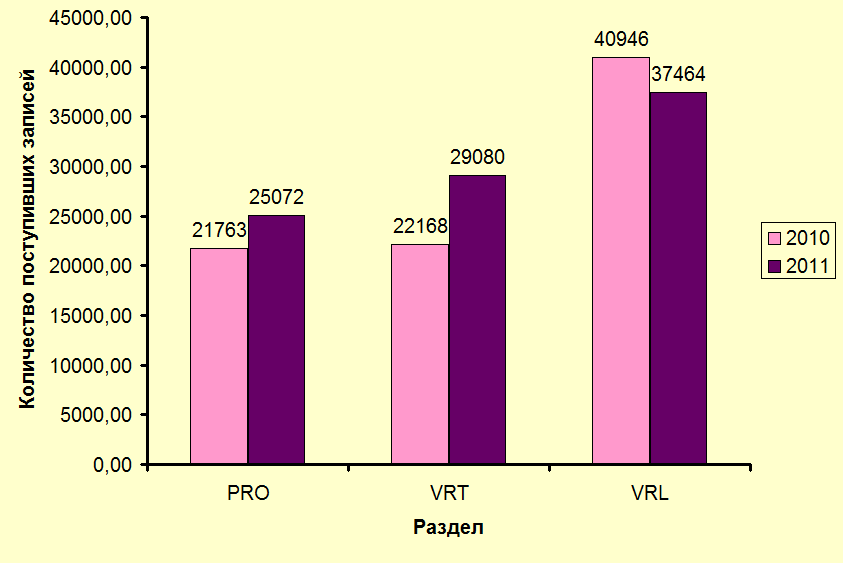
Я выбрала три раздела банка EMBL (PRO, VRT, VRL) и определила, сколько стандартных (класса STD) записей появилось в каждом из этих разделов за период с 26 февраля 2010 года по 27 мая 2010 и за тот же период 2011 года. В этом случае я использовала статистику релизов, которую нашла, пройдя по ссылке EMBL data notes на странице EMBL release. Результаты представлены ниже, в виде столбчатой диаграммы.
Если же сделать запрос обычным способом, через Query Form, (с 1-го января по 31-е марта включительно) то результаты получаются следующие:
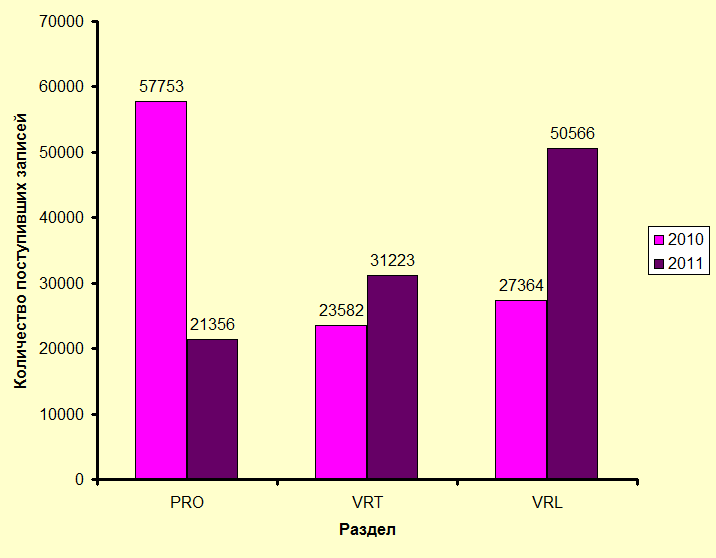
Из диаграмм видно, что скорость поступления записей в разделы прокариот и позвоночных растет, а для вирусов она снизилась.
Задание 2. Описание гена DDR в записи банка EMBL
Ген находится в комплементарной цепи, следовательно его направление - обратное.
Число кодирующих участков: 17
Длина первого кодирующего участка: 141
Длина последнего кодирующего участка: 85
Длина первого интрона: 118
Длина последнего интрона: 93
Задание 3. Нахождение белка по фрагменту гена.
Самый длинный кодирующий участок (1049547..1049793) был выделен в отдельный файл с помощью kodomo-count. Я запустила команду seqret -sask. После этого в ответ на запросы программы ввела имя входного файла (BA000025.embl), стартовую позицию вырезаемого участка, его конечную позицию. Далее нужно ввести "n", если цепь, на которой лежит участок, прямая, и "y", если обратная, а также название выходного файла.
Затем перейти по ссылке http://blast.ncbi.nlm.nih.gov/, выбрать пограмму blastх, которая по заданной нуклеотидной последовательности ищет последовательность белка; в поле "Enter Query Sequence" ввести вырезанную последовательность (или, например, загрузить fasta-файл с локального компьютера); в поле "database" выбрать банк SwissProt и нажать "BLAST".
Результаты поиска: Были получены последовательности с процентами идентичности 90%,91%, а также два результата с процентом идентичности 100%, и одинаковыми низкими значениями E-value=1e-52. При этом второй из этих двух белков оказался именно тем, что описан в файле BA000025.embl - Q08345 (Домен-содержащий рецептор 1 дисковидного эпителия) (/db_xref="UniProtKB/Swiss-Prot:Q08345"). Участок этого белка с 285 по 366 остаток соответствует моему экзону.
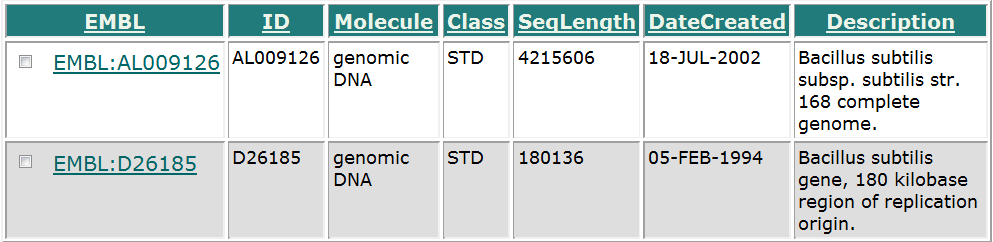
Задание 4. Ссылки из записи банка Swiss-Prot на записи банка EMBL.
E-mail: yan1303@yandex.ru
Официальный сайт ФББ
Ваши предложения :)))
Проекты
Главная страница