Геномные браузеры
Задание 1. EnsEMBL
Для начала я попробовала поискать по последовательности участок данного мне гена DDR (а именно его самого длинного
экзона, последовательность которого я получила командой seqret sw:*_BACSU; на первом занятии
второго блока).
В результате была найдена всего одна находка с идентичностью 100% и очень низким E-value=4.8e-215,
что соответствует данному участку в геноме человека.
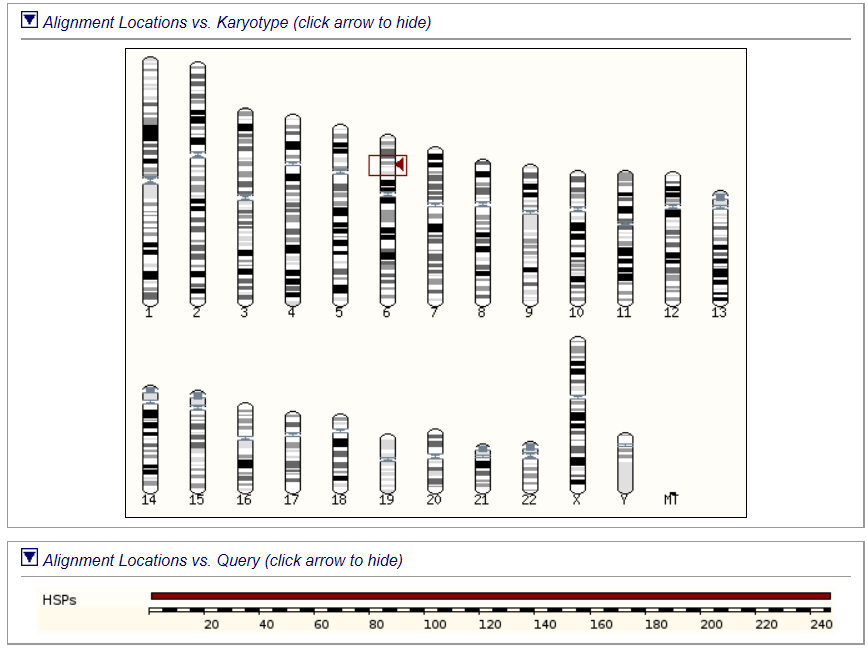
На странице результатов есть картинки, отображающие положение выравниваемых участков в хромосомах и в самом Query,
что, на мой взгляд, очень познавательно.
В данном случае, так как совпадение 100%-е, то координаты выравнивания, как и в Query, от 1 до 247.
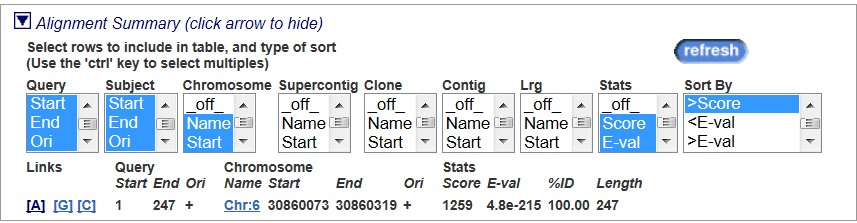
Ниже расположено поле с краткой характеристикой выравнивания. Эта характеристика оформлена в виде таблицы
(в моем случае всего из одной строки), и, что очень приятно, пользователь может сам изменять содержимое таблицы,
выбрав в соответствующих полях нужные ему характеристики, и нажав Refresh.
Собственно представленное ниже
выравнивание я получила, пройдя по ссылке "А" внизу страницы:
Query location : unnamed 1 to 247 (+)
Database location : 6 30860073 to 30860319 (+)
Genomic location : 6 30860073 to 30860319 (+)
Alignment score : 1259
E-value : 4.8e-215
Alignment length : 247
Percentage identity: 100.00
Query: 1 gtccactgtaacaacatgcacacgctgggagcccgtctgcctggcggggtggaatgtcgc 60
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct: 30860073 gtccactgtaacaacatgcacacgctgggagcccgtctgcctggcggggtggaatgtcgc 30860132
Query: 61 ttccggcgtggccctgccatggcctgggagggggagcccatgcgccacaacctagggggc 120
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct: 30860133 ttccggcgtggccctgccatggcctgggagggggagcccatgcgccacaacctagggggc 30860192
Query: 121 aacctgggggaccccagagcccgggctgtctcagtgccccttggcggccgtgtggctcgc 180
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct: 30860193 aacctgggggaccccagagcccgggctgtctcagtgccccttggcggccgtgtggctcgc 30860252
Query: 181 tttctgcagtgccgcttcctctttgcggggccctggttactcttcagcgaaatctccttc 240
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct: 30860253 tttctgcagtgccgcttcctctttgcggggccctggttactcttcagcgaaatctccttc 30860312
Query: 241 atctctg 247
|||||||
Sbjct: 30860313 atctctg 30860319
Пройдя по ссылке "Chr:6" (ее видно на рисунке), я получила еще много полезных картинок, где отмечены гаплотипы и участки, подвергавшимися корректировкам Genome Reference Consortium - GRC. , картинку с общей характеристикой хромосомы (например,%GC повторов, количества известных генов и вариаций), а также статистическую таблицу с информацией о том, сколько генов в данной хромосоме кодирует, например, разные виды РНК, и т.д.
Далее я перешла по ссылке "С" (Contig View), получила такую огромную цветастую картинку, со множеством непонятных надписей, обозначений и прочих вещей. По-моему, вполне естественно, что все это вызвало у меня дикий испуг и панику, но все же я попыталась разобраться, что сие означает =) Попробую описать свои догадки и впечатления:
Насколько я поняла, "Region in details" позволяет нам загрузить гены, их вариации, консервативные участки последовтельности, а также аннотации. На этой странице есть 3 главных панели (изображения), назначение первой из которых - показать на хромосоме фрагмент размером 1 Mb с моей находкой в центре. Моя находка расположена на хромосоме, несущей также участок с генами главного комплекса гистосовместимости (major histocompatibility complex - MHC), для которого было описано восемь основных европейских гаплотипов. Они перечислены под этим участком в "списочке" с красными метками. Следующее изображение - Top panel - дает наглядное представление того, как расположены гены в хромосоме. Полоска в верхней части этой панели, раскрашенная в темно- и светло-голубой цвета, изображает отдельные контиги. Гены, кодирующие белки, псевдогены обозначены различными цветами (пояснения - в легенде).
На третьем изображении - главная панель - треки данных(гены, выравнивания и т.д.). Треки, находящиеся над голубой линией, находятся на главной цепи ДНК, а те, которые под ней - на комплементарной. Порядок треков можно изменить с помощью "полосы прокрутки" или меню слева. Изображение можно увеличить, используя zoom. Назначение третьей картинки - показать все транскрипты Ensemble и кДНК NCBI RefSeq/ENA, выровнявшиеся с участком хромосомы, в котором расположена находка. Картинка также иллюстрирует GC-состав этого участка.
Можно узнать названия генов, их локализацию в хромосомах, координаты, тип участка последовательности, направление цепи, аннотацию.
Если искать ген по названию, то находится 7 генов DDR1. Для каждого есть 2 ссылки: одна ведет на страничку с транскриптами и кратким Gene Summary, вторая - "Region in details" (по структуре как Contig view).
На главной странице EnsEMBL можно найти обновления последнего (64) релиза, можно почитать, как использовать EnsEMBL и даже посмотреть video-tutorial, можно пользоваться различными средствами, такими как BLAST/BLAT, BioMart и др. Радует то, что можно загрузить свои данные, проанализировать их и сохранить в своем аккаунте (для этого нужно зарегистрироваться). Еще мне очень понравилось то, что с главной страницы можно перейти в блог, где представлены последние новости. Также там есть ссылки на Twitter, Facebook и другие социальные сети, и я, как пользователь Facebook, даже поставила "Мне нравится" для EnsEMBL! =)
В целом EnsEMBL оставил хорошее впечатление своим приятным интерфейсом, удобной организацией сайта. Также мне очень понравилось, что там есть странички с подсказками и инструкциями для пользователя, и даже video-tutorial на YouTube. Я бы даже сказала, что это своего рода "забота" о пользователях.
Задание 2. Другие геномные браузеры
Если сравнивать еще три браузера с EnsEMBL, то я бы сказала, что Vega вообще мало чем отличается от него по представлению информации, только она специализирована на размещении результатов проекта "HAVANA" по ручной аннотации геномов позвоночных ; в NCBI, на мой взгляд, все достаточно неплохо, обозначения и структура отличаются от EnsEMBL, а разобраться в нем так же тяжело. UCSC, по-моему, уступает трем вышеперечисленным браузерам, т.к. понять, что там для чего, вообще крайне тяжело.
E-mail: yan1303@yandex.ru
Официальный сайт ФББ
Ваши предложения :)))
Проекты
Главная страница