Исследование структуры тРНК
- Краткое описание структуры в файле 1FFY.pdb В файле приведены координаты атомов следующих молекул:
изолейцил-тРНК (1 молекула), изолейцил-тРНК-синтетаза (1 молекула)
(взяты из STAPHYLOCOCCUS AUREUS).
Для исследования была выбрана цепь Т, представляющая 1FFY тРНК со следующей последовательностью:
[917] 5' - GGGCUUGUAGCUCAGGUGGUUAGAGCGCACCCCUGAUAAGGGUGAGGUCGGUGGUUCAAGUCCACUCAGGCCCAC - 3' [972],
где 917 и 972 - номера первого и последнего нуклеотида.
На 3'-конце отсутствует триплет CCA, к которому могла бы присоединиться аминокислота. - Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи
между азотистыми основаниями ((выходной файл программы) ).
В соответствии с полученными данными
акцепторный стебель состоит из участка 1-7 и комплементарного ему участка 72-66.
Т-стебель - из комплементарных участков 49-53 и 61-65;
D-стебель - из участков 10-13 и 22-25;
антикодоновый стебель - из участков 26-32 и 38-44.
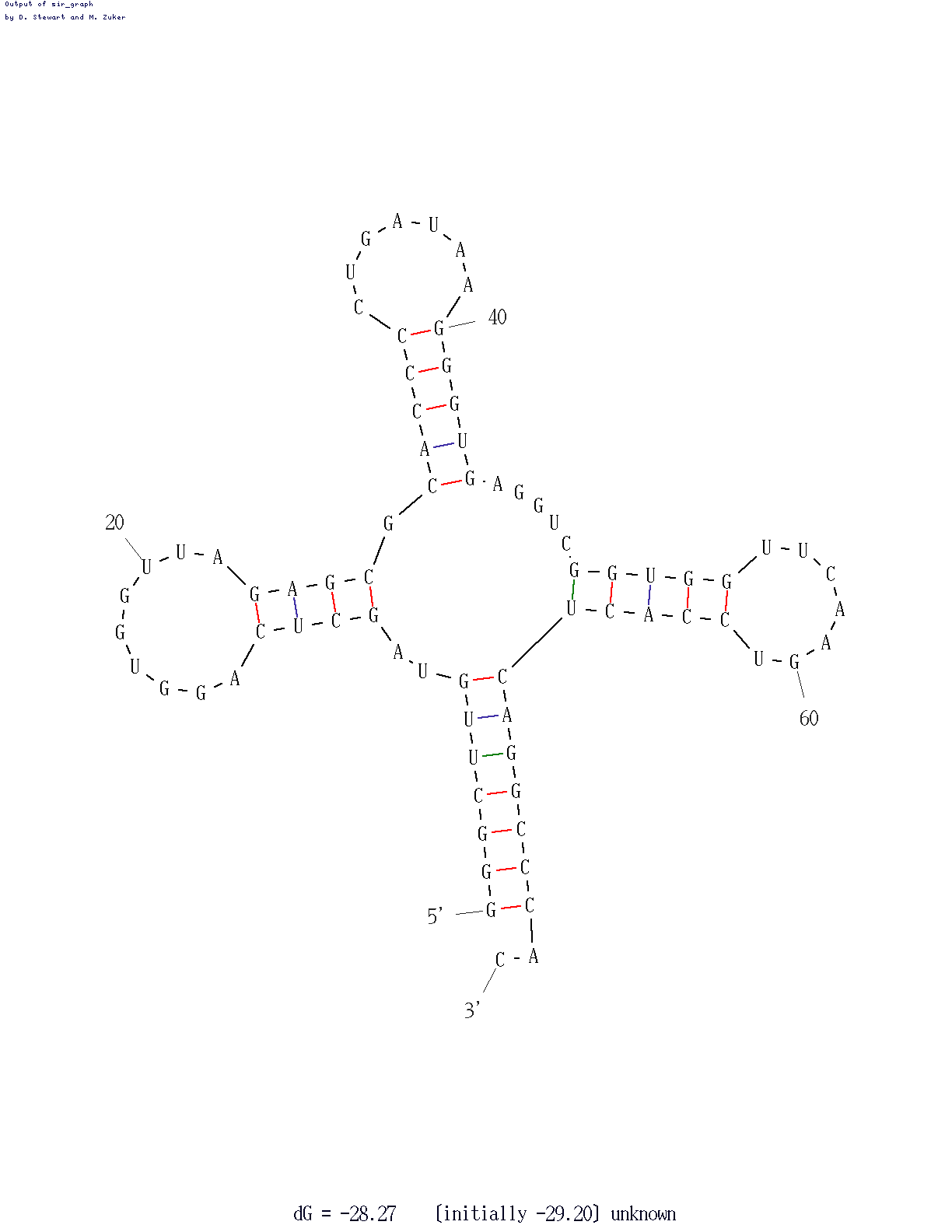
Рис.1. Вторичная структура 1FFY тРНК из STAPHYLOCOCCUS AUREUS.
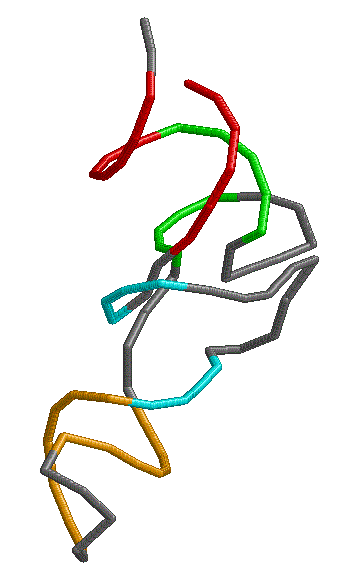
Скрипт для получения изображения:
load 1FFY_RNA.PDB
background white
select all
backbone 150
wireframe off
cpk off
color gray
select 1-7:T
color red
select 66-72:T
color red
select 61-65:T
color green
select 49-53:T
color green
select 10-13:T
color cyan
select 22-25:T
color cyan
select 26-32:T
color orange
select 38-44:T
color orange
На рис.1 - изображения остова исследуемой тРНК, где акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжевым. Структуру стеблевых дуплексов поддерживают 19 канонических и 4 неканонических пар оснований.
Например, пара A44 - G26:
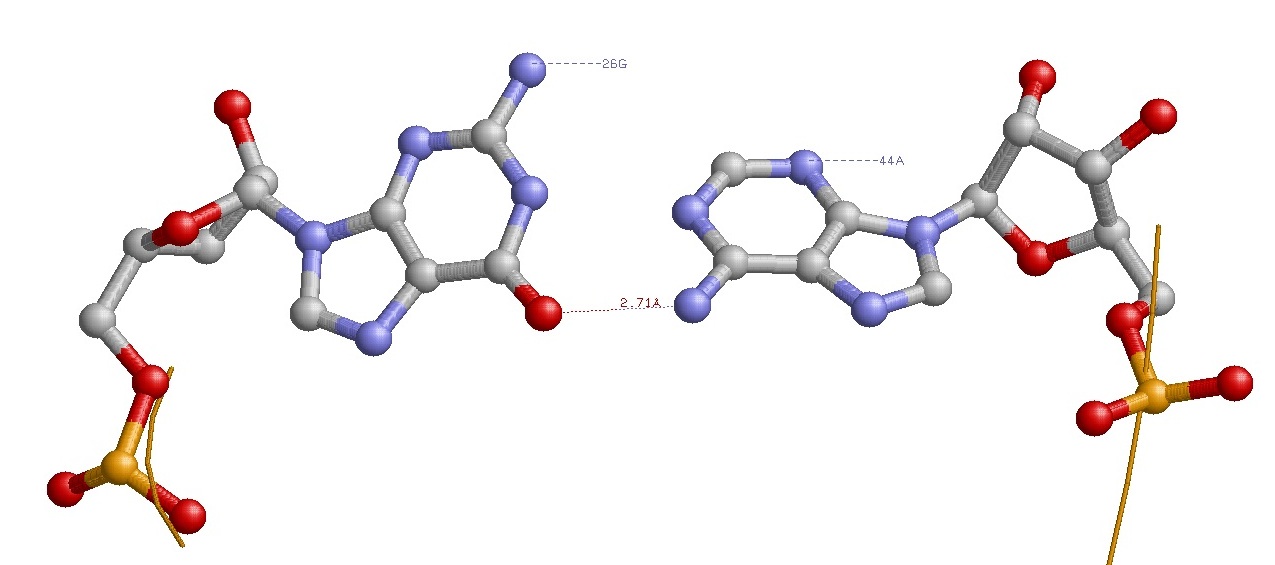
Нужно отметить также
а)наличие вариабельной петли (45-48);
б)отсутствие остатка тимидина в Т-петле;
в)отсутствие дигидроуридинов в D-петле;
*Дополнительное задание.Антикодон изолейцина в антикодоновой петле: UAG (36-35-34)
- Исследование третичной структуры
1. Была исследована возможность стекинг-взаимодействия между основаниями конца акцепторно стебля и начала Т-стебля
и между антикодоновым и D-стеблем. Площадь перекрывания на этих участках очень мала
(менее 3 кв. Å), поэтому был найден другой участок со стекинг-взаимодействием двух пар азотистых
оснований: G50-C64 и U51-A63 с площадью перекрывания 10.34 кв.Å, что является достаточно большой величиной
в сравнении с максимальным перекрыванием (11.95) и минимальным (0.00).
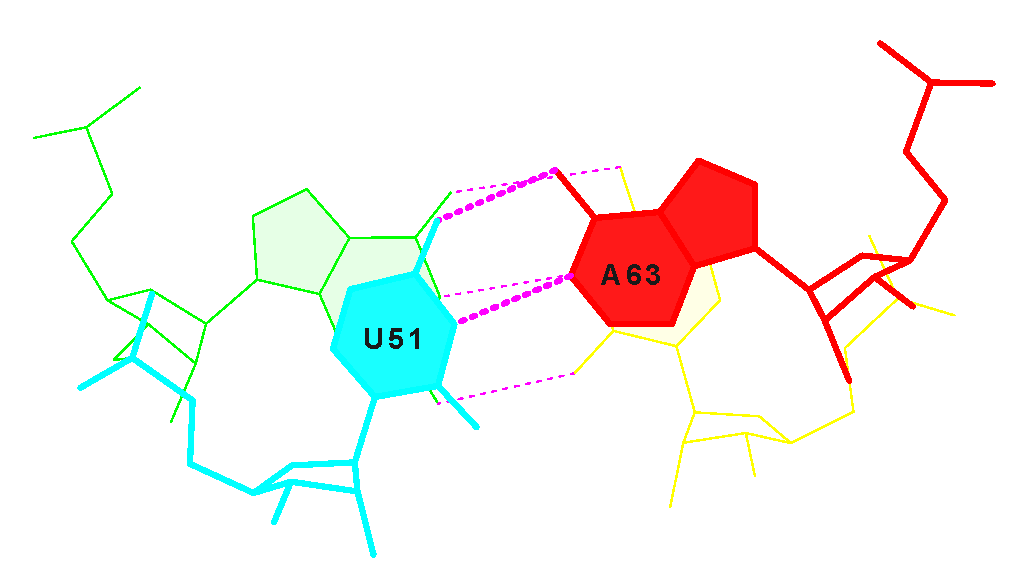
2. Также были обнаружены дополнительные водородные связи между основаниями D- и Т-петель: 54U-58A, 55U-18G, 19G-56C, 16G-17U (2 канонические и 2 неканонические).
- Предсказание вторичной структуры тРНК
Участок структуры Позиции в структуре (по результатам find_pair) Результаты предсказания с помощью einverted Результаты предсказания по алгоритму Зукера Акцепторный стебель 5'-1-7-3'
5'-66-72-3'
(7 пар)предсказано 5 пар из 7 реальных 5'-1-7-3'
5'-66-72-3'
(7 пар)D-стебель 5'-10-13-3'
5'-22-25-3'
(4 пары)предсказано 2 пары из 4 реальных 5'-10-13-3'
5'-22-25-3'
(4 пары)Т-стебель 5'-49-53-3'
5'-61-65-3'
(5 пар)ничего не предсказано 5'-49-53-3'
5'-61-65-3'
(5 пар)Антикодоновый стебель 5'-26-32-3'
5'-38-44-3'
(7 пар)предсказано 6 пар из 7 предсказано 5 пар
5'-27-31-3'
5'-39-43-3'
Общее число канонических пар нуклеотидов 19 13 19
Лучшее изображение, полученное с помощью mfold (второе по счету):
Программа einverted нашла инвертированные повороты только при значениях параметра "Minimum score threshold" 12 и меньших (последовательно ставились значения, равные 50, 40, 30 и т.д.). Надо заметить, что при значениях данного параметра <12, результаты получались такими же, как и при значении 12.
Изображение было получено с помощью web-версии программы mfold. Параметру P придавались значения 5, 10. При P=10 было получено предсказание, наиболее близкое к реальной структуре. Оно незначительно отличается от реальной структуры засчет исключения из антикодонового стебля первой и седьмой пар нуклеотидов (две крайние), которые, кстати, являются неканоническими.