Занятие 9: Трансмембранные белки
Задание1:
Таблица 1. Описание трансмембранных белков с известной 3D структурой
| PDB код | Тип
(спираль, баррель) |
Число цепей, образующих одну ТМ единицу (баррель или набор спиралей) |
Число трансмембранных участков в ТМ единице | Число остатков в одном трансмембранном участке (типичное, минимальное, максимальное) |
Толщина мембраны в ангстремах | Наклон спиралей/тяжей к нормали | Какая мембрана, организм, "код транспортера", функция белка |
| 3a3y | спираль | 3 | 13 | A:1(17), 2(22), 3(23), 4(13), 5(10), 6(22), 7(19), 8(24), 9(22), 10(19), 11(21) B:1(29) G:1(19) |
31.9 ± 1.8 Å | A: 13° B: 36° G: 11° |
организм: Squalus acanthias (Spiny dogfish) функция: Na,K-АТФаза мембрана: плазматическая мембрана Эукариот код транспортера: 3.A.3.1.6 |
| 1k9n | спираль | 5 | 5 | A: 1(22) B: 1(22) C: 1(22) D: 1(22) E: 1(22) |
30.5 ± 1.4 Å | A: 20 ° B: 23 ° C: 21 ° D: 21 ° E: 20 ° |
организм: Homo sapiens функция: регулятор кальциевой АТФ-азы, ркгуляция кальциевого насоса мембрана: мембрана эндоплазматического ретикулума код транспортера: 1.A.50.1.1 |
| 3han | спираль | 1 | 7 | A: 1(22), 2(20), 3(18), 4(23), 5(22), 6(20), 7(21) | 29.6 ± 2.2 Å | 24° | организм: Halobacterium halobium функция: транспорт протонов за счет энергии света мембрана: мембрана археи код транспортера: 3.E.1.1.1 |
| 3bry | баррель | 1 | 14 | 1(9), 2(9), 3(9), 4(9), 5(10), 6(9), 7(9), 8(9), 9(9), 10(10), 11(10), 12(9), 13(10) | 25.4 ± 1.5 Å | A: 2° | организм: Burkholderia pickettii (Ralstonia pickettii) (Pseudomonas pickettii) функция: утилизация толуола, транспорт жирных кислот мембрана: наружная мембрана грам-отрицательных бактерий код транспортера: 1.B.9.2.3 |
| 1ek9 | баррель | 3 | 12 | A: 1(11), 2(13), 3(11), 4(9) B: 1(11), 2(13), 3(11), 4(9) C: 1(11), 2(13), 3(11), 4(9) |
24.6 ± 1.4 Å | A: 51° B: 51° C: 51° |
организм: Escherichia coli функция: необходим для усиления экспрессии генов, кодирующих белки наружной мембраны грам-отрицательных бактерий мембрана: наружная мембрана грам-отрицательных бактерий код транспортера: 1.B.17.1.1 |
| 1uun | баррель | 8 | 16 | A: 1(10), 2(9) B: 1(10), 2(9) E: 1(10), 2(9) F: 1(10), 2(9) H: 1(10), 2(9) G: 1(10), 2(9) D: 1(10), 2(9) C: 1(10), 2(9) |
40.7 ± 2.1 Å | A: 54° B: 54° E: 54° F: 54° H: 54° G: 54° D: 54° C: 54° |
организм: Mycobacterium smegmatis функция: Порин. Образует водопроводящий канал, который способствует проникновению транспорту катионов, ионов железа, аминокислот, фосфат-ионов. Участвует в транспорте бета-лактамазы и некоторых гидрофильных антибиотиков. мембрана: клеточная стенка грам-положительных бактерий код транспортера: 1.B.24.1.1 |
По данным Lipid Composition Atlas толщина липидного слоя сильно варьирует и зависит от организма и от типа мембраны.
Например, у архей значения толщины липидного слоя колеблются в интервале 28-33Å ;
для наружной мембраны грам-отрицательных бактерий эти значения 20-28Å ;
для клеточной стенки грам-положительных бактерий замечено интересное исключение: толщину ок. 40Å имеет внутримембранная часть порина Mspa;
для мембраны эндоплазматического ретикулума и плазматической мембраны эукариот эти значения вообще очень сильно варьируют.
Длина (по количеству аминокислотных остатков) трансмембранных α-спиралей приблизительно в 2-2.5 раза больше, чем длина β-баррелей из-за того, что в α-спирали имеют более компактную структуру.
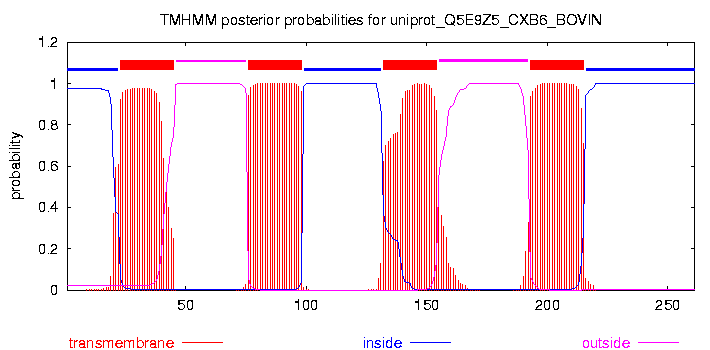
Задание 2: Аннотирование трансмембранного участка белка Q5E9Z5 (CXB6_BOVIN)
По предсказаниям UniProt:
CXB6_BOVIN ("каналосвязывающий бета-6 белок")- кластер из плотно упакованных пар трансмембранных каналов - коннексонов, через которые низкомолекулярные вещества диффундируют из одной клетки в другую. Каждый коннексон, в свою очередь, состоит из гексамера коннексинов.
Принадлежит к семейству 1.A.24 (The Gap Junction-forming Connexin Family)
трансмембранные участки: 23-45, 76-98, 132-154, 193-215
внеклеточные участки: 46-75, 155-192
цитоплазматические (внутриклеточные) участки: 1-22, 99-131, 216-261
Результат TMHMM
# uniprot_Q5E9Z5_CXB6_BOVIN Length: 261 # uniprot_Q5E9Z5_CXB6_BOVIN Number of predicted TMHs: 4 # uniprot_Q5E9Z5_CXB6_BOVIN Exp number of AAs in TMHs: 89.60724 # uniprot_Q5E9Z5_CXB6_BOVIN Exp number, first 60 AAs: 20.98675 # uniprot_Q5E9Z5_CXB6_BOVIN Total prob of N-in: 0.97697 # uniprot_Q5E9Z5_CXB6_BOVIN POSSIBLE N-term signal sequence uniprot_Q5E9Z5_CXB6_BOVIN TMHMM2.0 inside 1 22 uniprot_Q5E9Z5_CXB6_BOVIN TMHMM2.0 TMhelix 23 45 uniprot_Q5E9Z5_CXB6_BOVIN TMHMM2.0 outside 46 75 uniprot_Q5E9Z5_CXB6_BOVIN TMHMM2.0 TMhelix 76 98 uniprot_Q5E9Z5_CXB6_BOVIN TMHMM2.0 inside 99 131 uniprot_Q5E9Z5_CXB6_BOVIN TMHMM2.0 TMhelix 132 154 uniprot_Q5E9Z5_CXB6_BOVIN TMHMM2.0 outside 155 192 uniprot_Q5E9Z5_CXB6_BOVIN TMHMM2.0 TMhelix 193 215 uniprot_Q5E9Z5_CXB6_BOVIN TMHMM2.0 inside 216 261
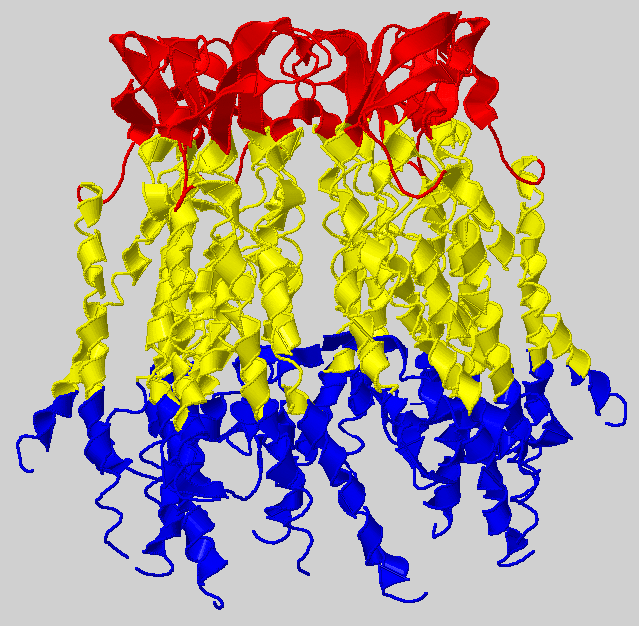
Задание 3: Сравнение с гомологом с известной структурой CXB2_HUMAN (PDB ID:2ZW3)
По данным PDBTM гомолог имеет 6 цепей, в каждой из которых по 4 трансмембранных α-спирали длиной 20, 24, 26 и 26 остатков.
Выравнивание в формате msf
Файл jar
изображение выравнивания с разметкой в JalView
Поиск в BLAST подтвердил гомолог: он оказался первым в выдаче BLAST с E-value=2e-133.
Первые два метода предсказания дали совершенно идентичные результаты. Метод сравнения с гомологом дал достаточно точное предсказание трансмембранных участков (с точностью до нескольких остатков, однако внутриклеточные и внеклеточные структуры предсказал как-то инвертированно.
Вывод:Данный белок является α-спиральным с четырьмя трансмембранными участками, предсказанными всеми методами. Внутриклеточная и внеклеточная части белка сходны между собой по размерам и не сильно большие.
Дополнительное задание 4: Проверка правила "positive inside" для гомолога CXB2_HUMAN (PDB ID:2ZW3)
Чтобы проверить правило "positive inside", я выделила красным положительно заряженные атомы аргинина и лизина (атомы азота радикала, не входящие в состав остова) и атом азота в кольце гистидина (тот, который может протонироваться); синим цветом я выделила отрицательно заряженные атомы, а именно аотмы кислорода в радикалах аспартата и глутамата.
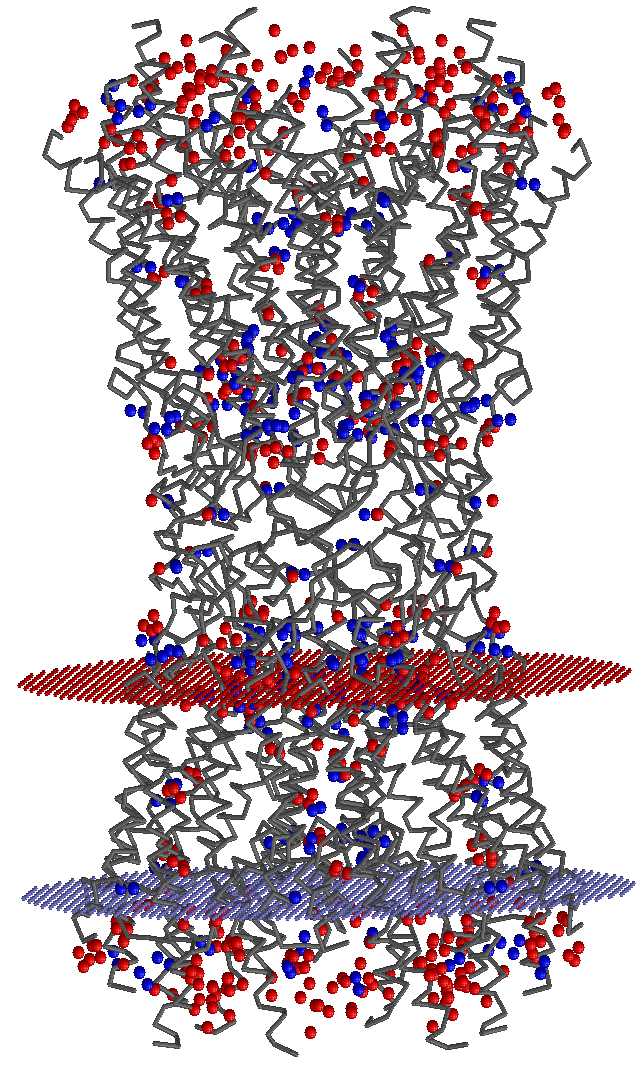
Очевидно, что на внутренней стороне мембраны очень много красных шариков - большой положительный заряд. Также видно, что количества красных и синих шариков в толще мембраны и снаружи от нее примерно одинаковы, т.е. положительный и отрицательный заряды скомпенсированы. Итак, правило "positive inside" действительно выполняется. Это нужно для закрепления белка в мембране посредством кулоновских взаимодействий (внутренняя поверхность клеточной мембраны заряжена отрицательно).
E-mail: yan1303@yandex.ru
Официальный сайт ФББ
Ваши предложения :)))
Проекты
Главная страница