Занятие 3: Алгоритмы реконструкции филогенетических деревьев.
Занятие 3
Задание1: Укоренение в среднюю точку.
Для укоренения дерева, полученного методом neighbor-joining на
прошлом занятии, в среднюю точку, я воспользовалась программой retree.
Сначала программа выдала какое-то укорененное дерево (видимо, в произвольную точку):

Затем я нажала М (что означает "Midpoint root the tree") и получила
дерево, укорененное в среднюю точку:

(IF2_CLOB1:0.36777,((IF2_LACLM:0.34903,IF2_STRPN:0.27519):0.08432, (IF2_STAES:0.21496,((IF2_LISMO:0.21104,IF2_BACAN:0.16766):0.00788, IF2_BACSU:0.11823):0.00932):0.02534):0.04092,IF2_LACAC:0.41978);
Укоренение произошло в ветвь {CLOB1,LACAC} vs {LISMO,BACAN,BACSU,STAES,LACLM,STRPN}, что не соответствует правильному дереву.
Деревья, построенные по принцмпу максимальной экономии, нельзя укоренить, потому что они не используют гипотезу молекулярных часов, т.е. расстояния в них не рассчитываются, а для рассчета средней точки как раз расстояния и нужны. Дерево, построенное с помощью алгоритма UPGMA нет смысла укоренять в среднюю точку, т.к. данный алгоритм выдает уже укорененное дерево.
Задание 2: использование внешней группы.
В качестве внешней группы был использован белок семейства IF2 из кишечной палочки (Escherichia coli) - IF2_ECOLI. Затем было построено выравнивание последовательностей программой muscle, результат подан на вход программе fprotpars, а полученное дерево обработано программой retree. В конце я убрала вручную внешнюю группу.
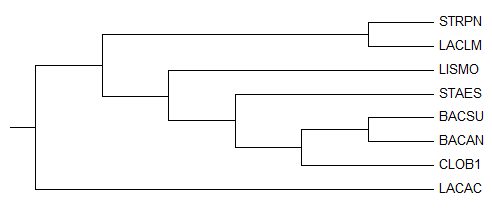
У полученного дерева правильны лишь две ветви:
{LACLM,STRPN} vs {CLOB1,LACAC,LISMO,BACAN,BACSU,STAES}
{BACAN,BACSU} vs {CLOB1,LACAC,LACLM,STRPN,LISMO,STAES};
остальное - неверно.
Задание 3: Бутстреп.
Консенсусное дерево из программы fconsense
+---------------------------------------IF2 LACAC
|
| +-------IF2 BACAN
| +--88.5-|
+-100.0-| +-100.0-| +-------IF2 BACSU
| | | |
| | +-100.0-| +---------------IF2 CLOB1
| | | |
+-------| +-100.0-| +-----------------------IF2 STAES
| | |
| | +-------------------------------IF2 LISMO
| |
| +-----------------------------------------------IF2 LACLM
|
+-------------------------------------------------------IF2 STRPN
Если сравнивать полученное только что дерево с результатом fprotpars на исходном выравнивании,
то можно смело утверждать, что алгоритм Бутстрэп не дал никакого результата, т.к. оба дерева
абсолютно идентичны: они обладают одинаковым набором ветвей. Дерево неверно.
Species in order:
1. IF2 STRPN
2. IF2 LACLM
3. IF2 LISMO
4. IF2 STAES
5. IF2 BACSU
6. IF2 BACAN
7. IF2 CLOB1
8. IF2 LACAC
Sets NOT included in consensus tree:
Set (species in order) How many times out of 100.00
.....**. 10.50
....*.*. 1.00
Среди исключенных ветвей правильных тоже, к сожалению, нет.
E-mail: yan1303@yandex.ru
Официальный сайт ФББ
Ваши предложения :)))
Проекты
Главная страница