Занятие 4: Реконструкция деревьев по нуклеотидным последовательностям. Анализ деревьев, содержащих паралоги.
Занятие 4
Задание1: Построение дерева по нуклеотидным последовательностям
| Мнемоника бактерии | АС записи EMBL, описывающей полный геном | Координаты рРНК |
| CLOB1 | CP000726 | 9282-10783 |
| LACAC | CP000033 | 59255-60826 |
| LACLM | AM406671 | 511423-512971 |
| BACAN | AE016879 | 29129-30635 |
| BACSU | M34836 | 9810-11364 |
| LISMO | AL591978 | 96266-97811(compl) |
| STAES | AE015929 | 1598006-1599559(compl) |
Для STRPN 16S rRNA не была найдена.
Выравнивание последовательностей 16S rRNA, выданное muscle, было подано на вход программе fdnadist, а полученная матрица расстояний - на вход программе fneighbor. В результате было получено следующее дерево:

В нем всего лишь две ветви совпадают с правильным деревом:
{BACAN,BACSU} vs {CLOB1,LACAC,LACLM,STRPN,LISMO,STAES},
{LISMO,BACAN,BACSU,STAES} vs {CLOB1,LACAC,LACLM,STRPN}
Все остальные - неверны. Реконструкция по аминокислотным последовательностям ни разу не дала нам более или менее похожего результата, а реконструкция по нуклеотидным последовательностям, я бы сказала, еще хуже, по-видимому потому, что среди аминокислот все-таки можно выделить некоторые функциональные группы, в которых аминокислоты обладают сходными свойствами, а в случае с нуклеотидами мы практически лишены такой возможности.
Задание 2: Построение и анализ дерева, содержащего паралоги
Последовательность действий:
Из файла proteo.fasta была создана база данных (makeblastdb -in proteo.fasta -out p5 -dbtype prot)
С помощью программы blastp были найдены гомологи отобранных белков (blasp -query CLPX_BACSU.fasta -out para -db p5 -evalue 0.001)
17 из найденных белков относились к выбранным мною восьми бактериям;
Получила последовательности этих белков в Uniprot;
Сделала их выравнивание программой muscle;
Далее воспользовалась программами fprotdist и fneighbor.
В результате получила следующее дерево:
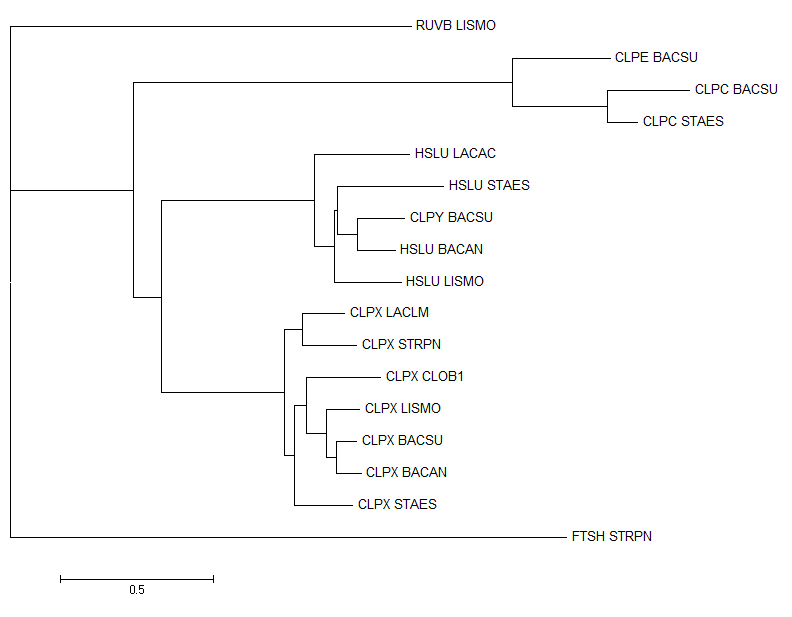
Паралогами являются: CLPE_BACSU и CLPC_BACSU
Ортологи: CLPC_BACSU и CLPC_STAES;
CLPX_LACLM, CLPX_STRPN, CLPX_CLOB1, CLPX_LISMO, CLPX_BACSU, CLPX_BACAN, CLPX_STAES
E-mail: yan1303@yandex.ru
Официальный сайт ФББ
Ваши предложения :)))
Проекты
Главная страница