Поиск в нуклеотидных банках данных, выравнивание геномов.
Задание 1. Поиск геномов в базе данных Nucleotides NCBI.
При выполнении данного задания я использовал advanced search в банке нуклеотидных последовательностей Nucleotides NCBI, в результате мой запрос выглядел следующим образом: ((genome[Title]) AND complete[Title]) AND Clostridium[Organism].
Таким образом мы получаем список всех полных геномов представителей рода Clostridium присутствующих в базе данных. Я решил выбрать для выполнения данного задания бактерий этого рода в связи с тем, что в прошлом семестре в рамках выполнения практикумов по биоинформатике я рассматривал геном Clostridium tetani. Далее, в левой части экрана, в графе "source databases" я выбрал фильтр "RefSeq" ограничив таким образом выдачу только последовательностями из это базы данных. В результате в выдаче было около 233 различных геномов. Далее просмотрев список, я выбрал два генома двух различных видов клостридий, относительно близких по длине генома, но в то же время не слишком близких. В рамках данного рода, наблюдается довольно большой разброс длин геномов (от 2 до 6 мегабаз), таким образом близость геномов по длинне, может рассматриваться как признак относительно большего родства двух рассматриваемых видов, проведя несколько раз выравнивание последовательностей длин геномов разных пар клостридий, я пришёл к выводу что брать геномы различающиеся по размеру больше чем на мегабазу не очень целесообразно, в тоже время для последовательностей слишком близких по длинне Dotplot часто выглядит как сплошная линия, что указывает на высокий процент совпадений, что тоже не очень интересно. В результате для данного задания были выбраны бактерии Clostridium perfringens strain 346/91 и Clostridium botulinum strain ZJK-3, c длинами геномов 3 и 3.8 мегабаз соответственно.
Задание 2. Выравнивание с использованием blastn и megablast. Dotplot.
После того как мы выбрали организмы, остаётся только провести выравнивание и перейти к анализу результатов. Параметры выравниваний как blastn так и megablast мной не менялись. На вход программе предлагались идентификаторы последовательностей NZ_CP075964.1 (Clostridium perfringens) и NZ_CP070936.1 (Clostridium botulinum)
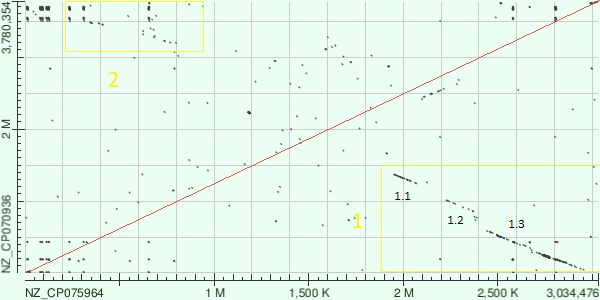
Выше вы видите dotplot схему полученную в результате обработки последовательностей алгоритмом выравнивания blastn с модификацией megablast. Красная линия посередине была нанесена мной для наглядности. На схеме отчётливо видно, что между 1.9 и 3 мегабазми последовательности Clostridium perfringens лежит участок имеющий высокую степень сходства с участком между 0.2 и 1.4 мегабазми Clostridium botulinum. Обозначим его цифрой 1 (на рисунке жёлтая), по углу наклона данного участка можно понять, что относительно Clostridium botulinum он инвертирован, поэтому я отметил его жёлтой рамкой (в дальнейшем я буду использовать жёлтую рамку для обозначения инверитированных участков). его можно визуально разделить на три участка которые мы обозначим как 1.1; 1.2; 1.3. Мы можем видеть, что через 1.1 и 1.2 можно провести условную прямую, которая будет проходить выше, чем 1.3, исходя из этого мы можем предположить, что между этими участками (1.2 и 1.3) произошла инсерция. При этом если визуально сместить 1.1 и 1.2 так, чтобы прямая проходила через все три участка, мы обнаружим что единой линии они не образуют, следовательно между 1.1 и 1.2, а также 1.2 и 1.3 существуют крупные делеции. Между 3.0 и 3.7 мегабазми Clostridium botulinum (0.1-0.8 perfringens) существует что-то вроде инвертированного участка, обозначим его цифрой 2. Увы он настолько плохо различим, что его можно принять за шум.
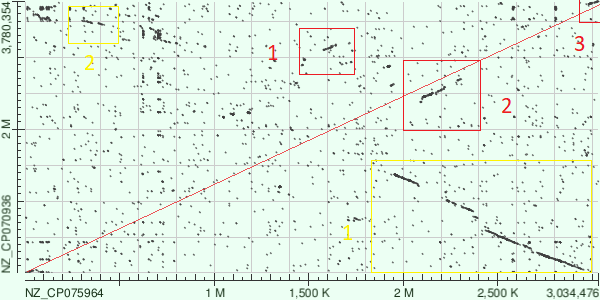
Теперь рассмотрим dotplot схему полученную в результате обработки без привлечения megablast. Поскольку шаг blastn меньше чем у megablast, мы можем наблюдать повышение точности выравнивания. Участок который мы обозначили на предыдущем dotplot'e жёлтой цифрой 1 теперь выражен значительно сильнее. Также участок отмеченный жёлтой цифрой 2, теперь пусть и слабо, но всё же выражен, так что теперь мы уже не можем принять его за шум. По направлению красной линии появилось 3 новых участка с высокой степенью сходства. Первый из них, очень сильно смещён обозначим его красной цифрой 1. Длина данного участка составляет 100 килобаз, можно конечно предположить что такое большое смещение результат инсерции, но это маловероятно, скорее всего данный фрагмент является мобильным генетическим элементом. Второй участок обозначим цифрой 2 (на рисунке красная), он относительно слабо смещён относительно линии, можно предположить что где-то перед ним есть инсерция, или что линия проведена не очень точно. Третий участок обозначим цифрой 3 он лежит на линии, так что нам не приходится говорить о делециях и инсерциях в окрестности данного участка.