Практикум 15.
Сборка генома de novo.
Для начала я получил файл исходного секвенирования бактерии Buchnera aphidicola (SRR4240379)
Триммирование.
По рекомендации я создал файл adapters.fasta состоящий из содержимого файлов папки /NGS/adapters.
Теперь мы можем удалить адаптеры, для этого мы воспользуемся командой: TrimmomaticSE -phred33 SRR4240379.fastq.gz noad.fq.gz ILLUMINACLIP:adapters.fa:2:7:7
Выдача программы: Input:7400155 Surviving:7269852 (98.24%) Dropped:130303 (1.76%)
Исходя из выданных данных мы можем утверждать, что 1.76% чтений являлись адаптерами.
Теперь мы удалим все последовательности длинна которых менее 32 нуклеотидов и нуклеотиды низкого качества. Для этого выполним команду: TrimmomaticSE -phred33 noad.fq.gz trimmed.fq.gz TRAILING:20 MINLEN:32
Результаты:
Input: 7269852 Surviving:6974267 (95.93%) Dropped: 295585 (4.07%)
Давайте теперь сравним размер файлов полученный на разных этапах очистки с исходным файлом:
167 mb SRR4240379.fastq.gz 164 mb noad.fq.gz 156 mb trimmed.fq.gz
Velveth.
Для подготовки k-меров необходимой длины (31) нам необходимо выполнить команду:
velveth Assem 31 -short -fastq trimmed.fq.gz
В результате выдача программы представляла собой папку Assem, содержащую 8 файлов: contigs.fa Graph LastGraph Log PreGraph Roadmaps Sequences stats.txt
Velvetg.
Теперь перейдём непосредственно к сборке, для этого мы воспользуемся командой: velvetg Assem.
Выдача программы:
N50: 25646 А также содержимое файла: stats.txt
Для анализа статистики воспользуемся командой: sort -nk2 stats.txt В результате мы теперь можем наглядно увидеть наиболее длинные и соответственно наиболее короткие контиги, в нашем случае:
Три самых крупных контига:
№ Длина__________________________Покрытие
6 49912 0 0 0.000000 35.907237 35.907237
9 49262 0 0 0.000000 34.772177 34.772177
5 33085 0 1 0.000000 36.259030 36.259030
Теперь отсортируем нуклеотиды по покрытию, для этого я снова использовал sort, наиболее интересная находка: 133 1 3 3 0.000000 474299.000000
Данный контиг обладает до абсурдного большим покрытием, в выдаче также присутствовало несколько контигов длины 1 также обладавших внушительными покрытиями, но этот превосходит ближайшего конкурента в 20 раз.
Megablast.
Рассмотрим выравнивания контигов: 5, 6, 9 на седьмую хромосому.
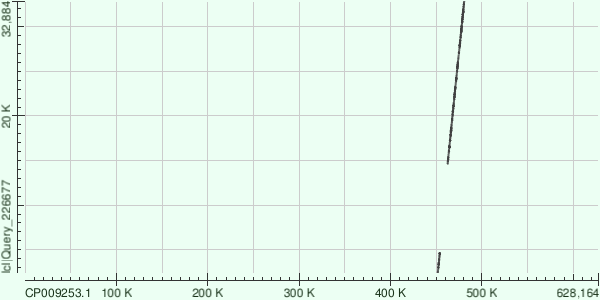
9-ый контиг.
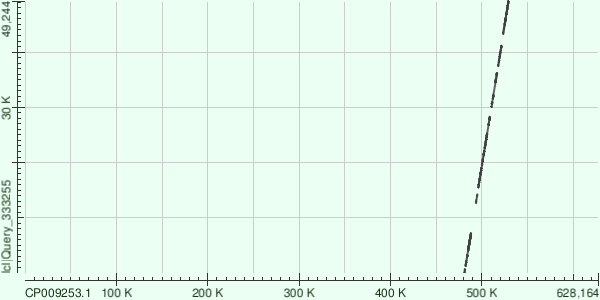
6-ой контиг.
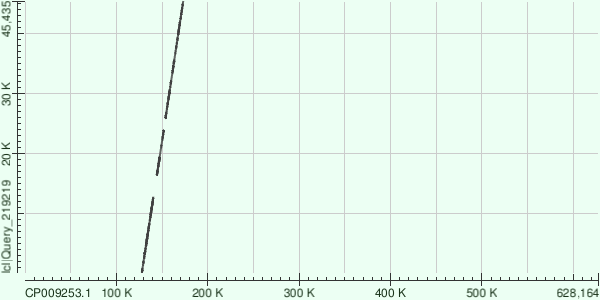
5-ый контиг.