Структуры нуклеиновых кислот.
Задание 1.
С помощью программы fiber, пакета 3DNA были получены файлы формата PDB содержащие информацию о структурах трёх молекул ДНК в различных формах, ссылки на файлы представлены ниже:
(5xGATC ДНК А-форма.) (5xGATC ДНК B-форма.) (10xGC ДНК Z-форма.)Задание 2.
Визуальное сравнение экспериментальной и сгенерированной структур.
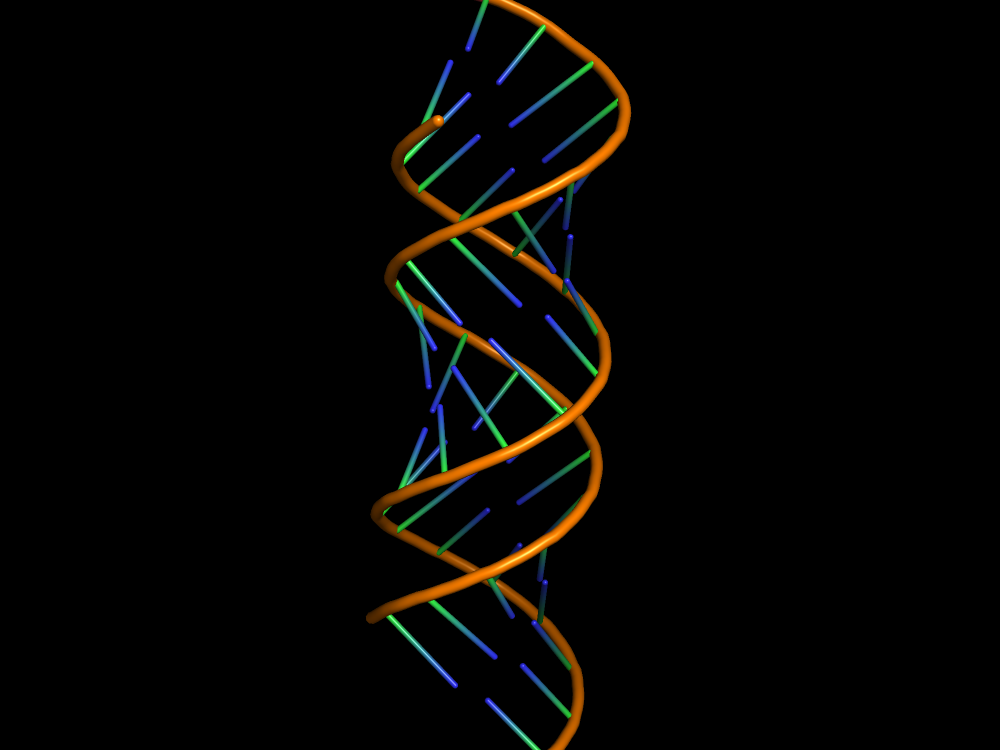
Рис. 1 Сгенерированная структура ДНК в A-форме.
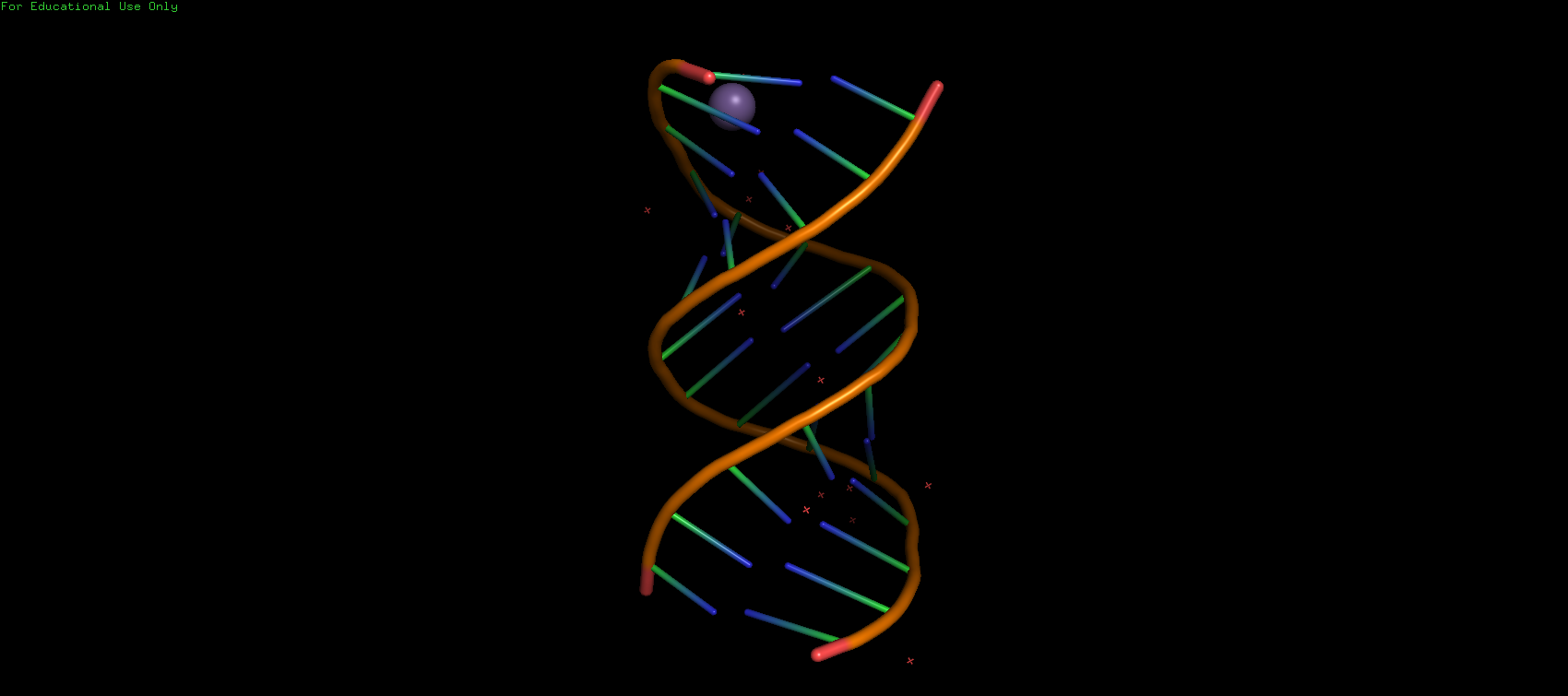
Рис. 2 Экспериментально определённая структура ДНК в A-форме.
Как легко заметить, сгенерированная программой структура является более конденсированной и симметричной нежели структура полученная экспериментально, вероятно силовое поле использованное для создания модели плохо отражает действительность. У обоих форм наблюдается чётко выраженный переход между большой и малой бороздками.
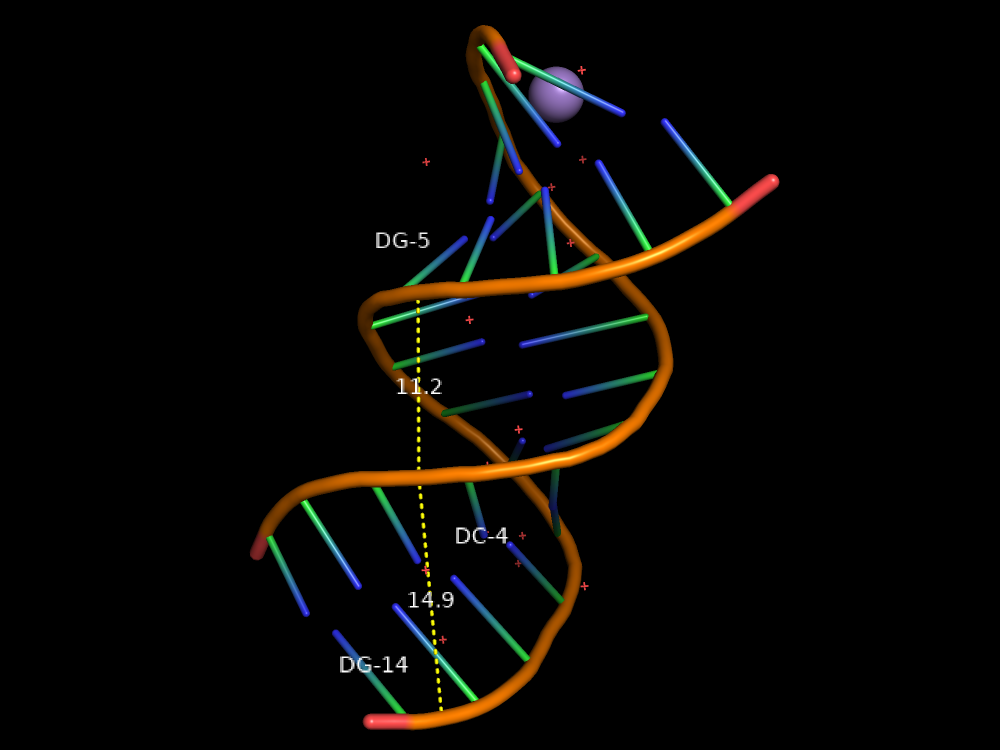
Рис. 3 Ширина большой и малой бороздок.
Ширины бороздок соответствуют расстоянию между атомами фосфора остатков DG-14 и DC-4 (большая бороздка) и DC-4 DG-5 (малая бороздка).
Пространственное расположение атомов азотистого основания (тимин DT7) относительно бороздок ДНК.
Рис. 4 Красным отмечены атомы направленные в сторону большой бороздки, синим в сторону малой.
В сторону большой бороздки обращены атомы: c7n1, c7c2, c7o2.
В сторону малой бороздки обращены атомы: c7c4, c7c5, c7c7, c7o4.
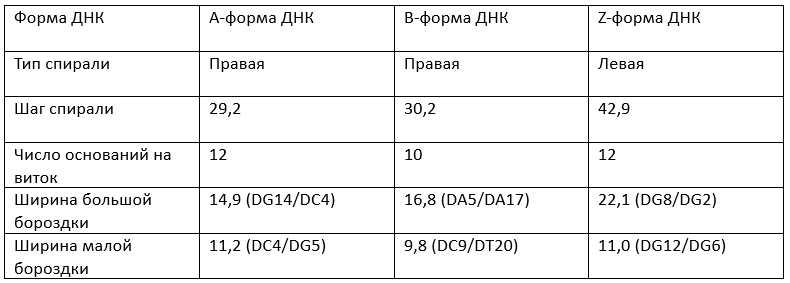
Таблица 1. Сравнение структур форм ДНК.
Задание 3.
Анализ структуры тРНК.
Средние торсионные углы молекул.
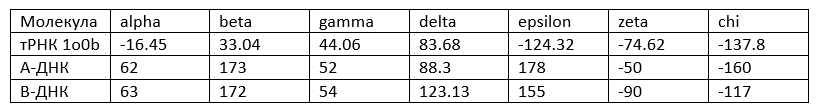
Табл 2. Сравнение средних торсионных углов нуклеотидов в тРНК и ДНК различных форм.
Как мы можем видеть торсионные углы нуклеотидов в тРНК разительно отличаются от таковых в обоих формах ДНК из-за чего определить то, в какой форме находится РНК представляется сложной задачей.
step Xp Yp Zp XpH YpH ZpH Form
1 GG/CC -1.08 8.29 2.49 -4.65 7.90 3.54 A
2 GG/CC -1.50 8.11 2.63 -4.41 8.12 2.60 A
3 GG/CC -1.99 8.50 2.21 -7.87 6.66 5.41 A
4 GU/AC -1.69 8.45 1.94 -4.40 7.96 3.43 A
5 UA/UA -1.95 8.41 1.97 -5.95 7.84 3.59 A
6 AC/GU -1.89 7.48 3.46 -4.49 6.97 4.38
7 CG/CG -1.92 8.39 2.27 -8.32 7.41 4.54 A
8 GA/UC -1.63 8.29 1.89 -3.13 7.88 3.22 A
9 AG/CU -1.70 8.36 2.27 -6.32 8.11 3.04 A
10 GG/CC -1.49 7.73 3.10 -6.13 6.85 4.72 A
11 GU/AC -0.87 6.88 3.73 -2.12 6.67 3.98
12 UU/GA 2.75 4.15 1.01 0.93 4.13 1.08
13 UA/UG --- --- --- --- --- --- ---
14 AU/UU 1.06 7.08 2.39 -1.13 6.85 3.24
15 UU/AU -0.48 8.37 2.04 -2.97 8.01 3.20
16 UC/GA -2.22 7.91 2.22 -4.33 7.70 2.83 A
17 CC/GG -2.25 8.23 2.20 -8.03 6.96 4.95 A
18 CG/CG -2.03 8.54 2.16 -7.88 6.17 6.23 A
19 GG/CC -2.17 8.26 1.82 -6.40 7.01 4.74 A
20 GC/AC -1.42 8.59 1.73 -3.26 8.30 2.86
21 CG/CA -3.24 7.18 3.56 -4.84 6.33 4.87
22 GC/GC -1.72 8.05 2.59 -4.47 7.93 2.93 A
23 CC/GG -1.52 8.43 2.55 -6.67 7.92 3.82 A
Тем не менее если обратится к команде analyze пакета 3DNA, можно обнаружить что алгоритм распознает форму тРНК как А-форму в большинстве случаев, в остальных программа просто не даёт ответа, в связи с чем можно предположить, что состояние в котором находится тРНК приближено к A-форме ДНК заметно сильнее, нежели к другим формам.
Водородные связи и стебли.

Выше вы можете видеть координаты всех стеблей полученные при помощи команды Analyze.

А здесь вы можете увидеть информацию о дополнительных водородных связях полученную при помощи той же программы.
Неканонические пары нуклеотидов.

А здесь представлены пары неканонических оснований.
Стекинг.
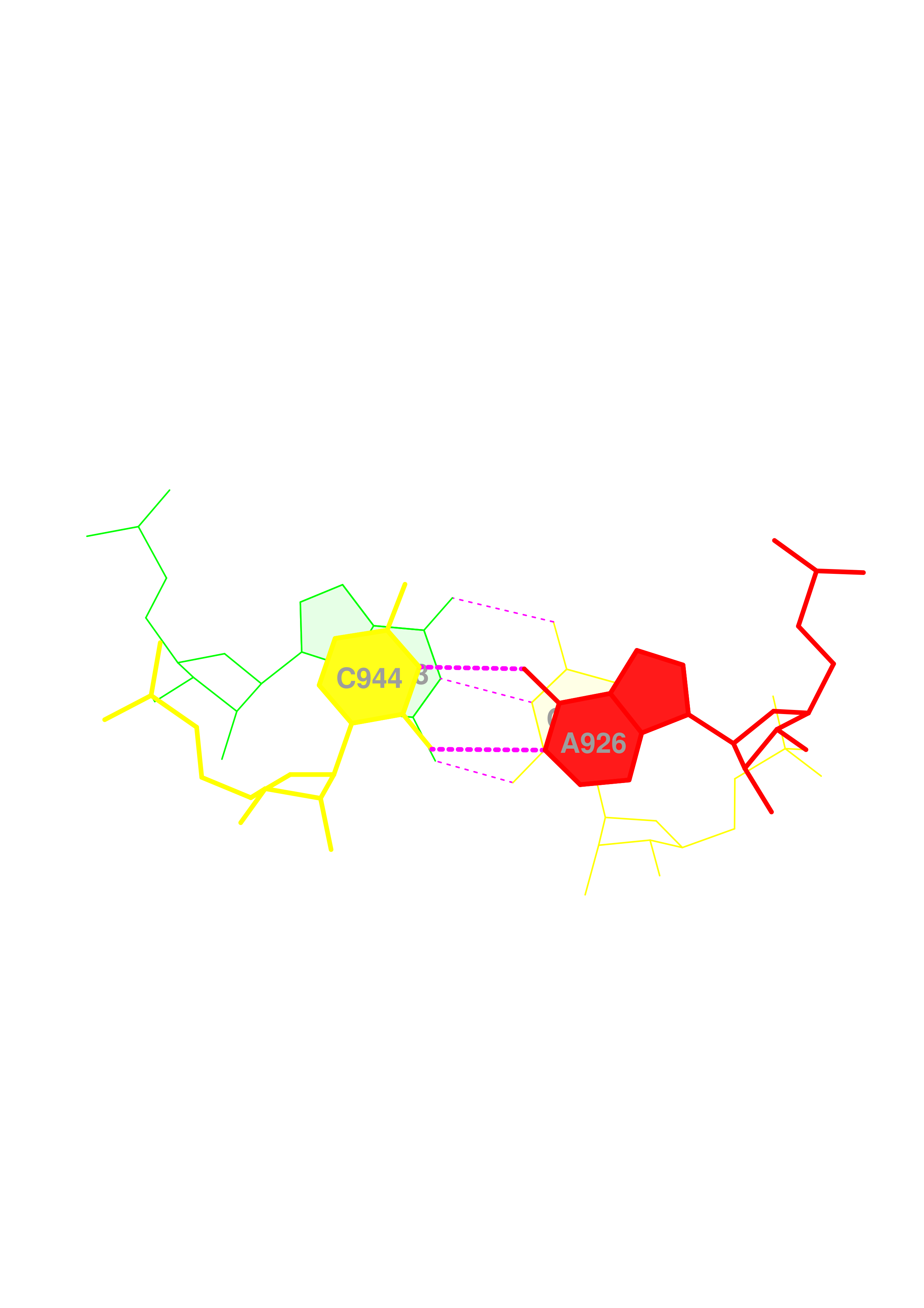
Изображение пары оснований с самым большим перекрыванием.
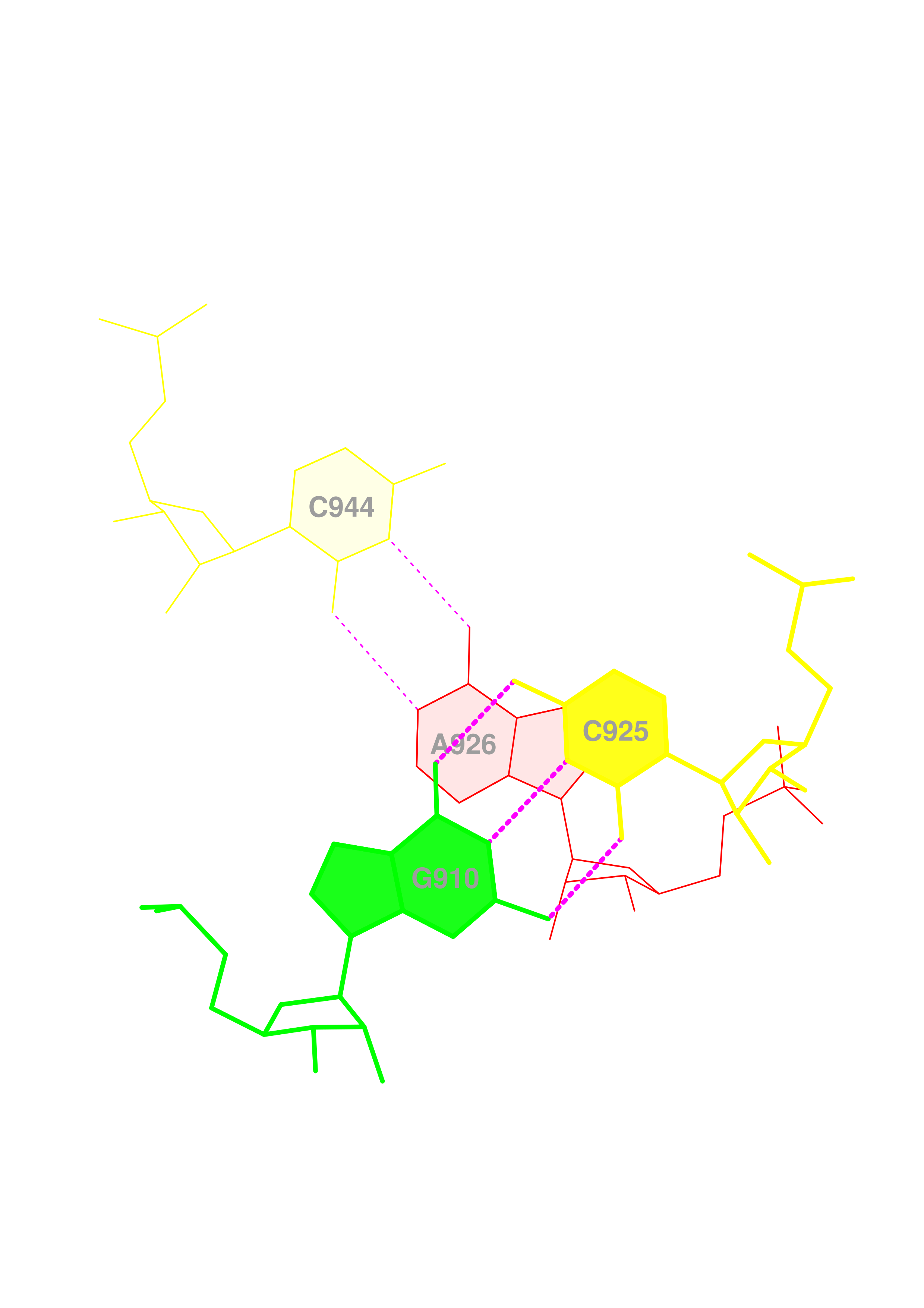
Изображение пары оснований с самым маленьким перекрыванием.