Практикум 6:
Сравнение предсказаний трансмембранных участков в бета-листовом белке.
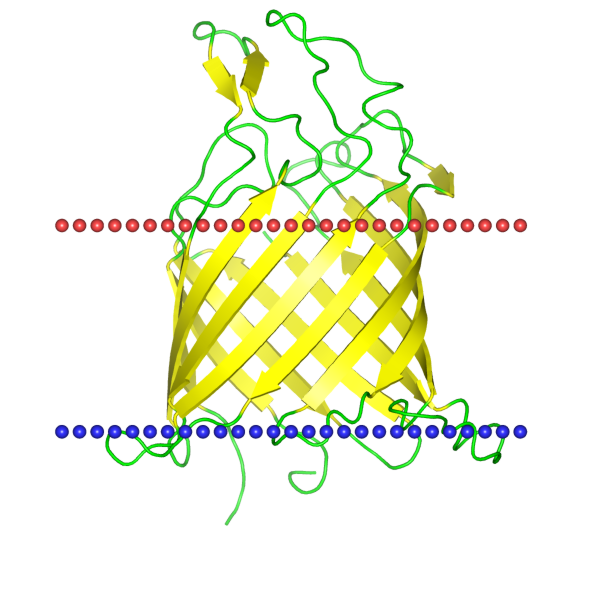
Русское название: Нуклеозид-специфичный каналообразующий белок Tsx Английское название: Nucleoside-specific channel-forming protein Tsx PDB ID: 1tly Uniprot ID: TSX_ECOLI Организм: Escherichia coli Локализация: Внешняя мембрана ЦПМ грамм-отрицательной бактерии.
Краткое описание белка.
Данный белок является субстратспецифичным каналом для нуклеозидов и дезоксинуклеозидов, имеет большую афинность к дезоксинуклеозидам, свободные азотистые основания не переносит, является составным элементом рецептора колицина K. Координаты трансмембранных сегментов(OPM): 1(13-22),2(34-44),3(48-57),4(77-85),5(101-110),6(122-132),7(137-146),8(163-173),9(181-189),10(215-224),11(227-235),12(262-272)
DeepTMHMM.
Последовательность данного белка мы получаем из UniprotKB.
Координаты трансмембранных сегментов(ТMHMM): 1(36-49), 2(54-65), 3(70-80), 4(97-108), 5(124-135), 6(141-153), 7(160-173), 8(180-193), 9(203-215), 10(233-244), 11(249-264), 12(282-293)
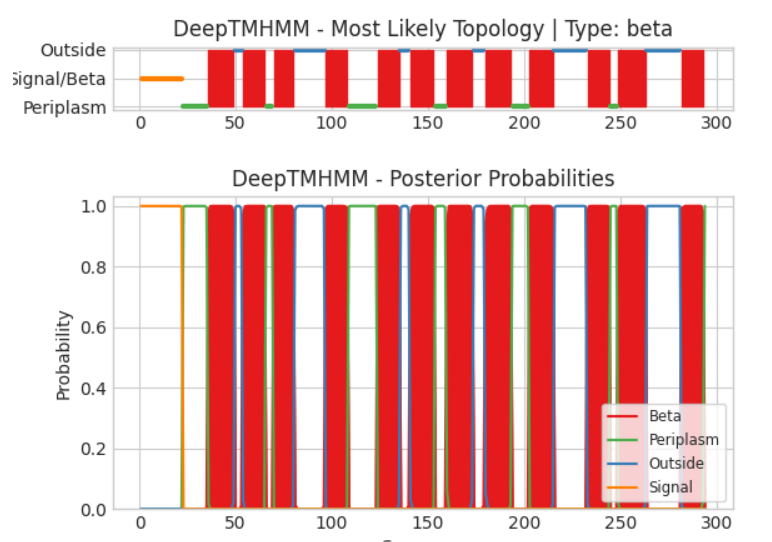
Выше вы видите перед собой графическую визуализацию результатов TMHMM, по оси x отложена вероятность принадлежности структуры определённой группе, выделенной по локализации, по оси y отложены координаты соответствующие последовательности искомого белка. Трансмембранных сегментам соответствуют элементы группы 'Beta'.
Ссылка на выдачу DeepTMHMM в формате gff3
.Сравнивая результаты выданные TMHMM с данными из OPM замечаем, что в обоих случаях предполагается 12 трансмембранных сегментов, однако первый из них (13-22 OPM) не был выявлен как трансмембранный TMHMM, а также, лишь 10 ( 1 2 3 4 5 6 7 8 9 11 OPM) из них совпадают, при этом предсказанные TMHMM сегменты, лишь частично перекрываеются с таковыми из OPM, для некоторых совпадений этот сдвиг незначителен и составляет 2 нуклеотида, для других составляет более 7 нуклеотидов, создаётся впечатление, что последовательность взятая из UniprotKB может отличаться на несколько аминокислотных остатков от использованной при анализе для OPM. Помимо "потерянного" первого сегмента, TMHMM предположила наличие трансмембранного сегмента у С-конца на позициях 282-293. Также 9 сегмент предсказанный TMHMM вообще не перекрывается с данными из OPM, а 12 сегмент из OPM не был обнаружен TMHMM. В целом создаётся картина того, что TMHMM хорошо подходит для того чтобы восстановить структуру трансмембранного сегмента у искомого белка в очень общем виде, но не является достаточно точным инструментом для того чтобы однозначно определить расположение тех или иных сегментов.
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке.
Русское название: CntI экспортер псевдопалина Английское название: Pseudopaline exporter CntI Swissprot ID: CNTI_PSEAB Организм: Pseudomonas aeruginosa Локализация: Внутренняя мембрана ЦПМ грамм-отрицательной бактерии.
DeepTMHMM.
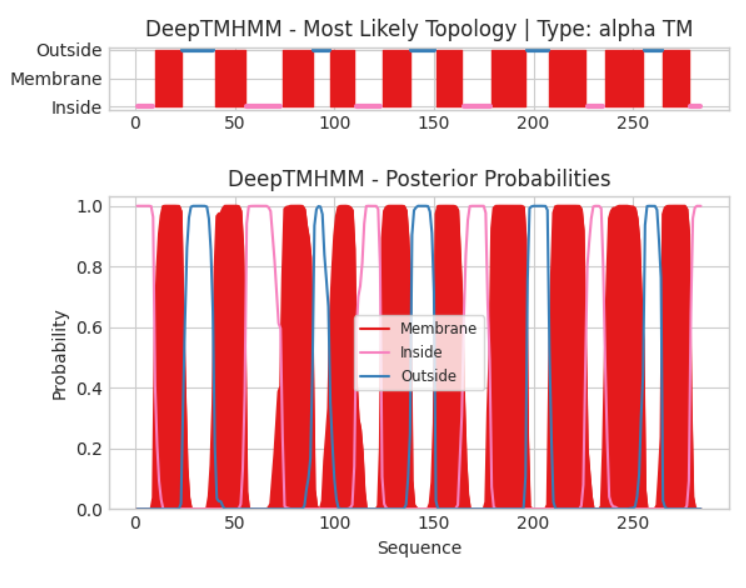
Последовательность данного белка мы получаем из UniprotKB. Координаты трансмембранных сегментов(ТMHMM): 1(10-23), 2(40-55), 3(74-89), 4(98-110), 5(124-138), 6(151-164), 7(179-196), 8(208-226), 9(236-255), 10(265-278)
Ссылка на выдачу DeepTMHMM в формате gff3
.PPM 3.0
Данные для топологии N-конца были взяты из предсказания TMHMM.

PPM3 также как и TMHMM предсказал 10 трансмембранных сегментов, причём все 10 равноценно накладываются друг на друга, но перекрываются лишь частично, 9-ые сегменты почти полностью перекрываются. В целом результаты сходятся значительно лучше чем в первом задании.