Практикум 8.
Подбор целевого контига.
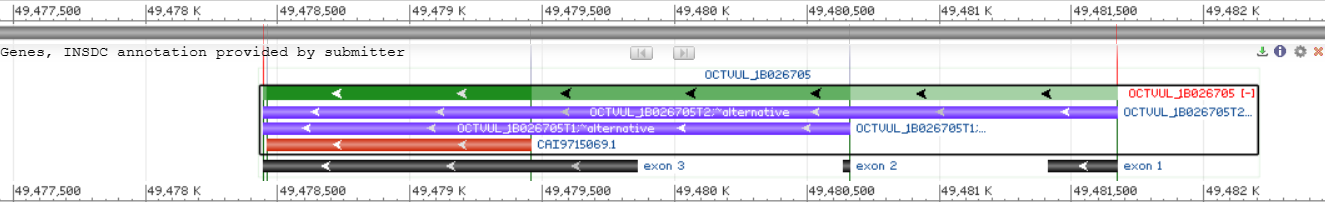
При выполнении данного задания я выбрал участок длиной 3228 нуклеотидов на первой хромосоме Octopus vulgaris, расположенный на участке 49,478,446..49,481,673. Зелёным отмечен ген, чёрным участки экзонов, синим варианты МРНК транскрибируемых с данного гена, красным CDS. Это ген продуктом экспрессии которого является ДНК-связывающий белок с "цинковыми пальцами".
Анализ последовательности с помощью алгоритмов BLAST.
Blastn.
Данный алгоритм подходит для сравнения последовательностей слабородственных и неродственных организмов, может быть использован для определения филогенетического положения организма по некодирующим участкам, поиска гомологичных последовательностей и.т.д. При анализе сиквенса данным алгоритмом я ограничил поиск таксоном Decapodiformes (taxid:215450). Эта группа моллюсков имеет общих предков с таксоном Octopoda, к которому и относится обыкновенный осьминог.
Алгоритм выдал 69/100 находок, все принадлежат к трём организмам: Thysanoteuthis rhombus, также известный как бриллиантовый кальмар (относится к Oegopsida), ещё один кальмар Sepioteuthis lessoniana (Myopsida) и каракатица Sepiola atlantica. Последовательности выданные blastn имеют высокий процент идентичности, однако покрытие не превышает 30%, но по диаграмме приложенной ниже можно видеть, что гомологичные участки выпадают как раз на CDS, так что вероятнее всего этот белок всё же присутствует и у каракатиц и кальмаров. При этом некодирующие участки почти никак не совпадают.

При использовании Megablast для той же задачи мы видим, что выдача выглядит совсем уж грустно, всего три находки с процентом покрытия 3%, это свидетельствует о том, что Megablast всё же желательно использовать только для очень близких таксонов, в пределах одного рода или отряда. При понижении параметра word size с 28 до 16 ситуация улучшается, выдача состоит из 30 находок, увы покрытие не превышает 20%, но как и в случае с blastn выпадает на участки CDS. Скорость алгоритма делает его удобным инструментом для поиска близкородственных участков в очень больших последовательностях (например при поиске ретровируса по протяжённости полного генома бактерии).
Blastx.
При анализе последовательности с помощью blastx параметров не менял, поставил выдачу первых 500 результатов. Алгоритм выдал 194/500 находок, из которых 181 принадлежит каракатице Sepia pharaonis, 11 кальмару Doryteuthis pealeii. Все выданные blastx находки являются последовательностями генов белков с цинковыми пальцами, а также белков с KRAB доменами. Данные домены участвуют в подавлении транскрипции генов, характерны для белков с цинковыми пальцами. Данный алгоритм может быть использован для анализа последовательностей которые являются (или предположительно являются генами) с целью выявить возможную функциональную нагрузку предполагаемого продукта.
Tblastx.
Увы несмотря на все мои старания алгоритм отказался выдавать что-либо вразумительное, ограничение по таксону, уменьшение выдачи до 10, ограничение по базам данных, не помогли мне заставить алгоритм работать. Сайт выдаёт ошибку, а в сообщении указано что привышен лимит оперативной памяти CPU. Алгоритм необходим для анализа эволюции белков вирусов, прокариот и других организмов для которых типичны частые мутации. Но увы, для более сложноорганизованных организмов, при использовании на малоизученных белках или для сравнения сильно эволюционно разошедшихся организмов малоэффективен.
Поиск генов рРНК в геноме Octopus vulgaris.
Использованные команды:
Создание базы данных.
-in GCA_951406725.2_xcOctVulg1.2_genomic.fna -dbtype nucl -out ../db/octopus_vulg_genomeДалее воспользуемся blastn, так-как в нашей задаче мы сравниваем гены неродственных организмов, точность blastn в данном случае делает его наилучшим вариантом для анализа.
blastn -task blastn -db ../db/octopus_vulg_genome -query 16s.fna -outfmt 7 -evalue 0.05
blastn -task blastn -db ../db/octopus_vulg_genome -query 23s.fna -outfmt 7 -evalue 0.05
Для 16S рРНК алгоритм выдал 33 находки, однако по всей видимости бльшинство из них идентичны, рассмотрим только те из них которые отличаются друг от друга. Например ген белка-траспортёра монокарбоксилата, скорее всего у этой находки самый крупный E-value, так что вероятнее всего он просто случайно оказался похож на ген рРНК, ну или автоматическая аннотация оказалась ошибочной. Среди интересных находок оказалась длинная некодирующая РНК единственная находка имеющая смысл, остальное: гипотетические белки, чего я понять не смог, так это того, почему в выдаче не оказалось 16s рРНК митохондрий, хотя в сборке, геном митохондрии имеется.
Для 23S рРНК имеется 91 находка, среди них также оказалось много последовательностей без аннотации, гипотетических белков и некодирующих РНК, снова в выдаче не оказалось рРНК митохондрии.