Мини-обзор генома Haemophilus haemolyticus
Резюме
В работе изучено несколько примечательных свойств генома и протеома бактерии Haemophilus haemolyticus: найдены координаты инициации и терминации репликации единственной хромосомы бактерии, была найдена интересная последовательность, повторяющаяся в геноме 23 раза, изучены частоты использования разных кодонов аминокислот, стопкодонов, рассмотрены длины различных белков, обнаружены некоторые закономерности в их распределении.
Довольно важно изучать свойства геномов различных микроорганизмов: манипулируя ими, можно и манипулировать и самими микроорганизмами. Работа выполнена как раз для изучения этих свойств.
Ключевые слова
Haemophilus haemolyticus; миниобзор генома H. haemolyticus; YadAlike family proteinВведение
Haemophilus haemolyticus — вид грамотрицательных бактерий (коккобациллы) как правило, непатогенных (установлено in vitro, что H. haemolyticus способна поражать клетки дыхательных путей человека), бактерии этого вида являются комменсалами человека (хотя и обнаруживаются в культурах выделенных из больных), колонизируя дыхательные пути [1]. Положение бактерии в современной систематике [2]:
Геном H. haemolyticus (представлен одной хромосомой 1,934,644 bp, 1 756 CDS, 60 генов тРНК и 19 генов рРНК [3]) (bp = пары нуклеотидов, CDS =англ., coding sequence, русск., кодирующая последовательность) в крайней степени сходен с геномом условнопатогенной Haemophilus influenzae, изза чего даже проводились исследования методов их отличия (что является важным при диагностике заболеваний, вызываемых H. influenzae). H. haemolyticus не колонизирует стерильные участки тела в отличии от ее родственника, что свидетельствует о том, что она является скорее комменсалом нежели патогенным видом. [4]
H. hnfluenzae способна вызывать пневмонию (вызывает коинфекции совместно с вирусом гриппа и риноврусами). Изначально именно данной бактерии была приписана ответственность за грипп Рихардом Пфайффером (1892), но в 20ом веке была установлена истинная вирусная природа заболевания [5]. В исследовании [1] показано, что способность H. influenzae связываться с культурой клеток дыхательных путей заметно понижается после внесения в нее H. haemolyticus.
Материалы и методы
1.Для построения графика1 “GCskew cumulative вдоль генома” использовался сервис “Genskewcc”, конкретнее говоря, его графическая версия[6]
2. Для нахождения повторяющейся последовательности использовал программу wordcount из пакета EMBOSS, для проверки вхождения последовательности в CDS, и нахождения этих CDS использовал программу, написанную на python (Программа1).
3.При подсчете частот разных кодонов использовалась Программа2, Таблица1 создана в Google Sheets
4. Гистограмма1 длин белков построена с помощью функций Google Sheets, Таблица2 создана там же
5. Подсчет частоты различных стопкодонов проделан с помощью Программы3.
Результаты и обсуждения
1. Нахождение координаты начала репликации oriC и координаты терминации репликации – ter.
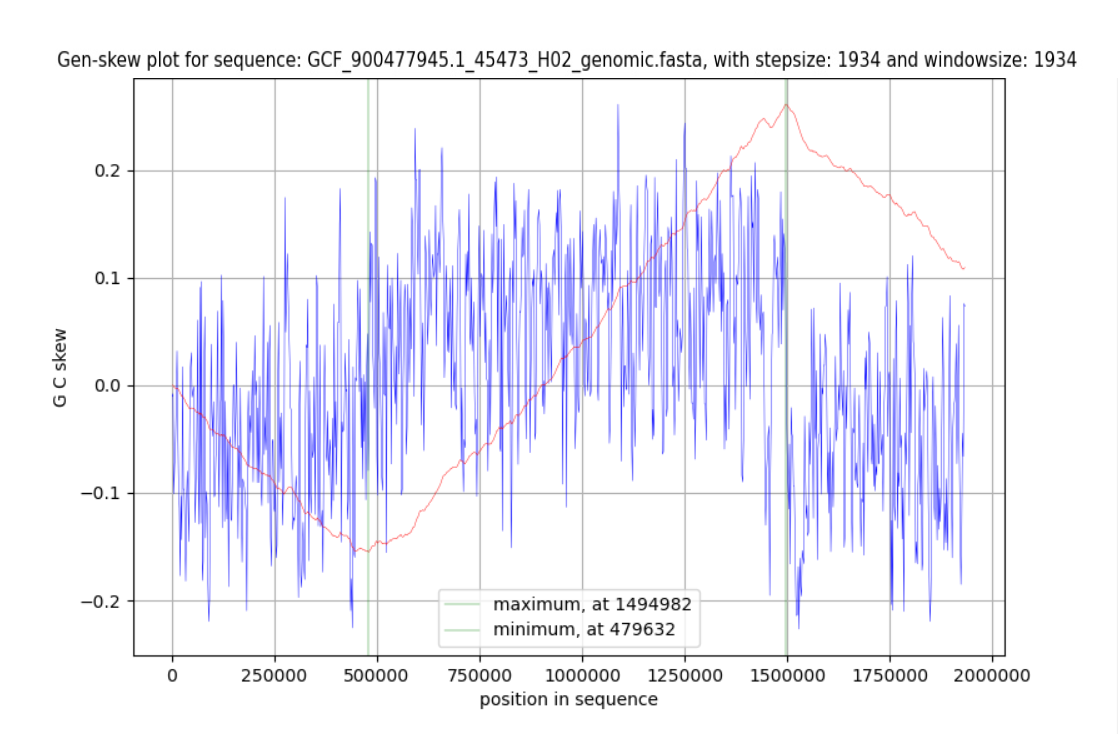
График1 отражает изменение значение GCSkew в пределах хромосомы H. Influenzae, по этому показателю, который рассчитывается по формуле: GCSkew = (G C)/(G + C), можно судить об отклонении нуклеотидного состава лидирующей и отстающей цепей, лидирующая цепь насыщена Гуанином (G), отстающая цепь –Цитозином (C). Ближе к oriC GCSkew принимает минимальное значение, в районе ter же максимальное. На графике синей линией обозначен GCSkew окрестности определенной позиции в геноме, красной показан тренд изменения этого показателя, а зеленой выделены его минимум и максимум. В данном случае координата oriC=479632(район минимума), а координата ter=1494982(район максимума).
2. Любопытная последовательность.
Была найдена последовательность, повторяющаяся в геноме H. haemolyticus 23 раза длиной 47 нуклеотидов : ‘CCTACCATTACCACCAAGGATAATGCTGATGGCTCTCATACCATTACА’.
Интересно то, что последовательность встречается все 23 раза в одной CDS, кодирующей белок, относящийся к семейству YadAподобных белков. Вероятно, эта последовательность представляет из себя консервативный белковый домен. Если транслировать ее в аминокислотную последовательность, то становится понятным, почему она состоит из 47, а не 48 нуклеотидов: последняя аминокислота (треонин) кодируется 4мя кодонами, а значит нет особой разницы в том, какой нуклеотид будет на последнем месте. Сама аминокислотная последовательность‘·P·T·I·T·T·K·D·N·A·D·G·S·H·T·I·T·’.
Для понимания общей картины я решил исследовать несколько таких повторяющихся последовательностей, и оказалось, что многие из них входят в CDS, кодирующую именно этот YadAподобный белок. Длина этой CDS= 7521 bp. Судя по информации в белковых базах данных, имеет транспортную функцию [7], но все же конкретно об этом белке известно немного. Возможно, этот белок представляет из себя огромное множество повторяющихся (т.е. одинаковых) длинных аминокислотных цепочек, что согласуется с его мембранным месторасположением.
3. Частоты использования кодонов, кодирующих одну и ту же аминокислоту или стопкодон.
Таблица1 отражает частоту вхождений разных кодонов в CDS H. Haemolyticus, наиболее часто встречающимися кодонами стали ‘AAA’ (28648 повторов) и ‘TTT’ (18467 повторов), кодирующие лизин и фенилаланин, соответственно. Наиболее часто кодируемые аминокислоты: лейцин (65376), лизин (44893), изолейцин (40724).
4. Гистограмма длин белков.
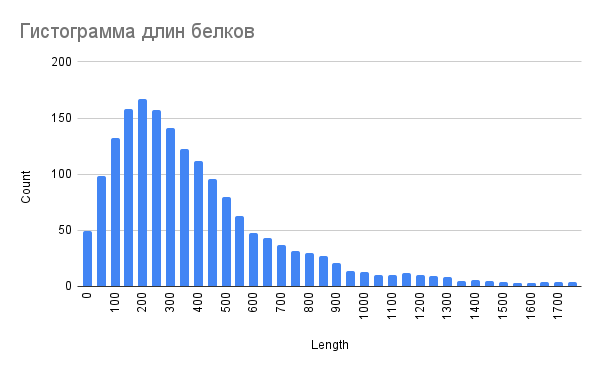
На гистограмме1 можно обнаружить два пика: белки длиной около 200 а.о. (аминокислотных остатков) и белки длиной около 1150 а.о. .
Логично интерпретировать первый пик, как длину большинства белков бактерии, но, что можно сказать насчет второго пика? Возможно, такой пик обусловлен функциональной особенностью таких белков.
По имеющимся данным (Таблица2) можно понять, что они выполняют разные функции в клетке: некоторые участвуют в репликации, некоторые находятся в мембране и участвуют в транспорте веществ и т.д.. Можно предположить, что для некоторых ферментов, мембранных белков требуются крупные субъединицы для выполнения своей ‘работы’ в клетке.
5.Частота разных стопкодонов.
Было обнаружено резкое различие в частотах встречаемости различных стопкодонов: ‘TAA’1374, ‘TAG’241, ‘TGA’ 155, помимо стандартных стопкодонов на концах последовательностей обнаружились и другие кодоны. Оказалось, что 3 из 8 таких генов являются псевдогенами (что объясняет ‘некорректность’ их последнего кодона), остальные, как мне кажется, являются либо неким браком, либо недоработкой.Заключение
Изучение свойств геномов и протеомов организмов является важной задачей для современных биологов, ведь понимая их, мы можем их изменять и, соответственно, использовать.В работе были изучены некоторые свойства генома H. haemolyticus: найдены координаты инициации и терминации репликации един-ственной хромосомы бактерии, была найдена интересная последовательность, повторяющая-ся в геноме 23 раза, изучены частоты исполь-зования разных кодонов аминокислот, стопкодонов, рассмотрены длины различных белков, обнаружены некоторые закономерности в их распределении по длинам, подсчитаны частоты различных стопкодонов, находящихся на концах последовательностей.
Сопроводительные материалы
В папке на Google drive: https://drive.google.com/drive/folders/1x0WdymERa-cLjlHstIO-TBc_WG84sv0E?usp=sharingСсылки
- Pickering, J. L., Prosser, A., Corscadden, K. J., de Gier, C., Richmond, P. C., Zhang, G., Thornton, R. B., & Kirkham, L. A. (2016). Haemophilus haemolyticus Interaction with Host Cells Is Different to Nontypeable Haemophilus influenzae and Prevents NTHi Association with Epithelial Cells. Frontiers in cellular and infection microbiology, 6, 50. https://doi.org/10.3389/fcimb.2016.00050
- Haemophilus haemolyticus_J in GBIF Secretariat (2021). GBIF Backbone Taxonomy. Checklist dataset https://doi.org/10.15468/39omei accessed via GBIF.org on 20211023.
- Fazal, M. A., Alexander, S., Grayson, N. E., DeheerGraham, A., Oliver, K., Holroyd, N., Parkhill, J., & Russell, J. E. (2019). Complete WholeGenome Sequence of Haemophilus haemolyticus NCTC 10839. Microbiology resource announcements, 8(25), e0023219. https://doi.org/10.1128/MRA.0023219
- Timothy F. Murphy, Aimee L. Brauer, Sanjay Sethi, Mogens Kilian, Xueya Cai, Alan J. Lesse, Haemophilus haemolyticus: A Human Respiratory Tract Commensal to Be Distinguished from Haemophilus influenzae, The Journal of Infectious Diseases, Volume 195, Issue 1, 1 January 2007, Pages 81–89, https://doi.org/10.1086/509824
- Slack M. (2015acquired pneumonia. Pneumonia (Nathan Qld.), 6, 26–43. https://doi.org/10.15172/pneu.2015.6/520fs
- сервис “Genskewcc” https://pypi.org/project/Genskewcc/#files)
- Uniprot, ‘Streptococcal surfaceanchored protein repeat’, https://www.uniprot.org/uniprot/A0A2X4R6L0