Практикум 8
Задание 1
Кажется, из-за того, что версий сборок геномов Mus musculus довольно много, файлы с контигами были давно удалены. Поэтому я решил взять фрагмент 3-ей хромосомы , содержащий довольно важный ген Sox2. Идентификатор хромосомы + координаты участка: NC_000069.7:34,703,000 - 34,708,000
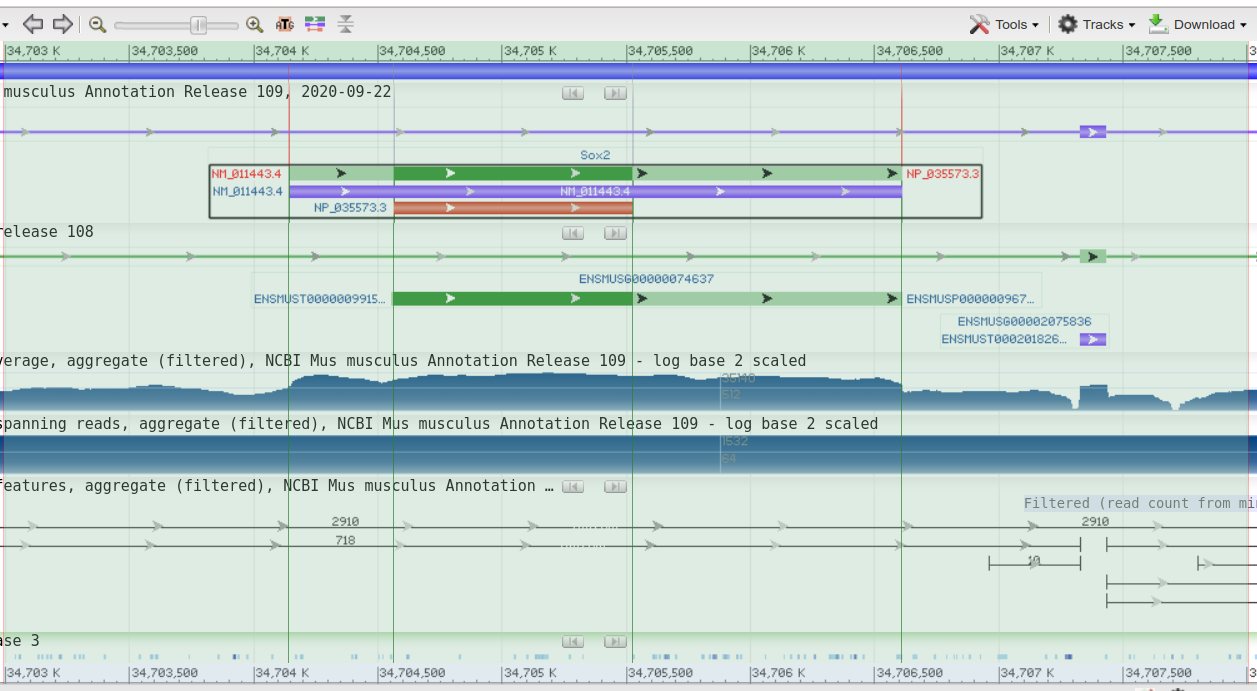
Судя по информации из геномного браузера, на этом участке целиком распологается ген, кодирующий Sox2 (fun fact: в гене лишь 1 экзон) и некоторая "белок-некодирующая" посл-ть (SOX2 overlapping transcript, довольно длинная: 578,350 bp )
Ссылка на fasta-файл с нуклеотидной посл-тью этого участкаЗапустил различные алгоритмы BLAST(blastn, megablat,tblastx, blastx), исключив находки, принадлежащие видам таксона Chordata. Параметры: word-size=3, кол-во находок=100, остальные не изменял. Далее про работу каждого алгоритма отдельно:
Blastn смог найти много интересных посл-тей: находками с самой высокой идентичностью были различные искусственные конструкции с Sox2. Что интересно, после таких конструкций были найдены различные гомологичные полс-ти из крайне разнообразных видов с довольно высоким сходством,так, например, выравнивание с пол-стью Andrena fulva(Рыжая горная пчела) имело per. identity = 82%, что позволяет предположить, что выбранный участок крайне консервативен.

Blastx также порадовал: были найдены все те же конструкции и другие посл-ти(любопытно, в выдаче не было той же пчелы , возможно, если бы я увеличил кол-во выдаваемых находок, то выравнивание с ней было бы выдано)
А вот tblastx и megablast не дали результатов: в результате поиска первого алгоритма вышла ошибка о нехватке вычислительных ресурсов для обработки данного запроса, а второй алгоритм не мог ничего найти в течение нескольких минут (что вполне логично, ведь megablast ищет среди крайне схожих полс-тей, а таксон Chordata из поля поиска я просто исключил)
Примеры использования различных алгоритмов BLAST: megablast- может, сравнение некоторых полиморфизмов мыши, которая есть "на руках", и полиморфизмов в различных сборках; blastn- поиск по нуклеотидной консерватичной полс-ти близких организмов (м.б., даже построение деревьев на основе выравниваний BLAST, хотя это звучит как-то странно); blastx- поиск белков, которые ,вероятно, гомологичны, белку, кодируемому посл-тью, подающейся на вход алгоритму (при этом посл-ть еще не была аннотирована) ; tblastx- поиск похожей непредсказанной белковой посл-ти.
Задание 2
Индексирование сделал локально (все же геном большой, на kodomo бы не поместился), команда: makeblastdb -in GCF_000001635.27_GRCm39_genomic.fna -dbtype nucl. После ее выполнения было создано несколько файлов в форматах: .ndb, .nin, .nsq, .nto , .nhr, .not, .ntf
Выданы посл-ти 16S и 23S рРНК, первая участвует в сборке малой субъединицы рибосомы + узнавании мРНК, вторая же имеет только структурную ф-цию (входит в состав среднего протуберанца большой субъединицы)
Выравнивание проведено с помощью команды: blastn -task blastn -query rrna.txt -db GCF_000001635.27_GRCm39_genomic.fna > ans.txt , параметры стандартные (word_size=11, etc). Пробовал запустить с word_size=4 для 16S рРНК, к сожалению, считалось слишком долго(. Blastn для разных посл-тей запускал отдельно.
Для первой посл-ти (16S_rRNA) была найдена лишь одна посл-ть с удовлетворительным E-value(9e-05), интересно, что она кодирует некую "18s RNA, related sequence 5" (конкретный участок, с которым произошло выравнивание, NC_000083.7:40,159,047-40,159,089).
Со второй же рРНК(23S_rRNA) ситуация менее тривиальная: найдено 9 посл-тей с E-value в диапозоне [6e-04, 2e-15]. Так, например, лучшее выравнивание пришлось на участок, который кодирует аминоацил-тРНК синтетазу, работающую в митохондриях.
P.S. blastn с word_size=4 завершил работу после 30 минут упорного труда моего CPU (по мнению ps aux, он был загружен на 99.9%!!!), но алгоритм нашел лишь 2 адекватные находки (1-ая - та, которую я описал, вторая - случайный даже неаннотированный участок X хромосомы). Поэтому я даже и не стал запускать blastn с таким же параметром для 23 S рРНК(т.к. долго и бесполезно).