Практикум 4
Задание 1
Последовательность белка CLPX Escherichia coli была выровнена со всеми белками протеомов выбранных мной бактерий. Находки, прошедшие порог в E-value = 1e-3 представлены ниже:
| Мнемоника | E-value |
| Q6NFB1_CORDI | 7.07e-05 |
| CLPX_LEIXX | 2.07e-180 |
| Q6ACQ0_LEIXX | 4.06e-04 |
| CLPX_ARTS2 | 0.0 |
| A0K1M3_ARTS2 | 1.19e-07 |
| RUVB_ARTS2 | 5.83e-04 |
| CLPX_BIFLO | 1.86e-149 |
| Q8G871_BIFLO | 1.43e-06 |
| RUVB_BIFLO | 3.60e-04 |
| Q8G3S2_BIFLO | 4.07e-04 |
| CLPX_MYCLE | 0.0 |
| CLPX_CLAMS | 3.17e-173 |
| CLPX_COREF | 0.0 |
| Q8FMH5_COREF | 2.38e-05 |
Задание 2
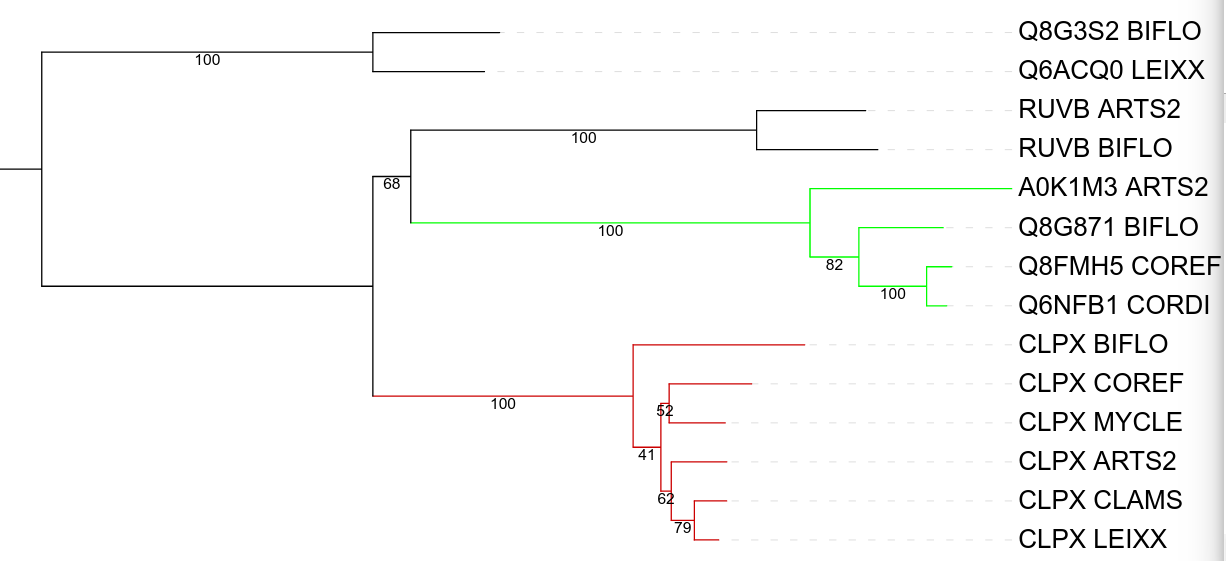
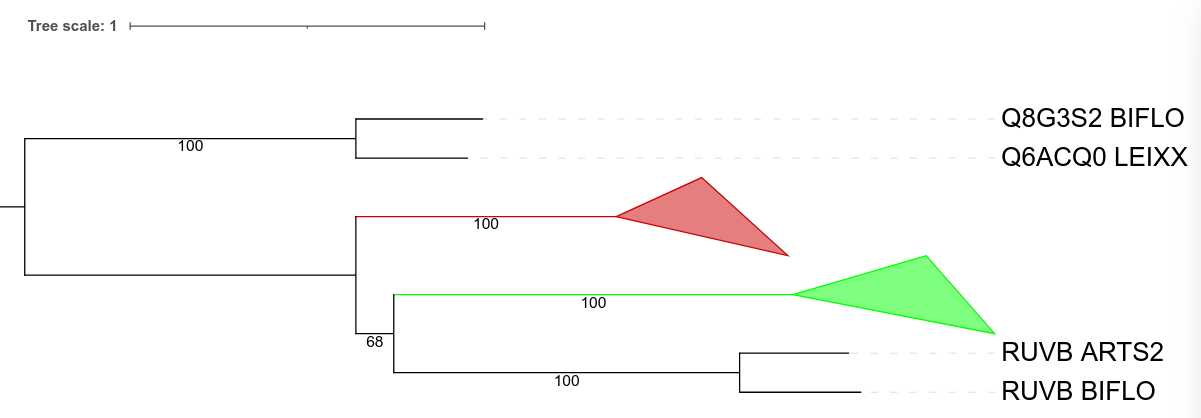
C помощью алгоритма FastMe построил дерево: отображение в newick формате. На дереве видны следующие пары предполагаемых ортологов: CLPX_CLAMS и CLPX_LEIXX; CPLX_MYCLE и CLPX_COREF; RUVB_ARTS2 и RUVB_BIFLO, и пары вероятных паралогов: CLPX_BIFLO и Q8G871_BIFLO; A0K1M3_ARTS2 И RUVB_ARTS2; RUVB_BIFLO и Q8G871_BIFLO.
Выводы
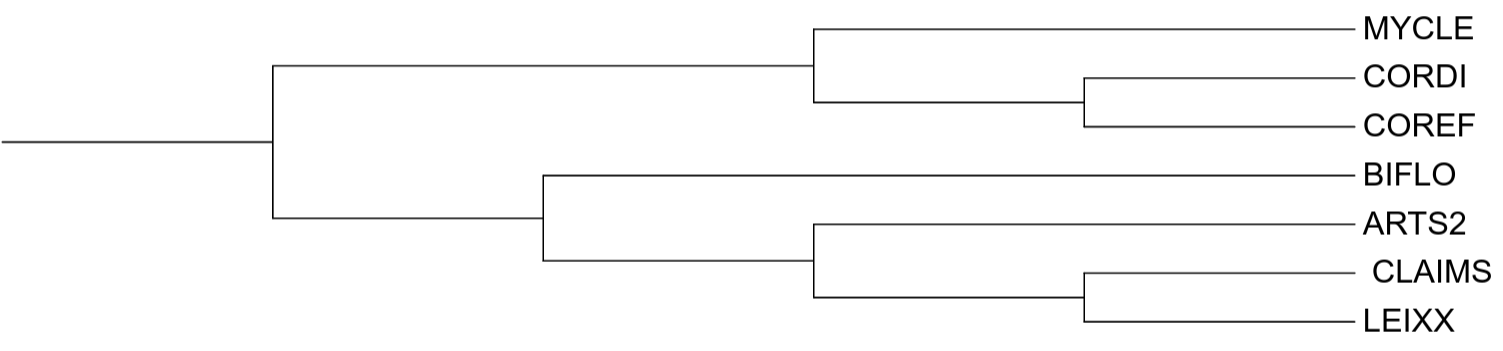
Интересно то, что топологическая структура 2-x ортологических групп довольно сильно похожа на филогенетическое дерево выбранных бактерий. Правда, видно, что расположение листа c BIFLO в красной ортологической группе отличается от его расположения на филогенетическом дереве, отмечу, что ветвь, отделяющая белок этого организма, есть лишь в 41 из 100 деревьев ("неподдержанная ветвь"), топология же зеленой группы согласуется полностью.
Все белки, входящие в состав красной группы, имеют мнемонику ClpX, участвуют в ATP-зависимой деградации белков. Белки зеленой группы же имеют разные мнемоники, но обладают одной общей активностью- ATP-связывающей, остальные же различаются: Q6NFB1- шаперон, а A0K1M3 им не является.