A-, В- и Z- формы ДНК. Структура РНK
Целью данного практикума было сравнить строение разных форм ДНК, и ДНК с РНК. А также научиться работать
с пакетом 3DNA. Пакет 3DNA - один из популярных пакетов программ для анализа и простейшего моделирования
структур нуклеиновых кислот, который работает под операционной системой LINUX.
Задание 1.
В этом задании мы учились использовать пакет 3DNA для получения пространственных структур ДНК. Ниже представленны ссылки на
полученные файлы в формате pdb. Цепи Z-формы состоят из последовательности GCGCGCGC. Благодаря этому она имеет такую форму.
A-форма
B-форма
Z-форма
Задание 2.
В данном задании необходимо было выбрать одну форму ДНК и определить, где малые и большие борозки, для того, чтобы у какого-то выбранного
основания раскрасить атомы. Красные смотрят в сторону большой борозки, а синие в сторону малой. Была выбрана A-форма.
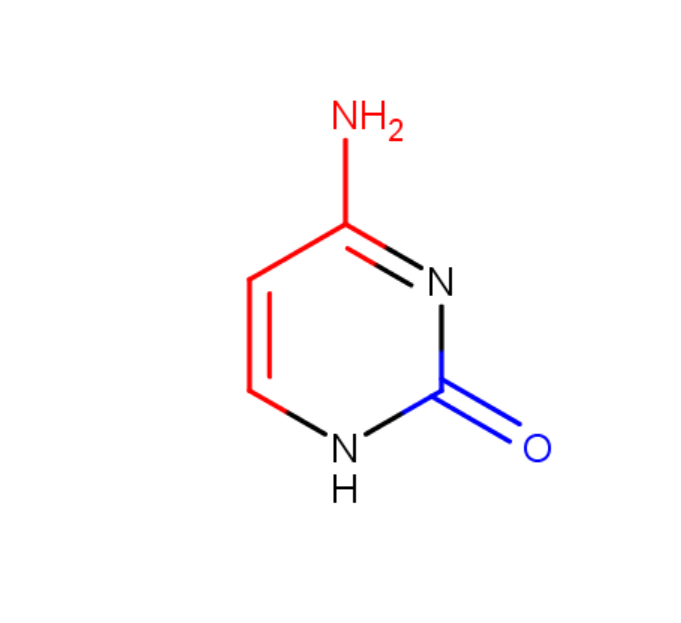
Рисунок 1. Цитозин
|
В сторону большой бороздки смотрят атомы: DC28: C6, C5, C4, N4.
|
В сторону малой бороздки смотрят атомы: DC28: C2, O2.
|
Неопределенного направления: DC28: N1, N3.
|
Таблица 1. Сравнение некоторых параметров разных форм ДНК
|
A-форма |
B-форма |
Z-форма |
| Тип спирали (правая или левая) |
Правая |
Правая |
Левая |
| Шаг спирали (A) |
28.03 |
33.75 |
43.50 |
| Число оснований на виток |
11 |
10 |
12 |
| Ширина большой бороздки (A) |
7.98 [DA]26:B.P #515 - [DG]9:A.P #165 |
17.21 [DG]29:B.P #575 - [DG]9:A.P #165 |
9.86 [DC]16:A.P #310 - [DG]29:B.P #575 |
| Ширина малой бороздки (A) |
16.81 [DG]9:A.P #165 - [DA]34:B.P #679 |
11.69 [DT]15:A.P #290 - [DA]30:B.P #597 |
18.30 [DC]30:B.P #597 - [DC]8:A.P #146 |
Задание 3.
Упражнение 1.
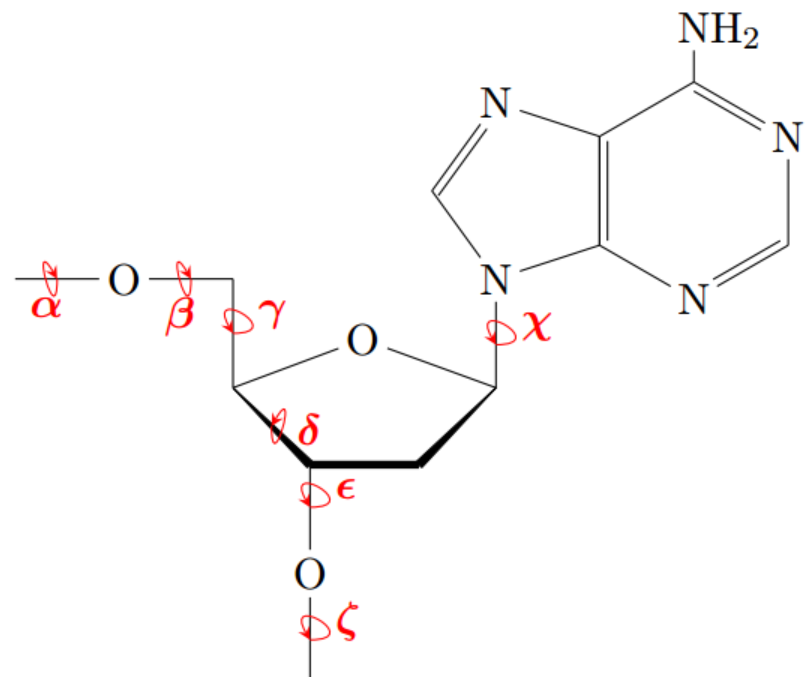
Таблица 2. Сравнение торсионных углов в градусах
|
|
Alpha |
Beta |
Gamma |
Delta |
Epsilon |
Zeta |
Chi |
| A-форма
|
-51.7 |
174.8 |
41.7 |
79.1 |
-147.8 |
-75.1 |
-157.2 |
| B-форма
|
-29.9 |
136.3 |
31.2 |
143.3 |
-140.8 |
-160.5 |
-98.0 |
| тРНК |
-30,13 |
35,25 |
39,83 |
85,46 |
-131,987 |
-52,33 |
-123,71 |
Трудно сравнивать значения столь различные, но кажется, что тРНК больше хороша на A-форму ДНК.
Близки по значениям углы: гамма, дельта, дзета и кси. (Gamma, Delta, Zeta, Chi)
Упражнение 2. Структура РНК.
Координаты стеблей в тРНК можно найти в файле XXXX.out, где XXXX - это PDB ID молекулы. Информация о структуре
была получена с помощью команды:
find_pair -t XXXX.pdb stdout | analyze
Исследовалась структура
1FFY и было обнаружено
4 стебля.
Координаты стеблей (указаны номера нуклеотидов)
| Strand 1 |
Strand 2 |
| 1 - 7 |
66 - 72 |
| 49 - 53 |
61 - 65 |
| 38 - 44 |
26 - 32 |
| 10 - 13 |
22 - 25 |
Неканонические пары оснований перечислены ниже. В файле XXXX.out
указано, что в структуре имеется 10 не Уотсон-Криковских взаимодействий.
Не всегда это очевидно из-за записей в файле, потому как взаимодействовать могут привычные
коплиментарные пары, но с образованием других водородных связей. Они выделены жирным шрифтом.
5_:[..U]U-*---G[..G]:..68
49_:[..G]G-*---U[..U]:..65
54_:[..U]U-**--A[..A]:..58
55_:[..U]U-**+-G[..G]:..18
36_:[..U]U-**+-U[..U]:..33
38_:[..A]A-**--C[..C]:..32
44_:[..A]A-**--G[..G]:..26
14_:[..A]A-**--U[..U]:...8
15_:[..G]G-**+-C[..C]:..48
16_:[..G]G-**--U[..U]:..17
Информацию о дополнительных водородных связях для стабилизации структуры тРНК можно найти все в том же
файле XXXX.out. Это те связи, которые не вошли в состав стеблей и характерны для обеих цепей.
(0.005) ....>T:..54_:[..U]U-**--A[..A]:..58_:T<.... (0.007)
(0.008) ....>T:..55_:[..U]U-**+-G[..G]:..18_:T<.... (0.011)
(0.006) ....>T:..15_:[..G]G-**+-C[..C]:..48_:T<.... (0.008)
(0.005) ....>T:..16_:[..G]G-**--U[..U]:..17_:T<.... (0.003)
(0.008) ....>T:..19_:[..G]G-----C[..C]:..56_:T<.... (0.004)
(0.011) ....>T:..36_:[..U]U-**+-U[..U]:..33_:T<.... (0.005)
© Нестеренко Екатерина 2018


