Практикум 11. Ресеквенирование. Поиск полиморфизмов у человека.
| № задания | задание | команда |
Часть I: подготовка чтений | ||
| 1. Анализ качества чтений | контроль качества чтений с помощью программы FastQC | fastqs chr20.fastq |
| 2. Очистка чтений | очистка чтений с помощью программы Trimmomatic. Отрезала с конца каждого чтения нуклеотиды с качеством ниже 20, оставила только чтения длиной не меньше 50 нуклеотидов | java -jar /usr/share/java/trimmomatic.jar SE -phred33 chr20.fastq ex2.fastq TRAILING:20 MINLEN:50 |
Часть II: картирование чтений | ||
| 3 картирование чтений | экспорт Hisat | Hisat2 export PATH=${PATH}:/home/students/y06/anastaisha_w/hisat2-2.0.5 |
| проиндексировала референсную последовательность | hisat2-build chr20.fastq>ex3 | |
| построила выравнивание прочтений и референса в формате .sam. | hisat2 -x /nfs/srv/databases/ngs/chashnikova98/ex3 -U /nfs/srv/databases/ngs/chashnikova98/ex2.fastq --no-softclip --no-spliced-alignment >chr20.sam | |
| 4.Анализ выравнивания | Перевела выравнивание чтений с референсом в бинарный формат .bam | samtools view -b chr20.sam>chr20.bam |
| Отсортировала выравнивание чтений с референсом (получившийся после картирования .bam файл) по координате в референсе начала чтения | samtools sort chr20.sam>chr20sort | |
| Проиндексировала отсортированный .bam файл командой samtools index | samtools index chr20.bam | |
Часть III: Анализ SNP | ||
| 5. Поиск SNP и инделей. | Создала файл с полиморфизмами в формате .bcf | samtools mpileup -uf chr20.fasta chr20_sorted.bam -o chr20_snp.bcf |
| Создала файл со списком отличий между референсом и чтениями в формате .vcf | bcftools call -cv snp.bcf -o snp.vcf | |
| Создала файл для работы annovar | ./convert2annovar.pl -format vcf4 polymorf.vcf -outfile polymorf.avinput | |
| 6. Аннотация SNP. | С помощью программы annovar проаннотировала полученные snp: | |
| использовала базу данных refgene | perl /nfs/srv/databases/annovar/annotate_variation.pl -out rs.refgene -build hg19 polymorf.avinput /nfs/srv/databases/annovar/humandb/ | |
| dbsnp | perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -out rs.snp -build hg19 -dbtype snp138 polymorf.avinput /nfs/srv/databases/annovar/humandb/ | |
| 1000 genomes | perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -out rs.1000g -buildver hg19 -dbtype 1000g2014oct_all polymorf.avinput /nfs/srv/databases/annovar/humandb/ | |
| Gwas | perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -out rs.gwas -build hg19 -dbtype gwasCatalog polymorf.avinput /nfs/srv/databases/annovar/humandb/ | |
| Clinvar | perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -out rs.clinvar -dbtype clinvar_20150629 -buildver hg19 polymorf.avinput /nfs/srv/databases/annovar/humandb/ | |
Часть1. Очистка чтений
| качество прочтений до очистки | качество прочтений после очистки |
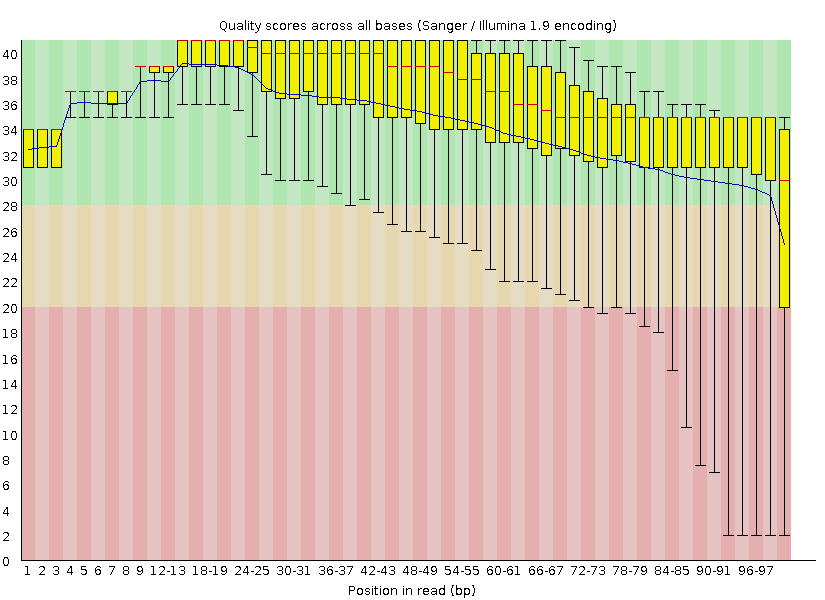 | 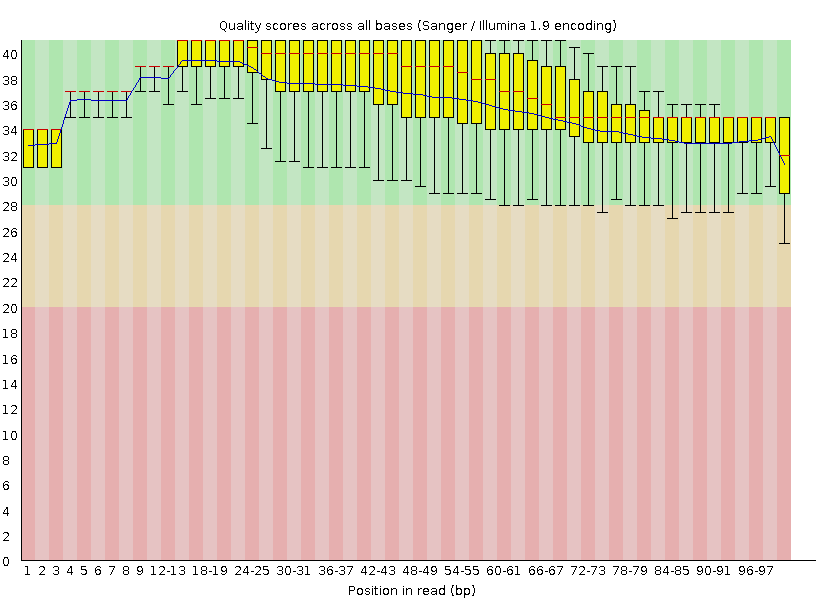 |
| количество чтений до очистки | количество чтений после очистки |
| 4661 | 4472 |
После очистки были исключены короткие чтения(длиной до 50 пар оснований), а также концы чтений, имеющие низкое качество. Таким образом мы повысили достоверность чтений, избежав допуск вероятных ошибок:
- короткие риды - чтение захватывает последовательности адаптеров (пропск последовательности днк между ними)
- ошибки в концах чтений за счет неправильной работы полимеразы
Часть2. Картирование чтений
Рис1. Вывод hisat2 --no-spliced-alignment --no-softclip -x chr20ind -U ex2.fastq -S align.sam

Как мы видим 17 чтений не были выровнены вообще, а 4455 выровнены 1 раз.
Часть III: Анализ SNP
Всего найдено 41 полиморфизм, все - точечные однонуклеотидные замены.
Таблица 2. Примеры полиморфизмов.
| хромосома | тип полиморфизма | координата | в референсе | в ридах | качество покрытия | покрытие |
| chr20 | 33903158 | замена | G | A | 3.01394 | 1 |
| chr20 | 33913681 | замена | A | C | 6.20226 | 1 |
| chr20 | 33918910 | замена | C | T | 73.0074 | 15 |
Таблица 3. Примеры полиморфизмов, аннотированных в разных базах данных.
| Координата | SNP | Quality и DP | refgene | dbsnp | 1000 genomes | Gwas | Clinvar |
| 34025983 | замена A на G | 176.016 | upstream hom | rs143383 | 0.3718 | - | остеоартрит |
| 33913681 | замена A на C | 6.20226 | intronic hom | rs6060372 | 0.326 | - | - |
| 48522330 | замена G на A | 196.009 | exonic het | rs495337 | 0.3976 | псориаз | - |
Всего rs есть у 30 полиморфизмов (нет у 11, по базе данных dbsnp). По базе данных 1000 геномов аннотированно 29, частота встречаемости от 0,1% до 98,61% (медиана 35,39%). GWAS - всего аннотировано 3 полиморфизма, связанных с весом, псориазом и атрофией гиппокампа. В Clinvar аннотирован 1 полиморфизм, связанный с остеоартритом. В RefSeq полиморфизмы подразделяются на exonic (внутри гена) , intergenic (пересечение генов), UTR5 (5'-нетранслируемая область) , UTR3 (3'-нетранслируемая область), intronic (интрон), upstream (у сайта начала транскрипции), downstream (у сайта окончания трансрипции), intergenic (пересечение генов).
Refgene: интронные - 29, внутри экзона - 8, 3 - на пересечении генов, upstream - 2, UTR5 -1. Как можно заметить больше всего полиморфизмов найдено в интронах, что логично, т.к. во-первых интронов в геноме намного больше, а во-вторых, мутации в интронах не так критичны для организма как мутации в экзонах. Также были найдены полиморфизмы на пересечении генов, upstream и UTR5, но их было значительно меньше.