Задание 1. Анализ качества чтений.
В качестве данных использовала чтения, картирующиеся на участок хромосомы человека (получены путем секвенирования РНК). Взяла файлы с одноконцевыми чтениями в формате fastq 20 хромосомы (выбрала первую реплику).
Также использовала разметку человеческого генома по версии Gencode19 для сборки hg19.
С помощью команды fastqc было оценено качество ридов. Т.к. оно оказалось достаточно хорошим, очистка не требовалась.
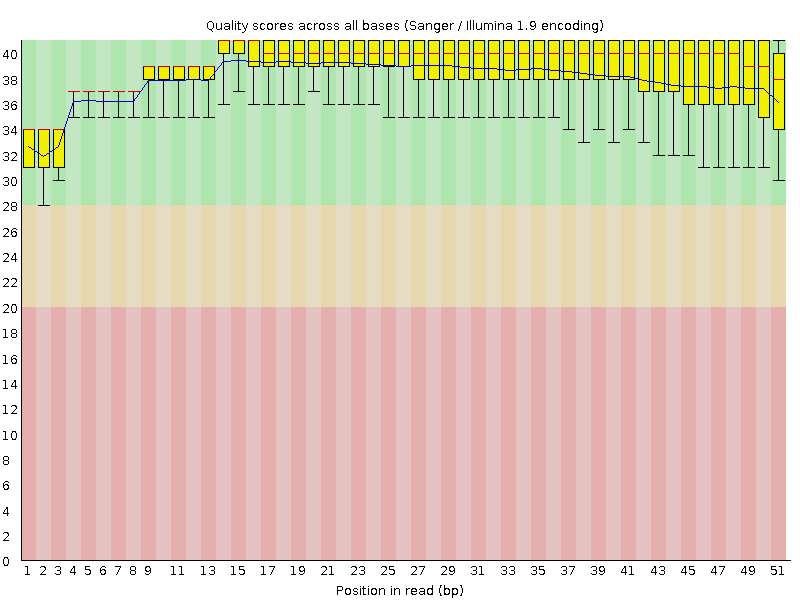 Всего ридов - 3565, длина 41-51, %GC - 55%.
Всего ридов - 3565, длина 41-51, %GC - 55%.
Задание 2. Картирование чтений .
| функция | команда |
| Индексирование референсной последовательности | export PATH=${PATH}:/home/students/y06/anastaisha_w/hisat2-2.0.5
hisat2-build chr20.fasta chr20
|
| Выравнивание чтений с референсной последовательностью | hisat2 -x chr20 -U chr20.1.fastq --no-softclip -S align.sam |
| Перевод файла в формат bam | samtools view align.sam -b -o align.bam |
| Сортировка выравнивания чтений и референса по координате референса | samtools sort align.bam -T file.txt -o sort.bam |
| Индексирование выравнивания чтений и референса | samtools index sort.bam |
Из команды 'hisat2 ...' была убрана опция '--no-spliced-alignment'.
Задание 3. Анализ выравнивания.
3507 чтения были выровнены 1 раз, 47 - 0 раз, 11 больше 1 раза.
Задание 4. Подсчет чтений.
Для выполнения данного задания использовался пакет Bedtools.
| функция | команда |
| bam файл > bed | /P/y14/term3/block4/SNP/bedtools2/bin/bedtools bamtobed -i sort.bam > chr20.bed |
| файл, с покрытиями ридов и генами, попавшими в покрытия | /P/y14/term3/block4/SNP/bedtools2/bin/bedtools intersect -a /P/y14/term3/block4/SNP/rnaseq_reads/gencode.genes.bed -b chr20.bed -c > ex4.bed |
| сортировка | sort -k 6 -r ex4.bed > ex42.bed |
| удаление нулевых покрытий | /P/y14/term3/block4/SNP/bedtools2/bin/bedtools intersect -a
/P/y14/term3/block4/SNP/rnaseq_reads/gencode.genes.bed -b ex42.bed -u > last.bed |
Задание 5. Анализ резльтатов.
Было найдено 3 гена:
| координаты | длина | направление цепи | описание | ген | число ридов, в которых встречается |
| 2447858 - 2447961 | 103 | - | protein_coding | RP4-734P14.4 | 8 |
| 2443608 - 2443683 | 75 | - | snoRNA | SNORD119 | 3 |
| 2442736 - 2443264 | 529 | - | protein_coding | SNRPB | 79 |
SNRPB - белок, закодированный этим геном, является одним из нескольких ядерных белков, которые встречаются среди малых частиц рибонуклеопротеина (snRNP) U1, U2, U4/U6.
Эти snRNPs участвуют в пре-мРНК сплайсинге.
Автоантитела у больных системной красной волчанкой часто распознают эпитопы на кодируемый белок. Для данного гена характерны два варианта транскриптов кодирующих различные изоформы (B и B').
Задание 6. Дополнительные задачи по bedtools.
| функция | команда |
| Получите из файла в выравниванием файл с чтениями в формате fastq | bedtools bamtofastq -i last.bam -fq last.fq |
| Получите файл с нуклеотидной последовательностью (.fasta) для одного из покрытых Вашими чтениями генов. | bedtools getfasta -fi chr20.fasta -bed last.bed > ex2chr20.fasta |
| Наберите из Вашей хромосомы 1000 случайных фрагментов по 200 нуклеотидов. | randomBed -g chr20.fasta -l 200 -n 1000 > chr20random.bed |
| |
© Чашникова Анастасия, 2016
|
 Всего ридов - 3565, длина 41-51, %GC - 55%.
Всего ридов - 3565, длина 41-51, %GC - 55%.