|
Задание 1. Анализ последовательности ДНК на основании данных, полученных из капиллярного секвенатора по Сэнгеру.
Мне были даны две последовательности ДНК(прямая и обратная), полученные с помощью секвенирования по Сенгеру.
Ссылка на прямую последовательность в ab1 формате
Ссылка на обратную последовательность в ab1 формате
Для обработки данных я использовала программу Chromas. Для обратной последовательности я открыла другое окно и использовала команду Reverse+Complement.
Сначала я обрезала 3' и 5'-концы, т.к. они нечитаемы из-за высокого уровня шума и смазанных низких пиков (читаемы - 50-672 нуклеотиды для прямой и 41-683 для обратной.
Далее после исправления ошибок Chromas (пропуск нуклеотидов, неспособность опознать нуклеотид(см. в примерах ниже)), были экспортированы
fasta-последовательности и выровнены с помощью программы needle пакета emboss.
| Gap_penalty | 10.0 |
| Extend_penalty | 0.5 |
| Length | 661 |
| Identity | 589/661 (89.1%) |
| Similarity | 595/661 (90.0%) |
| Gaps | 63/661 ( 9.5%) |
| Score | 2923.0 |
Рис1. Выравнивание прямой и обратной последовательности ДНК.
В целом хромотограмма очень хорошая, уровень шума низкий (не превышает примерно 10-20% сигнала) и пики сигнала высокие и четкие, коэффицент вероятности ошибки Q =15-25 для большинства нуклеотидов.
После выравнивания я еще раз просмотрела несовпадающие участки и повторно исправила ошибки, но уже намного более точно, т.к. использовала сразу 2 хроматограммы.
Очень часто проблемные участки на 1 хроматограмме можно четко определить на обратной.
Последовательность ДНК в fasta-формате, после окончательного редактирования.
Примеры исправлений.
Очень часто непонятные участки в выравнивании можно восстановить по обратной последовательности.
| пример1 | пример2 | пример3 |
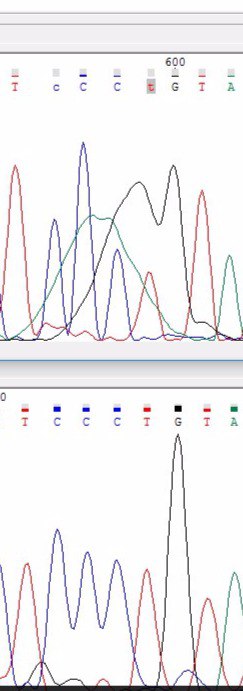 |  |  |
| программа не распознала 599 T(N) из-за пятна краски гуанина |
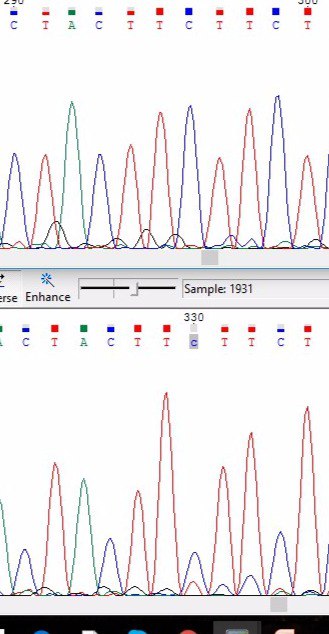
программа не распознала 330 С(N) из-за того, что пик был очень низкий и почти слился с шумом. (встречаются почти такие же по высоте пики, но там уровень шума почти нулевой) |
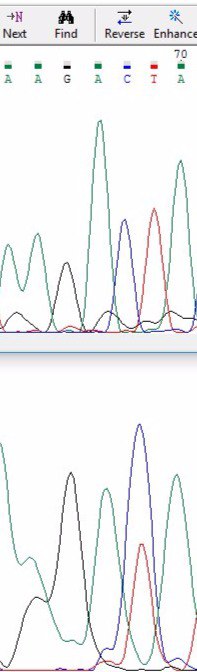
пример 3 взят из самого начала хроматограммы и выбран из-за того, что очень похож на полиморфизм.
Видно два пика друг над другом: нормальный цитозин и шум- тимин. Однако это просто ошибка, т.к.
на обратной последовательности мы должны бы были видеть обратную картину: два пика - нормальный тимин и шуи- цитозин.
Как можно заметить, это не так. Причины ошибки - возможно сдвиг рамки считывания и пр. |
| пример 4 |
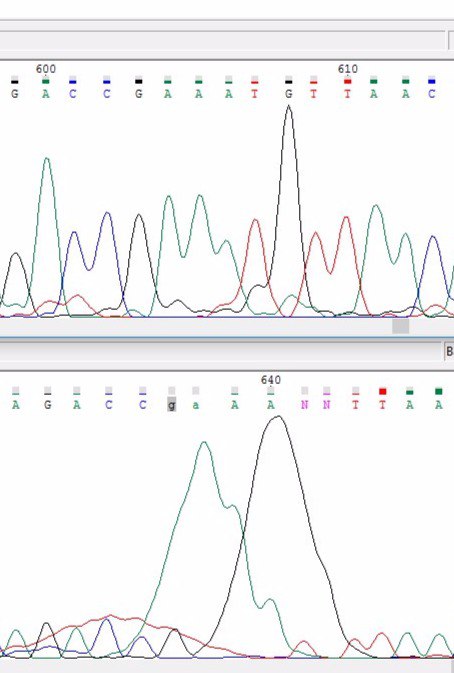 |
Внизу практически нечитаемый фрагмент хроматограммы.
Программа не распознала 637(N) G, 638(пропуск) A, 641(N) T и 642(N) G нуклеотиды.
Зеленый высокий размытый пик аденина обусловлен тем, что не всегда получаются хорошие пики при
нескольких идущих подряд одинаковых нуклеотидах. Chromas может пропускать нуклеотиды (638 А).
Также здесь виден высокий уровень шума (выше пиков основного сигнала)
и нечеткий пик гуанина. |
Задание 2. Пример нечитаемого фрагмента хроматограммы
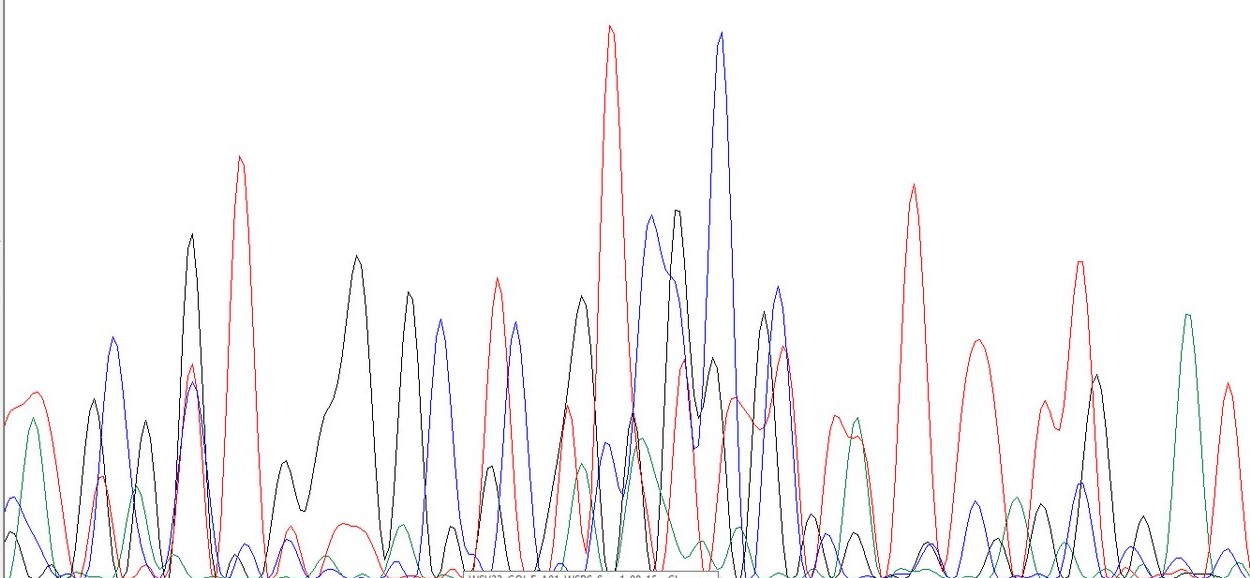
В качестве примера приведена хроматограмма WSV23_COI_F_AO1_WSBS-Seq-1-08-15, взятая из папки с "плохими" хроматограммами. Здесь мы видим:
- наложенные друг на друга пики (в начале хроматограммы). Для читаемых хроматограмм может означать полиморфизмы в гене, когда
прямая и обратная последовательность отличаются нуклеотидом или несколькими нуклеотидами.
- высокий уровень шума, на многих участках неотличимый от сигнала
- пики пологие, растянутые (зубцы могут указывать на несколько идущих подряд одинаковых нуклеотидов, здесь их не видно)
- неодинаковая ширина и высота пиков и расстояние между пиками
Скорее всего ДНК очень сильно загрязнена (солями или другими ДНК).
© Чашникова Анастасия, 2016
|





