Отобранные бактерии.
| Rhizobium etli | RHIEC |
| Rhodobacter sphaeroides | RHOS4 |
| Burkholderia cenocepacia | BURCA |
| Ralstonia solanacearum | RALSO |
| Escherichia coli | ECOLI |
| Salmonella typhimurium | SALTY |
| Yersinia pseudotuberculosis | YERPS |
| Haemophilus influenzae | HAEIN |
Cкобочная формула дерева.
((RHIEC, RHOSY),((RALSO, BURCA),(HAEIN, (YERPS, (ECOLI,SALTY)))))
Филогенетическое дерево.
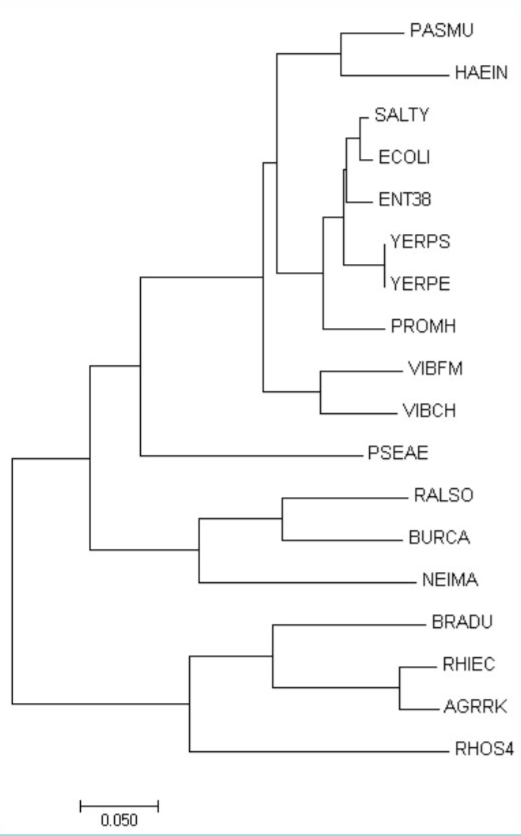
По приведенному в задании дереву было построено новое, включающее только отобранные бактерии.
| Новое дерево | Исходное дерево |
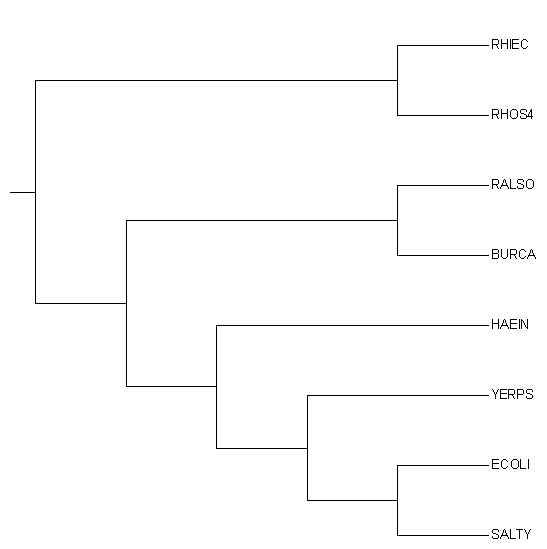 |  |
Нетривиальные ветви.
Мы называем ветвь нетривиальной, если она разбивает множество листьев на подмножества, в каждом из которых более одного элемента.
Данное дерево содержит 5 нетривиальных ветвей:
- (RHIEC, RHOS4) против (RALSO, BURCA, HAEIN, YERPS, ECOLI, SALTY)
- (RALSO, BURCA) против (RHIEC, RHOS4, HAEIN, YERPS, ECOLI, SALTY)
- (ECOLI, SALTY) против (RHIEC, RHOS4, RALSO, BURCA, HAEIN, YERPS)
- (ECOLI, SALTY, YERPS) против (RHIEC, RHOS4, RALSO, BURCA, HAEIN)
- (ECOLI, SALTY, YERPS, HAEIN) против (RHIEC, RHOS4, RALSO, BURCA)
| Практикум 2. Реконструкция филогении.
Таксономия отобранных организмов
| Rhizobium etli | RHIEC | Bacteria; Proteobacteria; Alphaproteobacteria; Rhizobiales; Rhizobiaceae; Rhizobium/Agrobacterium group; Rhizobium |
| Rhodobacter sphaeroides | RHOS4 | Bacteria; Proteobacteria; Alphaproteobacteria; Rhodobacterales; Rhodobacteraceae; Rhodobacter |
| Burkholderia cenocepacia | BURCA | Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Burkholderia; Burkholderia cepacia complex |
| Ralstonia solanacearum | RALSO | Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Ralstonia |
| Escherichia coli | ECOLI | Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacterales; Enterobacteriaceae; Escherichia |
| Salmonella typhimurium | SALTY | Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacterales; Enterobacteriaceae; Salmonella; Salmonella enterica; Salmonella enterica subsp. enterica |
| Yersinia pseudotuberculosis | YERPS | Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacterales; Yersiniaceae; Yersinia; Yersinia pseudotuberculosis complex |
| Haemophilus influenzae | HAEIN | Bacteria; Proteobacteria; Gammaproteobacteria; Pasteurellales; Pasteurellaceae; Haemophilus |
Сравнение филогенетического дерева и таксономии.
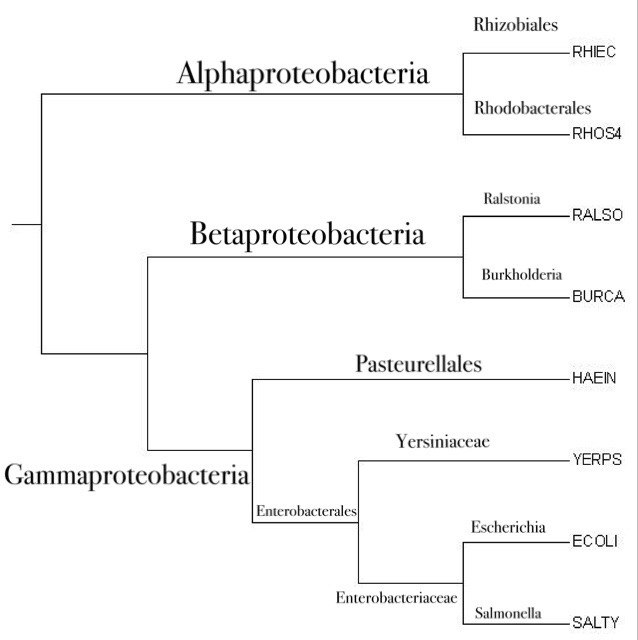
Легко заметить, что все организмы относятся к Bacteria и Proteobacteria. Далее приведены нетривиальные ветви, соответствующие определенным таксонам.
| Ветвь | Соответствующий таксон |
| (RHIEC, RHOS4) против (RALSO, BURCA, HAEIN, YERPS, ECOLI, SALTY) | Alphaproteobacteriа |
| (RALSO, BURCA) против (RHIEC, RHOS4, HAEIN, YERPS, ECOLI, SALTY) | Betaproteobacteria; Burkholderiales; Burkholderiaceae |
| (ECOLI, SALTY) против (RHIEC, RHOS4, RALSO, BURCA, HAEIN, YERPS) | Enterobacteriaceae |
| (ECOLI, SALTY, YERPS) против (RHIEC, RHOS4, RALSO, BURCA, HAEIN) | Enterobacterales |
| (ECOLI, SALTY, YERPS, HAEIN) против (RHIEC, RHOS4, RALSO, BURCA) | Gammaproteobacteria |
Задание 4. Построение и анализ дерева, содержащего паралоги.
Для белков, перечисленных выше нашла достоверных гомологов белка CLPX_ECOLI. Чтобы найти гомологов в заданных организмах,
воспользовалась файлами, лежащими на kodomo в директории /P/y16/term4/Proteomes (они содержат скачанные из Uniprot полные протеомы бактерий,
перечисленных выше). Провела поиск программой blastp гомологов (с порогом на E-value 0,001) по протеомам отобранных бактерий.
Использованные команды:
- cat file1 >> file2 - записала файлы с протеомами бактерий в 1
- makeblastdb -in file1.fasta -dbtype prot -out database.fasta - создание базы данных
- blastp -query СLPX_ECOLI.fasta -evalue 0.001 -db database.fasta -outfmt 6 -out proteins.fasta - получение списка найденных гомологов
- muscle -in 4ex.fasta -out 4exalign.fasta - выровняла последовательности гомологичных белков
Далее для построения дерева использовала мегу, применяя метод максимального сходства.
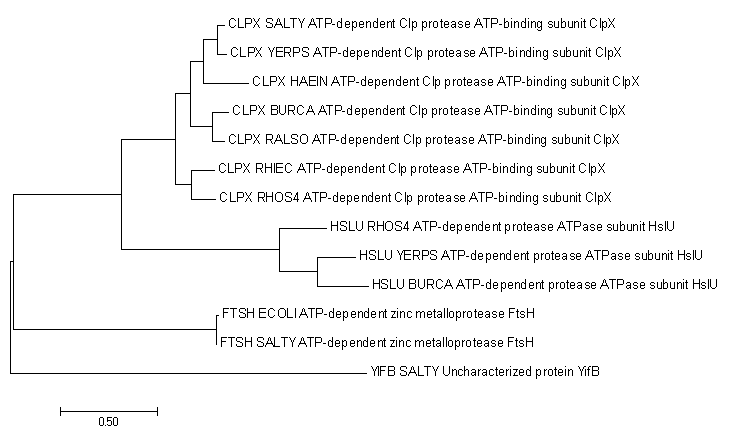
На данном дереве отражены гомологи CLPX_ECOLI во всех отобранных бактериях:
| организм | гомолог |
| SALTY | CLPX, FITSH, YIFB (скорее всего ген дуплицировался два раза, по отношению друг к другу - парологи) |
| YEPRS | CLPX, HSLU (дупликация, парологи) |
| HAEIN | CLPX |
| BURCA | CLPX, HSLU (дупликация, парологи) |
| RALSO | CLPX |
| RHIEC | CLPX |
| RHOS4 | CLPX, HSLU (дупликация, парологи) |
Cтоит отметить, что филогенетическое положение организмов не изменилось (по сравнению с двумя другими деревьями полченными ранее).
Два гомологичных белка будем называть ортологами, если они: а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования.
Два гомологичных белка из одного организма будем называть паралогами. Примеры ортологов:
- CLPX - для всех отобранных бактерий
- FITSH - SALTY, ECOLI
- HSLU - RHOS4, BURCA, YEPRS
|
|


