Базы последовательностей белков
В данном практикуме произведен анализ записей белка аспартатаминотрансферазы (идентификатор в базе Refseq NP_214350.1) из организма бактерии Aquifex aeolicus VF5 в различных базах данных.
Задание 1. Поиск идентификаторов записей белка аспартатаминотраснферазы в базах данных Uniprot, Uniref50, RefSeq Proteins, PDB.
В Таблице 1 приведены идентификаторы записей белка аспартатаминотрансферазы в базах данных Uniprot, Uniref50, RefSeq Proteins, PDB.
Таблица 1. Идентификаторы записей белка NP_214350.1 (аспартатаминотрансферазы) Aquifex aeolicus VF5 в разных базах данных.
| База данных | Идентификатор |
| Uniprot (ID) | AAT_AQUAE |
| Uniprot (AC) | O67781 |
| Uniref50 | UniRef100_O67781, UniRef90_O67781, UniRef50_O67781 |
| RefSeq Proteins | NP_214350.1. WP_010881286.1, AAC07746.1, AE000657.1, NC_000918.1 |
| PDB | - |
Задание 2. Описание истории изменений записи Uniprot для белка аспартатаминотрансферазы.
Дата создания первой версии: 01/08/1998
Дата создания последней версии: 11/03/2015
Комментарий некоторых произошедших изменений:
- В 8 записях добавлена информация об особенностях частей последовательности (о цепи и сайтах связывания):
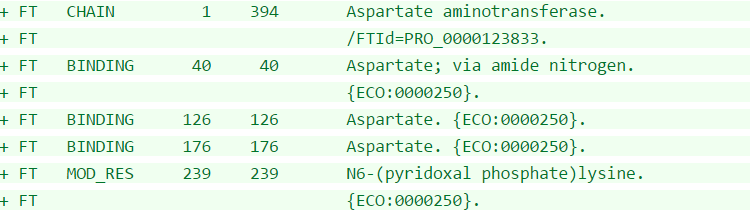
Рис.1. Пример добавления информации об особенностях частей последовательности.
- Добавлены 32 новые ссылки на другие базы данных:

Рис.2. Пример добавления ссылок на другие базы данных.
- Добавлена аннотация: 1 запись о номере публикации, 3 записи об авторах публикации, 2 записи о названии публикации, 1 запись о журнале и 10 записей с комментариями:

Рис.3. Пример добавления информации о публикации и комментариев.
Задание 3. Сравнение записи в Uniprot белка аминотрансферазы и белков-гомологов.
В этом задании рассматривались записи белка аспартатаминотрансферазы и 3 белков-гомологов: аспартатаминотрансферазы, аминотрансферазы I и II и аспартаттрансаминазы в организмах рода Thermus, родственным роду Aquifex. При анализе сравнивались функции, особенности строения, длина белка, наличие записи в PDB и источник появления записи.
Были обнаружены следующие сходства и различия:
- Проверенным экспериментально является только белок гомолога, по которому был произведен поиск остальных гомологов, последовательность остальных установлена аналитическим способом с помощью компьютерных методов по гомологии.
- Длина белка примерно одинакова у всех гомологов.
- Функции ни у одной записи не определены.
- Черты строения описаны только у аспартатаминострасферазы Aquifex aeolicus и Thermus thermophilus, при этом стуктуры Aquifex совпадают с частью структур thermophilus.
- PBD есть только для thermophilus.
В Таблице 2 представлены вышеупомянутые черты.
Задание 4. Пример обозначения полиморфизма в Uniprot.
- В Uniprot в строке FT (Feature Table) представлена информация об особенностях частей последовательности. В этой строке словом VARIANT обозначаются полиморфизмы, варианты в последовательности нуклеиновой кислоты.
Пример обозначения в Uniprot данного явления:
FT VARIANT 49 49 A -> P (in dbSNP:rs713598).
Пример взят из нуклеотидной последовательности геномной ДНК (идентификатор в Uniprot T2R38_HUMAN).
- В поле FT словом NON_STD обозначаются нестандартные (неканонические) аминокислоты: селеноцистеин и пирролизин - с соответствующими ключами (Selenocysteine, Pyrrolysine).
Пример обозначения в Uniprot данных явлений:- Селеноцистеин: FT NON_STD 196 196 Selenocysteine.
Пример взят из аминокислотной последовательности большой субъединицы формиатдегидрогеназы у организма Escherichia coli (strain K12) (идентификатор в Uniprot FDNG_ECOLI).
- Пирролизин: FT NON_STD 356 356 Pyrrolysine. {ECO:0000250}.
Пример взят из последовательности белка диметиламинметилтрансферазы MtbB1 у организма Methanosarcina acetivorans (strain ATCC 35395 / DSM 2834 / JCM 12185 / C2A) (идентификатор в Uniprot MTBB1_METAC).
- Селеноцистеин: FT NON_STD 196 196 Selenocysteine.
- Пример обозначения посттрансляционной модификации белка (обозначается словом MOD_RES - модифицированный аминокислотный остаток) на примере фосфорилирования тирозина в последовательности опухолевого белка p73 у человека (идентификатор в Uniprot P73_HUMAN):
FT MOD_RES 28 28 Phosphotyrosine; by SRC and HCK.
- Пример обозначения изоформ белка - продуктов альтернативного сплайсинга (обозначение VAR_SEQ) в последовательности опухолевого белка p73 у человека (идентификатор в Uniprot P73_HUMAN):
FT VAR_SEQ 400 636 Missing (in isoform 11).
- В той же последовательности присутствует пример аминокислотного остатка, участвующего в связывании иона (обозначение в FT - METAL):
FT METAL 262 262 Zinc. {ECO:0000250}.
- Сайты белка, участвующие в связывании с ДНК, обозначаются DNA_BIND (на примере белка STE12 в организме Kluyveromyces lactis (strain ATCC 8585 / CBS 2359 / DSM 70799 / NBRC 1267 / NRRL Y-1140 / WM37) (Yeast) (Candida sphaerica), идентификатор в Uniprot STE12_KLULA).
FT DNA_BIND 51 161 {ECO:0000250}.
- Дисульфидные связи в белке (обозначаются DISULFID) покажем на примере белка 108 у организма Solanum lycopersicum (Tomato) (Lycopersicon esculentum), идентификатор в Uniprot 108_SOLLC).
FT DISULFID 41 77 {ECO:0000250}.
- Удаление инициаторного метионина обозначается словом INIT_MET с ключом Removed (на примере неохарактеризованного человеческого белка C1orf198, идентификатор в Uniprot CA198_HUMAN).
FT INIT_MET 1 1 Removed. {ECO:0000269|PubMed:12665801,
FT ECO:0000269|PubMed:22814378}.