Семейства белковых доменов
Для выполнения заданий данного практикума была взята аминокислотная последовательность белка аспартатаминотрансферазы из организма бактерии Aquifex aeolicus VF5 (идентификатор в базе данных RefSeq NP_214350). Данная последовательность доступна в формате fasta.
Задание 1. Поиск доменов Pfam в последовательности моего белка.
Pfam - база данных, содержащая информацию о выравниваниях белковых доменов.
Для выполнения данного задания было необходимо осуществить поиск доменов по последовательности белка на сайте Pfam в разделе Sequence search. По умолчанию отбор производится по порогу E-value = 1.0 (Условия поиска).
Была найдена всего одна Pfam A находка, поиск по Pfam B не производился (Pfam A содержит курируемые данные, а Pfam B - некурируемые). Было найдено семейство Aminotran_1_2 (Aminotransferase class I and II), входящее в клан PLP_aminotran (CL0061), содержащий пиридоксальфосфат-зависящие ферменты. О самом семействе нет никаких данных, кроме того, что с другими последовательностями клана его объединяет такая черта, как связывание пиридоксальфосфатной группы с остатком лизина, помимо этого, на основе особенностей последовательностей данное семейство подразделяется на 2: класс I и II. Молекулярная функция заключается в трансферазной активности и связывании пиридоксальфосфатной группы.
С данным семейством было найден один матч входной последовательности длиной 363 остатка, другие характеристики находки: Bit Score 276.2, E-value 3.4e-82.
Был сохранен Seed данного семейства (ссылка Alignments), содержащий 48 последовательностей (всего же последовательностей 52439), в формате fasta.
Также было получено изображение Seed в JalView (окно Seed), которое можно увидеть и на Рис. 1.
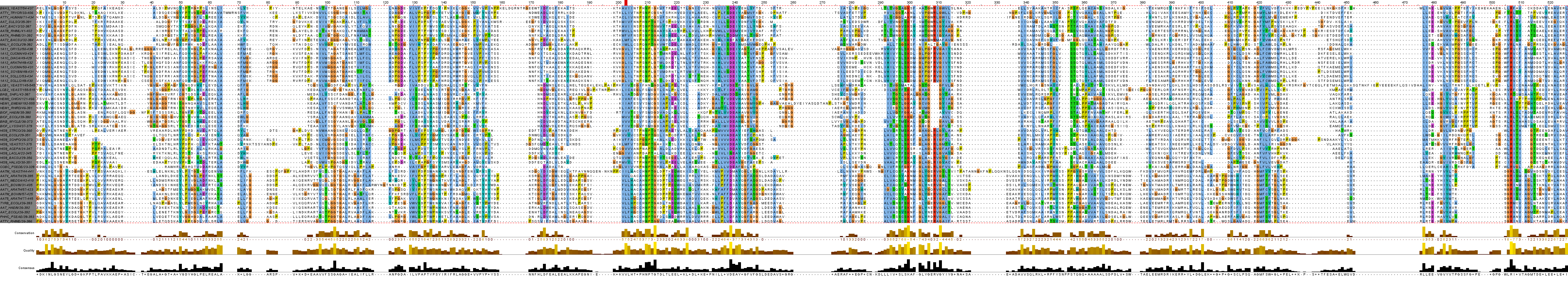
Задание 2.
Также я выбрала в данном выравнивании несколько колонок (237-242) в качестве блока (колонки отмечены символом В на Рис. 2 и в окнах JalView block и block_closer).
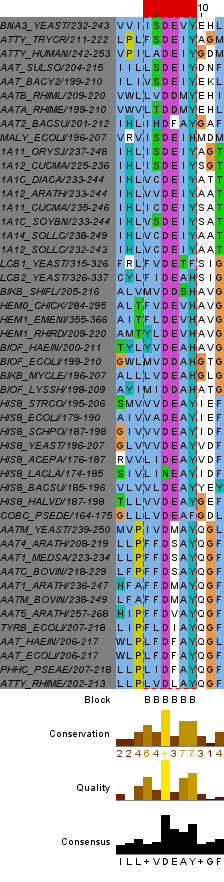
Блок в формате fasta.
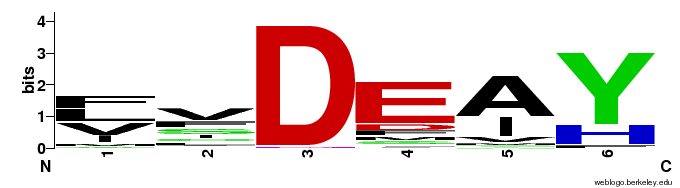
По последовательностям на участке данного блока на сервере Weblogo был получен logo данного мотива, показывающий распространенность различных остатков в каждой из позиций (Рис. 3).
Также из загруженного в JalView выравнивания Seed была сохранена консенсусная последовательность блока (в формате fasta).
Задание 3.
В данном задании я нашла последовательность из Seed, наиболее схожую с консенсусной последовательностью белка. Для это я построила парные глобальные выравнивания каждой последовательности из Seed c консенсусной (в формате fasta).
С результатами можно ознакомиться в Таблице 1.
Таблица 1. Сравнение Score парных глобальных выравниваний последовательностей из Seed c консенсусной.
| Идентификатор последовательности в UniProt | Score |
| 1A11_CUCMA | 585.0 |
| 1A11_ORYSJ | 540.0 |
| 1A12_ARATH | 629.0 |
| 1A12_CUCMA | 527.5 |
| 1A12_SOLLC | 634.5 |
| 1A14_SOLLC | 625.0 |
| 1A1C_DIACA | 623.0 |
| 1A1C_SOYBN | 614.0 |
| AAT_BACY2 | 346.0 |
| AAT_ECOLI | 483.5 |
| AAT_HAEIN | 465.5 |
| AAT_SULSO | 357.5 |
| AAT1_ARATH | 497.5 |
| AAT1_MEDSA | 525.0 |
| AAT2_BACSU | 317.0 |
| AAT4_ARATH | 482.5 |
| AAT5_ARATH | 489.5 |
| AATA_RHIME | 353.5 |
| AATB_RHIML | 311.5 |
| AATC_BOVIN | 477.0 |
| AATM_BOVIN | 450.5 |
| AATM_YEAST | 295.0 |
| ATTY_HUMAN | 194.5 |
| ATTY_RHIME | 370.5 |
| ATTY_TRYCR | 235.5 |
| BIKB_MYCLE | 214.0 |
| BIKB_SHIFL | 210.0 |
| BIOF_ECOLI | 136.5 |
| BIOF_HAEIN | 217.0 |
| BIOF_LYSSH | 172.5 |
| BNA3_YEAST | 246.5 |
| COBC_PSEDE | 158.5 |
| HEM0_CHICK | 224.0 |
| HEM1_EMENI | 225.5 |
| HEM1_RHIRD | 225.0 |
| HIS8_ACEPA | 233.5 |
| HIS8_BACSU | 247.5 |
| HIS8_ECOLI | 219.0 |
| HIS8_HALVD | 254.0 |
| HIS8_LACLA | 172.0 |
| HIS8_SCHPO | 174.0 |
| HIS8_STRCO | 220.5 |
| HIS8_YEAST | 185.0 |
| LCB1_YEAST | 137.5 |
| LCB2_YEAST | 124.5 |
| MALY_ECOLI | 175.5 |
| PHHC_PSEAE | 428.5 |
| TYRB_ECOLI | 461.5 |
По данным таблицы видно, что наиболее похожа на консенсусную последовательность с идентификатором 1A12_SOLLC.
Задание 4.
В данном задании, основываясь на полученном в прошлом задании logo, я составила сильный и слабый паттерны для поиска последовательностей, содержащих мотив с данным паттерном, по базе данных Swiss-Prot на сервере http://prosite.expasy.org/scanprosite/.
Сильный паттерн призван находить только гомологичные находки, что может достигаться усилением позиций и жертвой некоторых находок.
Составить сильный паттерн по logo длиной всего 6 остатков является непростой задачей, так как крайне высока вероятность, что такой короткий мотив будет найден в огромном количестве негомологичных последовательностей. Задачу усугубляет то, что позиции данного участка малоконсервативные, то есть имеется много вариантов даже среди последовательностей семейства, что не позволяет найти только гомологов (сервис даже не выдает результат, так как находок слишком много). Что интересно, даже при учитывании всех вариантов остатков в паттерне, не находится исходная последовательность аспартатаминотрансферазы (последовательности аспартатаминотрансфераз не находятся вообще, самый близкий результат при "прогоне" паттерна=консенсуса - гистидинолфосфатаминотрансферазы), а при сравнении данного участка в консенсусной последовательности и в исходном белке можно предположить, что в данном участке могли произойти индели:
FCUERG (участок исходной последовательности)
FVDEAY (тот же участок в консенсусной последовательности)
Попытка учесть индели приводит программу в нерабочее состояние.
Если попробовать добавить в паттерн по несколько остатков по бокам от паттерна, то можно получить немного более приятные результаты. В итоге было принято решение добавить в паттерн 3 остатка справа от блока и 1 слева.
Окончательный вариант паттерна: [IAV]-[VIM]-[ILVFACST]-D-E-[AVSTI]-Y-[GYIVSMD]-[GDHKVLE]-[FILGMV] (186 хитов в 186 последовательностях).
Изначально я составила паттерн, просто перечислив в каждой из позиций все имеющиеся в последовательностях Seed варианты остатков. Разумеется, это привело к выдаче огромного количества негомологичных последовательностей. Тогда были произведены следующие изменения:
- В первой позиции я отказалась от гидрофильных остатков и пролина в пользу гидрофобных, так как только они характерны для моего семейства.
- В позиции 2 пришлось усилить позицию, оставив лишь V, I, M, так как остальные остатки в колонке соответствуют в большей степени другим семействам.
- Позиции 4, 5, 7 усилены до единственной, самой распространенной буквы. Эти позиции одинаковы во всех находках, очевидно, они консервативны для данного семейства.
- Остальные позиции содержат остатки, сильно различающиеся по функциям и их распространенности в колонке, поэтому в них оставлен почти весь имеющийся набор остатков, только исключены в 2 с конца колонке - А, а в предпоследней - Q, так как они способствуют выдаче большого количества лишних последовательностей.
В результате все полученные последовательности относятся к семейству аминотрансфераз. Большую часть составляют гистидинолфосфатаминотрансферазы, также находятся тирозинаминотрансферазы и предполагаемые аспартатаминотрансферазы. Аспартатаминотрансферазы данный паттерн не находит, потому что, как уже было сказано выше, их паттерн в данном блоке сильно различается, что позволяет предположить наличие инделей в данном блоке (или просто высокую вариативность).
Слабый паттерн призван находить все находки, содержащие данный мотив (в том числе значительное количество негомологичных находок).
Для слабого мотива я ослабила первые две колонки до всех гидрофобных остатков, встречающихся в позициях, в 4 колонку добавила D и гидрофобные остатки, а также вернула остатки A и Q в 8 и 9 колонки соответственно, так как это позволило найти гораздо больше находок из моего семейства.
Слабый паттерн: [IAVFLM](2)-[ILVFACST]-D-[DEIAVFLM]-[AVSTI]-[HFY]-[AGYIVSMD]-[QGDHKVLE]-[FILGMV] (1221 находка в 1221 последовательности).
К сожалению, количество включений из других семейств колоссальное, что происходит за счет того, что выбранный блок содержит много малоконсервативных позиций, различающихся даже внутри семейства. В частности, помимо аминотрансфераз были получены находки: из семейства карбоксилтрансфераз, лиаз, дегидрогеназ, рецепторы брадикинина, сигнальные белки сети убиквитинизации, субъединицы АТФ-синтазы. Из "нужного" к находкам добавились несколько аспартатаминотрансфераз и глутаматпируватаминотрансфераз. Думаю, что если "разумно" расширять паттерны, то это все возможные находки. К сожалению, индели учесть не удается, находок получается больше, чем может выдать программа.
Задание 5.
Также я осуществила поиск последовательностей мотивов в последовательностях Seed с помощью сервиса MEME. Поиск производился до трех мотивов в каждой последовательности, а также использовалась опция Any number of repetitions (возможны находки нескольких повторяющихся мотивов в одной последовательности).
Cсылка на последовательности Seed.
Ссылка на результаты работы программы МЕМЕ.
Программа обнаружила три мотива (logo мотивов можно наблюдать на Рис.4 (a - c)).

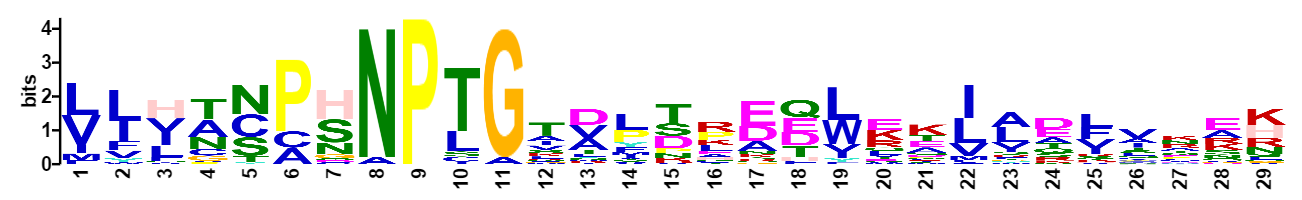

Из 48 последовательностей Seed все три мотива были найдены в 8 последовательностях, в одной - только второй, в двух - только первый, во всех остальных - первый и второй.
Ни в одной последовательности не было найдено повторяющихся мотивов.