Сравнение геномов
Задание 1. Построить карту сходства хромосом двух родственных бактерий.
Для выполнения данного задания я взяла два разных вида рода Brucella, у каждого из которых имеются 2 хромосомы: Brucella abortus 104M (хромосома 1, хромосома 2) и Brucella suis bv. 2 strain Bs143CITA (хромосома 1, хромосома 2) (ссылки даны на соответствующие последовательности в формате fasta).
Чтобы сравнить геномы двух бактерий, было нужно запустить алгоритм blastn для выравнивания 2 последовательностей (blast2seq) с сайта NCBI. Среди результатов работы данного алгоритма нас больше всего интересовала карта локального сходства геномов бактерий - изображение, позволяющее понять, какой участок генома одной бактерии соответствует определенному участку генома другой бактерии. Карта локального сходства полезна тем, что она отражает крупные эволюционные события, такие как вставки, делеции, транслокации, инверсии.
Я провела выравнивание каждой хромосомы одной бактерии с каждой хромосомой другой бактерии (алгоритм работал с параметрами по умолчанию). С результатом можно ознакомиться на Рис. 1а-d.
- Выравнивание хромосом 1 Brucella abortus 104M и Brucella suis bv. 2 strain Bs143CITA.
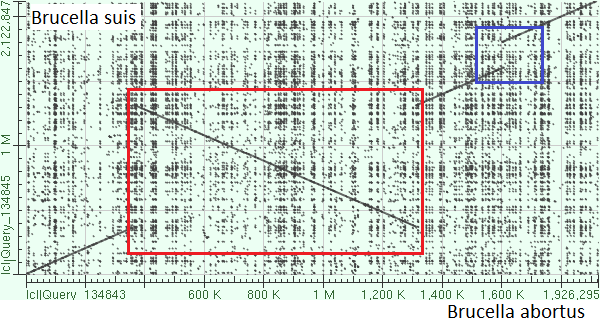
Рис. 1а. Карта локального сходства последовательностей хромосом 1 Brucella abortus 104M (по Ох) и Brucella suis bv. 2 strain Bs143CITA (по Оу). Рамками красного и синего цветов обозначены крупные эволюционные события. Координаты участков не указаны ввиду слишком сильной компактизации изображения (участки слишком длинные).
На данной карте локального сходства отображены следующие особенности:- Это выравнивание прямых последовательностей (когда выравниваются прямая с комплементарной, линии на карте идут из левого верхнего угла в правый нижний).
- Красной рамкой выделена инверсия очень протяженного участка.
- То, что хромосома Brucella abortus 104M примерно на 200 000 п.н. длиннее, чем хромосома Brucella suis bv. 2 strain Bs143CITA, сразу спровоцировало ожидание делеций или вставок. И действительно, синей рамкой выделено место, в котором либо произошла вставка в последовательность Brucella abortus 104M, либо делеция в хромосоме Brucella suis bv. 2 (т.к. последний участок выравнивания расположен по оси Ох позже, чем должен был). Вариант с транслокацией с 1 хромосомы на другую тоже можно рассмотреть.
- Выравнивание хромосом 2 Brucella abortus 104M и Brucella suis bv. 2 strain Bs143CITA.
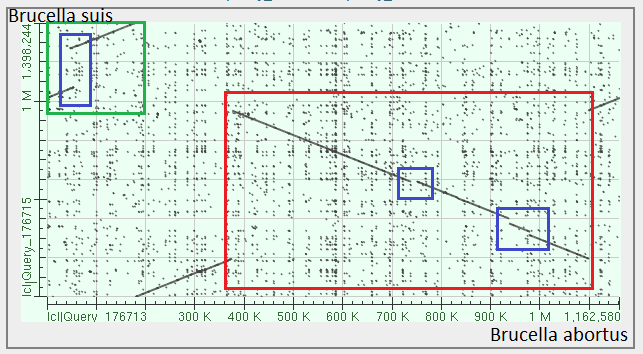
Рис. 1b. Карта локального сходства последовательностей хромосом 2 Brucella abortus 104M (по Ох) и Brucella suis bv. 2 strain Bs143CITA (по Оу). Рамками красного, синего и зеленого цветов обозначены крупные эволюционные события.
Эта карта еще более интересна:- Снова выравнивание прямых последовательностей (все-таки одна крупная инверсия происходит с большей вероятностью, чем несколько небольших; да и вообще было бы странно
ожидать выравнивания прямой цепи с комплементарной, поэтому в дальнейшем эта особенность отмечаться не будет).
- Красной рамкой выделена крайне масштабная инверсия.
- Синими рамками выделены места делеций/вставок (причем самая крупная в 1 слева рамке).
- Заметим, что 1 нуклеотид Brucella suis соответствует 176713 у Brucella abortus 104M, а начало хромосомы у Brucella abortus 104M выравнивается с концом хромосомы Brucella suis (зеленая рамка). Следовательно, у одного из видов произошла транслокация начального участка в конец (или наоборот , опять же определить нельзя).
- Выравнивание хромосомы 2 Brucella abortus 104M и хромосомы 1 Brucella suis bv. 2 strain Bs143CITA.
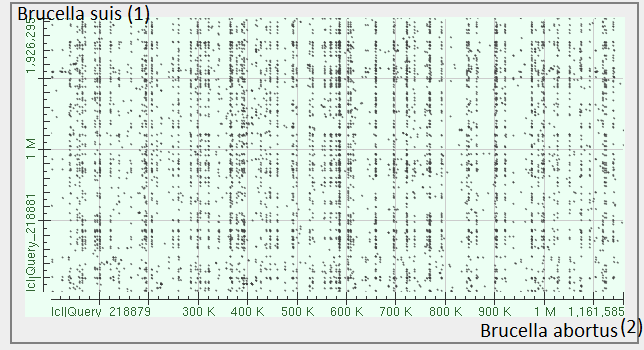
Рис. 1с. Карта локального сходства последовательностей хромосомы 2 Brucella abortus 104M (по Ох) и хромосомы 1 Brucella suis bv. 2 strain Bs143CITA (по Оу).
Никаких крупных участков в выравнивании не наблюдается. Это, в принципе, ожидаемый результат.
- Выравнивание хромосомы 1 Brucella abortus 104M и хромосомы 2 Brucella suis bv. 2 strain Bs143CITA.
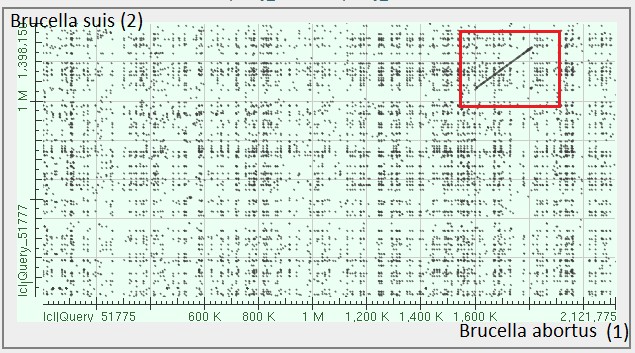
Рис. 1d. Карта локального сходства последовательностей хромосомы 1 Brucella abortus 104M (по Ох) и хромосомы 2 Brucella suis bv. 2 strain Bs143CITA (по Оу). Красной рамкой выделено крупное эволюционное событие.
Приближаемся к разгадке тайны делеции/вставки в 1 и 2 выравниваниях:
Красной рамкой выделен участок выравнивания, одинаковый для обеих хромосом. Если приглядеться к его местоположению, то можно увидеть, что он расположен как раз в месте крупного эволюционного события, отображенного на 1 и 2 картах. Вывод: это была транслокация, а не вставка/делеция. Чтобы убедиться в этом, я провела выравнивание тем же алгоритмом хромосом 1 и 2 для каждой бактерии по отдельности. Не привожу карты локального сходства, так как они абсолютно идентичны Рис. 1с. Таким образом, между хромосомами одной бактерии не было выявлено крупных участков сходства, то есть то событие - именно транслокация, а не делеция/вставка (однако пока неизвестно, от 1 хромосоме ко 2 или наоборот).
Чтобы попытаться выяснить природу этой транслокации, я "вырезала" из последовательности хромосомы 1 Brucella abortus 104M примерно соответствующий участок (с 1600000 по 1800000 п.н. - это определяется со шкалы на карте локального сходства) с помощью команды seqret пакета EMBOSS. Для полученной последовательности запустила blastn с сайта NCBI, ограничив находки родом Brucella. Как видно на Рис. 2, у большинства представителей рода этот участок присутствует в 1 хромосоме, а у части штаммов вида Brucella suis (но не у всех!) этот участок находится во 2 хромосоме.
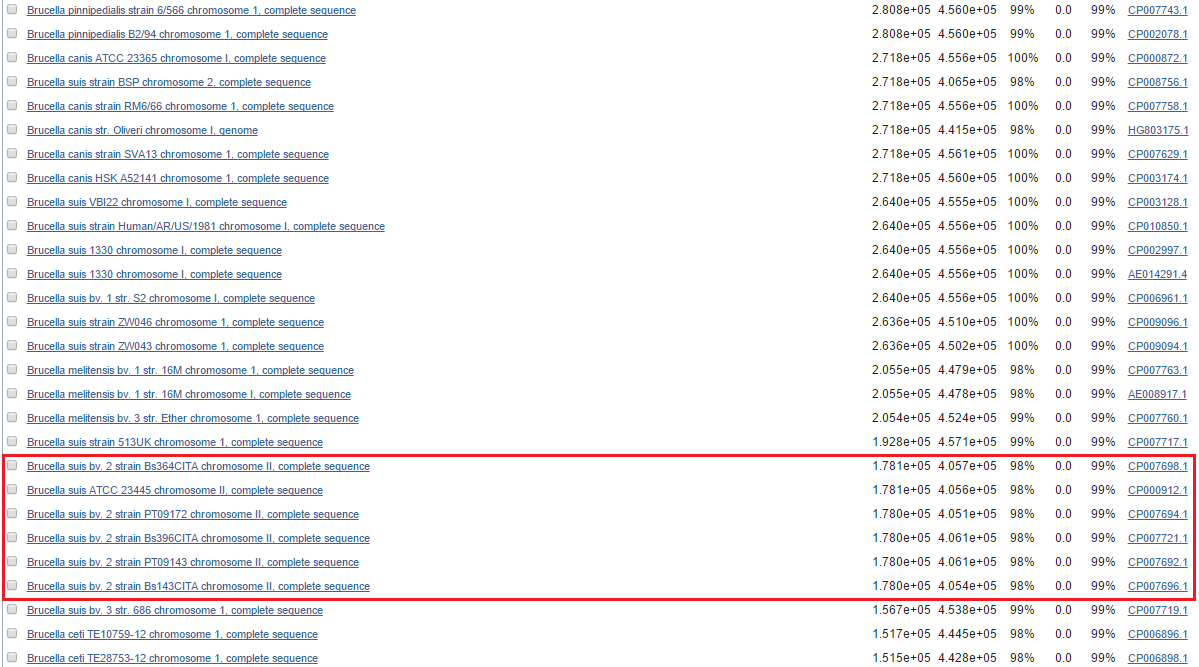
Рис. 2. Выдача blastn. В красной рамке находки, у которых интересующий участок находится во 2 хромосоме.
Данный участок слишком протяженный (напоминаю, около 200 000 п.н.), в нем содержится очень много генов и, полагаю, довольно много оперонов, поэтому крайне трудно определить, что эта транслокация дает организму, и как вообще влияет на жизнедеятельность то, в какой хромосоме находится ген. Я бегло просмотрела гены и обнаружила, что не могу даже высказать предположение о роли данной транслокации (если таковая вообще есть), так как совокупность генов на данном участке представляет собой некий "суп": там есть и гены, связанные с метаболизмом, и с ДНК- и РНК-полимеразами, и гены, кодирующие тРНК и рРНК, еще встречался оперон (предположительно), связанный с АТФазой.
Задание 2. Описать сходство и различие геномов близкородственных бактерий.
Для выполнения данного задания использовался метод построения нуклеотидного пангенома (NPG) с помощью пакета NPG-explorer.
Нуклеотидный пангеном - специальный формат для множественного выравнивания геномов, ориентированный на геномы близкородственных бактерий или архей.
Для выполнения этого задания я выбрала 4 штамма вида Brucella abortus, геном каждого из которых представлен двумя хромосомами: Brucella abortus bv. 1 str. 9-941, Brucella abortus bv. 2 str. 86/8/59, Brucella abortus bv. 6 str. 870, Brucella abortus bv. 9 str. C68.
Для работы пакета NPG-explorer было необходимо создать файл genomes.tsv , в котором содержится информация о том, откуда брать последовательности геномных ДНК и аннотации генов.
Затем я с помощью команд создала файл npge.conf, в котором содержатся параметры программы. Я изменила параметр MIN_IDENTITY на рекомендованное программой для моего случая значение 0.877.
Далее я получила нуклеотидный пангеном и много файлов с аналитической информацией, которую я визуализовала, скачав файл qnpge.exe с диска Р и запустив на своем компьютере.
Результаты сравнения геномов с помощью пакета NPGE
- G-блоки
Глобальные блоки (G-блоки) представляют собой синтеничные участки - объединения стабильных блоков. Они состоят из последовательно идущих во всех геномах s-блоков, перемежающихся блоками других типов (r-, h-, u- и m-), которые находятся внутри i-блоков.
Из данных файла global-blocks/blocks.gbi я выяснила, что всего в обеих хромосомах присутствуют 8 блоков (в идентификаторе блока первая буква обозначает тип блока, первое число - количество фрагментов в блоке и второе число - чило позиций в выравнивании блока):- g8x152
- g8x144
- g8x125
- g8x109
- g8x108
- g8x103
- g8x102
- g8x101
* Интересно, что для всех блоков, кроме g8x109, число позиций в выравнивании действительно соответствует указанному в идентификаторе. Для блока g8x109 средняя длина составляет 108 позиций, а минимальная - даже 107.
Далее из файла global-blocks/blocks.blocks я получила информацию о порядке расположения блоков хромосоме для каждого генома. Для удобства я сделала файл в Excel, где транспонировала данные в столбцы и удалила строчки, не содержащие g-блоков (ссылка). Как видно из Рис. 3а1, в хромосоме 1 для всех штаммов сохраняется одинаковый порядок g-блоков:- g8x103 >
- g8x144 >
- g8x102 >
- g8x101 <
- g8x108 >
- g8x109 >
- g8x125 >
- g8x152 <
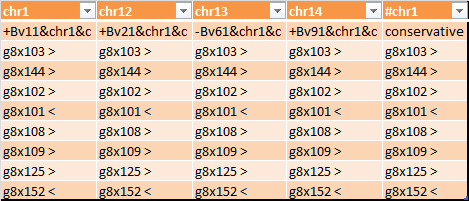
Рис. 3a1. Таблица с последовательностью g-блоков для хромосомы 1 всех штаммов.
При этом все блоки расположены на прямой цепи (символ ">"), кроме блоков g8x101 и g8x152 (символ "<").
На Рис. 3а2 показан участок выравнивания g- и i-блоков на хромосоме 1. Рисунок подтверждает совпадение порядка расположения блоков для всех штаммов.
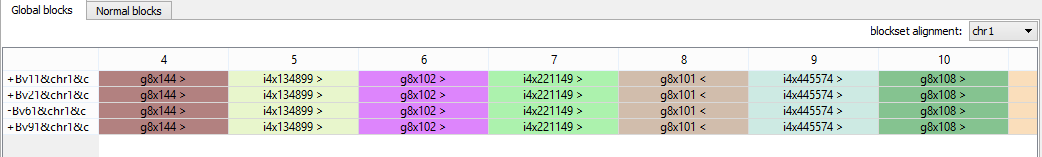
Рис. 3a2. Участок выравнивания i- и g-блоков для хромосомы 1 всех штаммов.
Ситуация с хромосомой 2 не так проста (Рис. 3b1). Если принять за эталон последовательность блоков в хромосоме у штамма Brucella abortus bv. 1 str. 9-941 (1 столбец), то у штамма Brucella abortus bv. 2 str. 86/8/59 сравнению с ним происходит транслокация трех последних блоков (g8x103, g8x144, g8x125), а у двух оставшихся штаммов - транслокация с инверсией (инверсию видно по изменению порядка следования блоков и по замене символов "<" и ">" на противоположный).
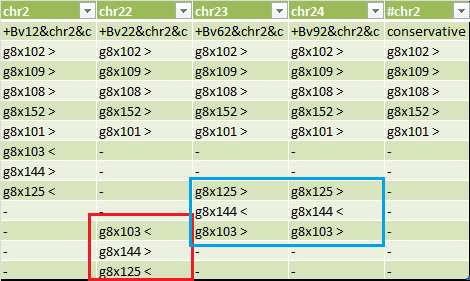
Рис. 3b1. Таблица с последовательностью g-блоков для хромосомы 2 всех штаммов. Красной рамкой выделена транслокация, синей - инверсия.
На Рис. 3b2 показан участок выравнивания блоков на хромосоме 2, который иллюстрирует наличие крупных эволюционных изменений в разных геномах бактерий.
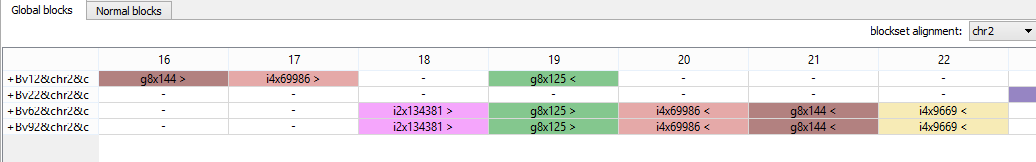
Рис. 3b2. Участок выравнивания i- и g-блоков для хромосомы 2 всех штаммов.
- S-блоки
Стабильные блоки (S-блоки) - ядро генома (информация была взята из файла href='pangenome/pangenome.info'>pangenome/pangenome.info).
- Число S-блоков: 8 (в 1 g-блоке только 1 s-блок)
- Cуммарная длина (процент от длины генома в среднем): 7540 (0.05%)
- Процент консервативных позиций в объединенном выравнивании s-блоков (т. е. сходство геномов): 0.916812
- R-блоки (блоки с повторами). Использовала данные файла pangenome/pangenome.bi.
r26x103 - самый часто встречающийся участок (всего 26 повторов во всех геномах, в обеих хромосомах, везде от 2 до 4 раз).
Длина: 103 позиции.
При этом некоторые последовательности данного повтора совсем не содержат генов (аннотированных программой): по одной во второй хромосоме у каждого штамма, кроме Brucella abortus bv. 9 str. C68 - у него 2; также по одной в первой хромосоме у штаммов Brucella abortus bv. 9 str. C68 и Brucella abortus bv. 6 str. 870. Я провела поиск алгоритмом blastn соответствующей последовательности у представителей вида Brucella abortus и затем проверила по аннотации в GenBank, нет ли в исследуемом участке у указанных выше организмов генов некодирующих РНК. У большинства штаммов в соответствующем участке присутствует мобильный генетический элемент, опознающийся как псевдоген, формальная трансляция которого похожа на BRA0551 (было определено по сходству с IS711 транспозазой ORFB); либо данный участок непосредственно аннотирован как повтор (repeat_region) (Рис. 4a).
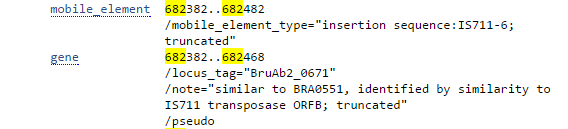
Рис. 4a. Информация о гене из GenBank.
У других последовательностей в области этого повтора присутствуют следующие гены: некоего гипотетического белка (либо DK55, либо BruAb1), регуляторный белок фотосинтетического аппарата, белок DDE-домена транспозазы, пептидазы (предположительной). Также есть крайне интересный пример: в 1 хромосоме у Brucella abortus bv. 1 str. 9-941 в одном из повторов присутствует ген белка из семейства белков фагового хвоста. Еще есть странные находки: во второй хромосоме у Brucella abortus bv. 1 str. 9-941 2 раза встречается некий ген, обозначенный "00000000". По аннотации генов в GenBank обнаружила, что данные последовательности тоже принадлежат мобильному элементу на комплементарной цепи. Таким образом, можно заключить, что этот повтор по большей части связан с вирусным вмешательством в геном бактерии. На Рис. 4b - участок выравнивания.
Рис. 4b. Участок выравнивания блока r26x103 для всех штаммов.
- H-блоки ("полустабильные" блоки)
На примере этих блоков предлагалось исследовать крупные делеции. Чтобы без труда попасть "в яблочко", я искала идентификатор hn, где n - число, не равное 4 (так как чаще всего 4 фрагмента просто встречаются в одном типе хромосомы во всех геномах).
Для примера я взяла блоки:- h3x499 (длина 499 позиций) - участок присутствует во второй хромосоме штаммов Brucella abortus bv. 1 str. 9-941, Brucella abortus bv. 2 str. 86/8/59 и
Brucella abortus bv. 6 str. 870, то есть в оставшемся штамме (Brucella abortus bv. 9 str. C68) произошла делеция этого участка (или наоборот во всех штаммах,
кроме последнего, закрепилась вставка данного участка). Если запустить по этой последовательности blastn, то можно увидеть, что у большинства штаммов вида она присутствует (Рис.
5a). В данном участке частично содержится ген никель-зависимого транскрипционного фактора. На Рис. 5b - участок выравнивания.
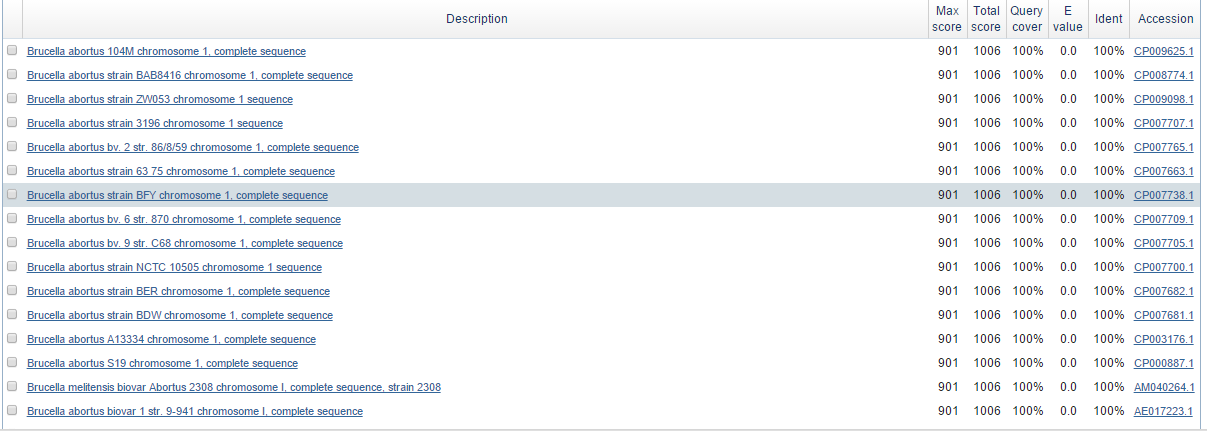
Рис. 5a. Информация о последовательности h3x499 (выдача blastn).
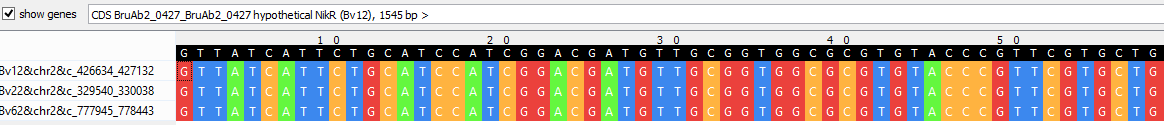
Рис. 5b. Участок выравнивания хромосомы 2 для штаммов Brucella abortus bv. 1 str. 9-941, Brucella abortus bv. 2 str. 86/8/59 и Brucella abortus bv. 6 str. 870.
- h6x105 (длина 105 позиций) - участок присутствует в обеих хромосомах у штаммов Brucella abortus bv. 1 str. 9-941 и Brucella abortus bv. 2 str. 86/8/59, а
у остальных штаммов - только в хромосоме 1, то есть в хромосоме 2 у них произошла делеция. Участок выравнивания, на котором видно, что довольно много неидентичных позиций и гэпов, на Рис. 5с.
- h3x499 (длина 499 позиций) - участок присутствует во второй хромосоме штаммов Brucella abortus bv. 1 str. 9-941, Brucella abortus bv. 2 str. 86/8/59 и
Brucella abortus bv. 6 str. 870, то есть в оставшемся штамме (Brucella abortus bv. 9 str. C68) произошла делеция этого участка (или наоборот во всех штаммах,
кроме последнего, закрепилась вставка данного участка). Если запустить по этой последовательности blastn, то можно увидеть, что у большинства штаммов вида она присутствует (Рис.
5a). В данном участке частично содержится ген никель-зависимого транскрипционного фактора. На Рис. 5b - участок выравнивания.
- U-блоки - уникальные последовательности из одного генома,у них нет гомологов среди всех геномов, кроме самой себя.
Присутствует всего один экземпляр такой последовательности: u1x104 (длина 104 позиции) из хромосомы 1 Brucella abortus bv. 9 str. C68. Участок последовательности приведен на Рис. 6a.

Рис. 6a. Участок последовательности блока u1x104.
Я провела поиск данного участка с помощью blastn. Алгоритм выдал 192 находки, среди которых были и те штаммы Brucella abortus, у которых ее, казалось бы, не должно быть, потому что данный участок был аннотирован программой как уникальный, относительно других исследуемых штаммов. Однако это можно объяснить тем, что участок слишком короткий (104 позиции), поэтому вероятность встретить ее у родственников слишком высока. К сожалению, создавшаяся ситуация не позволяет принять решение о возможности появления этого участка путем горизонтального переноса.
Но не будем отчаиваться, ведь в находках еще много интересного:- Нашлись последовательности клонов Stealth virus (красная рамка на Рис. 6b).
- Большое число находок (правда, с небольшим процентом покрытия) принадлежат гену субъединицы 1 цитохромоксидазы разных организмов (синяя рамка на Рис. 6b).
- Были найдены даже участки геномной ДНК 11 хромосомы человека (в названии последовательности сказано "клон 11 хромосомы" - не знаю, как это интерпретировать в рамках человеческого генома) (зеленая рамка на Рис. 6 b).
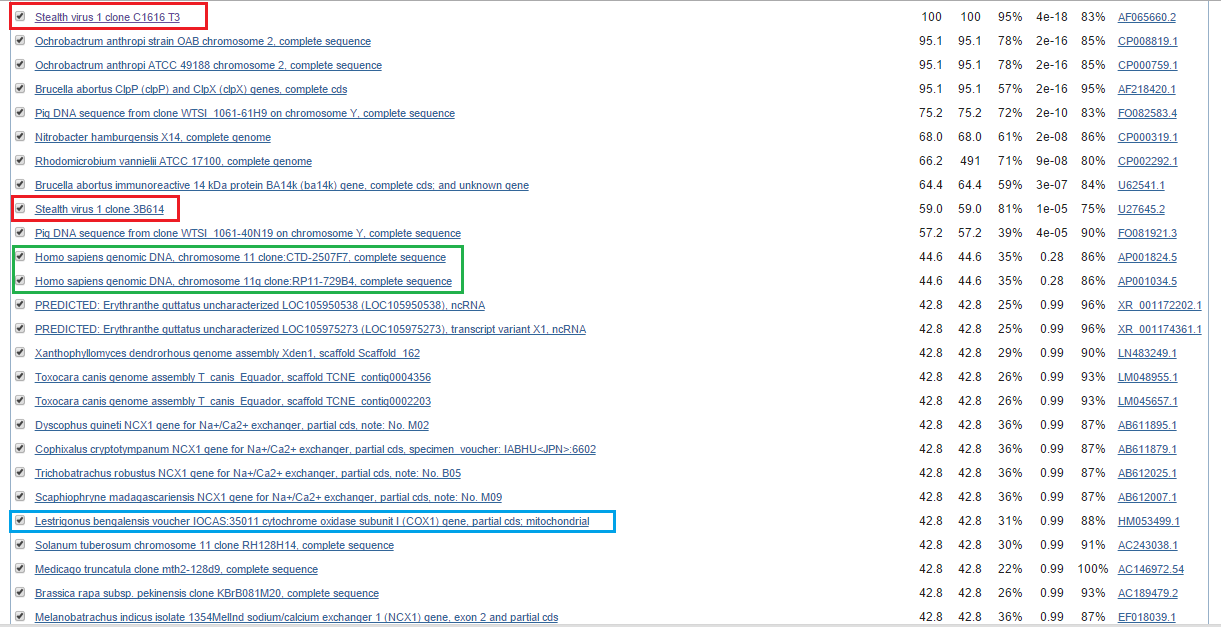
Рис. 6b. Выдача blastn для последовательности блока u1x104. В рамках красного, синего и зеленого цветов - интересные находки.
- Примеры расхождений между аннотациями генов из одного блока.
- На 1 хромосоме (блок h4x9606) присутствует очень интересная вещь: последовательность из Brucella abortus bv. 6 str. 870, видимо, взята на комплементарной цепи (значок "-"
около названия последовательности), поэтому в большинстве случаев в выравнивании на этом месте у этой бактерии какой-то один ген, а у всех остальных другой, причем длина
выравнивания участка может полностью совпадать у всех бактерий. Один из примеров на Рис. 7: у этой бактерии в этом месте ген белка из семейства соА-Трансфераз III, а у всех
остальных - ген регулятора транскрипции LysR, узнающего мотив "спираль-поворот-спираль".
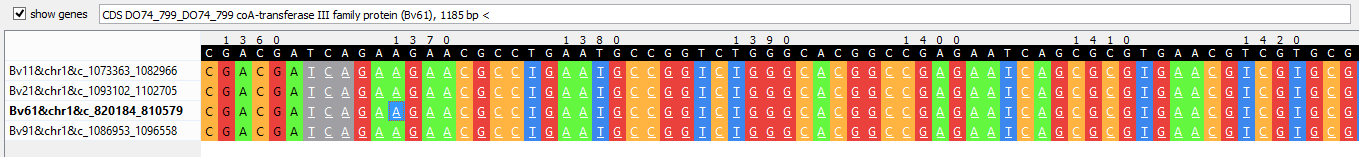
Рис. 7. Участок выравнивания последовательностей блока h4x9606.
- На 2 хромосоме (блок h4x24258) есть такой случай: сначала одновременно заканчивается (ген расположен на комплементарной цепи)
ген внутренней транспортной системы, зависящей от связывания, правда, только у 3 бактерий
из 4, что уже выделяется из общего случая (Рис. 8а). Затем в 2 из 3 геномов появляется новый стоп-кодон, перед которым не было старта предыдущего гена, и идет
ген внеклеточного связывания, и буквально через 30 позиций программа выделила старт-кодоны в этих последовательностях, хотя гены еще довольно долго продолжаются;
при этом в
третьей бактерии продолжается предыдущий ген (Рис. 8b). Потом у третьей бактерии предыдущий ген "заканчивается" (старт-кодон),
а стоп-кодон гена внеклеточного связывания располагается не сразу, как было у
двух других штаммов, а через небольшой пропуск (Рис. 8с).
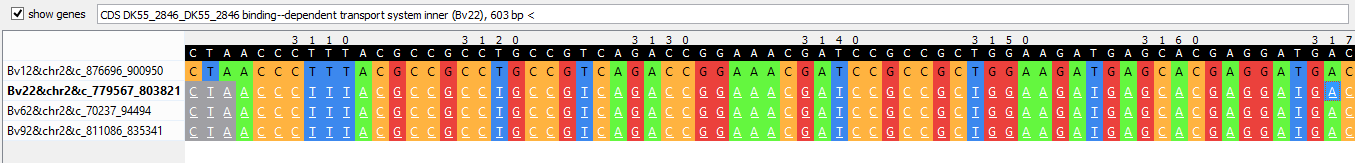
Рис. 8a. Участок выравнивания последовательностей блока h4x24258 (начало гена внутренней транспортной системы).
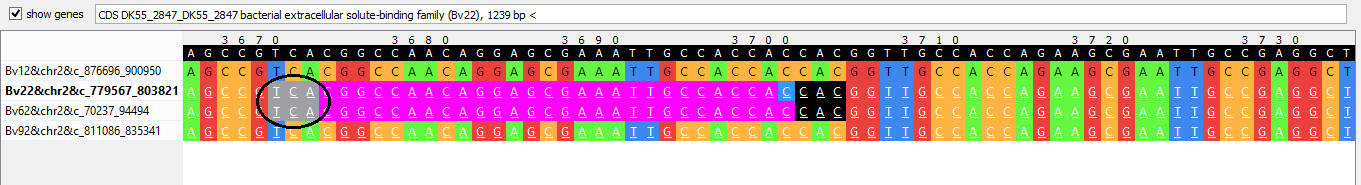
Рис. 8b. Участок выравнивания последовательностей блока h4x24258 (начало у 2 бактерий гена внеклеточного связывания).
Рис. 8с. Участок выравнивания последовательностей блока h4x24258 (начало гена внеклеточного связывания у оставшейся бактерии).
- На 1 хромосоме (блок h4x9606) присутствует очень интересная вещь: последовательность из Brucella abortus bv. 6 str. 870, видимо, взята на комплементарной цепи (значок "-"
около названия последовательности), поэтому в большинстве случаев в выравнивании на этом месте у этой бактерии какой-то один ген, а у всех остальных другой, причем длина
выравнивания участка может полностью совпадать у всех бактерий. Один из примеров на Рис. 7: у этой бактерии в этом месте ген белка из семейства соА-Трансфераз III, а у всех
остальных - ген регулятора транскрипции LysR, узнающего мотив "спираль-поворот-спираль".
Впечатления
Данная работа, в первую очередь, напомнила мне о том, что у бактерий, оставшихся в живых после вирусной инфекции, могут оставаться в хромосоме участки вирусного генома. И, как оказалось, не просто могут, а присутствуют в большом количестве.
Также мне понравилась "блоковая" система организации множетсвенного выравнивания: отделение стабильных и других функционально различных участков позволяет проще анализировать эволюционные события.