Чтение последовательностей по Сэнгеру
Задание 1. Получение последовательности фрагмента ДНК.
В данном задании было необходимо проанализировать хроматограммы из капиллярного секвенатора по Сэнгеру и на основе этого отредактировать последовательности, сгенерированные программой.
Капиллярный секвенатор по Сэнгеру выдает файлы с хроматограммой и последовательностью в формате .ab1. В моем случае анализировались результаты секвенирования ДНК некоего животного с Беломорской биологической станции МГУ, представленные в виде файлов с прямой (F в названии файла - 'forward chain') и обратной (R - 'reverse chain') цепями (прямая цепь, обратная цепь).
Для просмотра и редактирования результатов секвенирования использовалась программа Chromas (Lite).
Результатом выполнения задания являются:
- Выравнивание прямой и комплементарной к обратной последовательности с отмеченными измененными нуклеотидами в проекте JalView (JalView).
- Файл в формате fasta с "чистой" прочтенной последовательностью, то есть последовательностью, в которой принято решение о нахождении определенного нуклеотида в каждой позиции ("чистая" последовательность).
Workflow
Для начала я открыла в программе Chromas (Lite) файл с прямой последовательностью, а также в еще одном окне получила цепочку, комплементарную к обратной цепи (опция Reverse+Complement).
Для того, чтобы перейти к анализу и редактированию последовательности, требовалось выровнять прямую и комплементарную к обратной последовательности. Интересно, что первый читаемый нуклеотид на прямой последовательности (17 позиция) соответствует такому же нуклеотиду на комплементарной к обратной цепи под номером 53. Однако это вполне объяснимо, так как в начале перевернутой хроматограммы оказался конец прочтения обратной последовательности, длина которого может варьировать, а также в конце прочтения часто наблюдаются длинные нечитаемые области.
Известно, что не только конец, но и начало полученных при секвенировании последовательности и хроматограммы может иметь очень низкое качество и не читаться. Это часто происходит в из-за наличия коротких фрагментов, на которые отжигается праймер. Поэтому требовалось определить границы 5' и 3' нечитаемых областей и впоследствии удалить эти области.
Координаты нечитаемых областей указываются по прямой цепи.
- 5' нечитаемый участок (прямая): 1-17
- 3' нечитаемый участок (прямая): 674-717
- 5' нечитаемый участок (компплементарная к обратной): нуклеотиды не представлены в прямой цепи
- 3' нечитаемый участок (компплементарная к обратной): 652-683
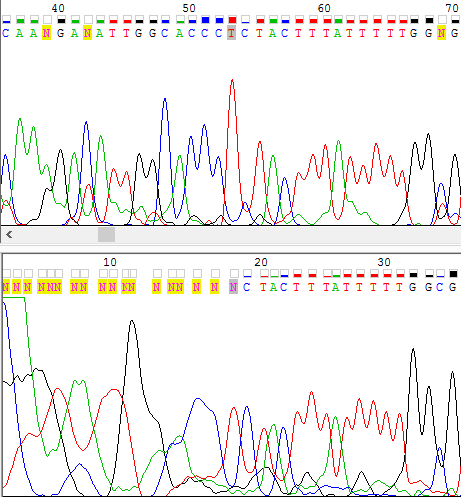
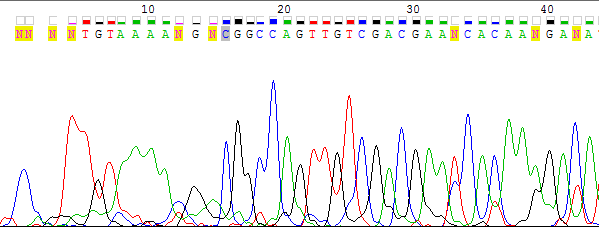
Здесь я хочу прокомментировать выбор границ участков. На Рис. 1 можно видеть проблемный участок 5' нечитаемой области на прямой последовательности (снизу) и соответствующий участок на комплементарной к обратной (сверху). Видно, что на прямой последовательности до 18 нуклеотида идет участок с плохим качеством хроматограммы, обозначенный нуклеотидами N (в позиции может быть любой из нуклеотидов). Но я посчитала, что уже начиная с 17 нуклеотида идут вполне читаемые пики, поэтому я решила заменить в прямой цепи в 17 позиции N на T. Это можно подтвердить на комплементарной к обратной последовательности, в которой в соотвествующей позиции наблюдается очень хороший пик тимина, а также совпадают дальнейшие пики.
Кстати, 5'нечитаемый участок комплементарной к обратной цепи довольно короткий (до 14 позиции включительно по этой цепи), и эти нуклеотиды уже не представлены на прямой цепи.
На Рис. 1a можно видеть 5'нечитаемый участок комплементарной к обратной цепи.
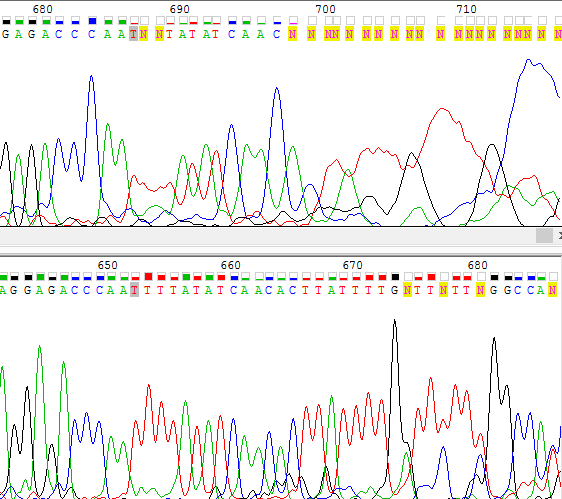
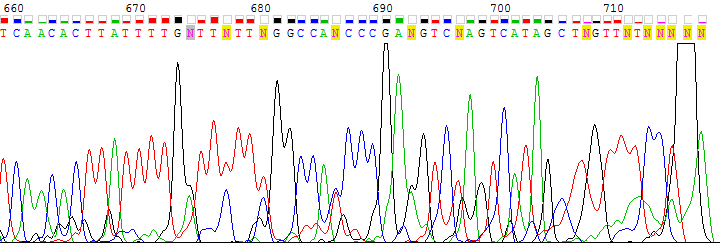
3' нечитаемую область не удалось проверить по комплементарной к обратной цепи, в ней нечитаемый участок начинается раньше в силу тех же причин, что и для несовпадения 5' участков прямой и комплементарной к обратной цепей. На Рис. 2 можно видеть, что нечитаемый участок комплементарной к обратной цепи (сверху) начинается приблизительно с 686 (652 по прямой цепи) нуклеотида, в прямой же цепи (снизу) после соответствующего нуклеотида идет еще десятка два довольно читаемых пиков, несмотря на повышенный уровень фона.
На Рис. 2a дополнительно показан 3' нечитаемый участок прямой цепи (с 674 позиции): хроматограмма низкого качества и много N в последовательности.
По поводу качества хроматограммы можно сказать следующее:
- В обеих цепях уровень сигнала примерно в 7 раз превышает уровень фона.
- По мере приближения к концам хроматограммы пики становятся ниже и шире в обеих цепях, часто исчезают четкие границы между пиками. Также в прямой сильнее выражен разброс пиков по размеру (общая масса более низких пиков прерывается очень высокими пиками), тогда как в комплементарной к обратной цепи пики чуть меньше различаются по высоте. Та же тенденция наблюдается и при сравнении уровня фона в двух цепях: в прямой фоновые пики сильно различаются по высоте, в комплементарной к обратной цепи высота пиков более однородна. Общим является увеличение фоновых пиков при приближении к концам хроматограмм.
- Сила сигналов в прямой цепи различается примерно в 2-3 раза при сравнении относительно низких и высоких пиков, в комплементарной к обратной цепи эта разница выражена слабее - примерно в 1.5-2 раза отличаются пики. Также нельзя не заметить, что высота пиков зависит от нуклеотида: в прямой цепи самые высокие пики принадлежат G (в 2.2 раза выше), немного ниже пики А (в 1.7 раз выше С и Т); в комплементарной к обратной цепи самые высокие пики у С (в 2 раза выше), затем идет Т (в 1.5 раза выше).
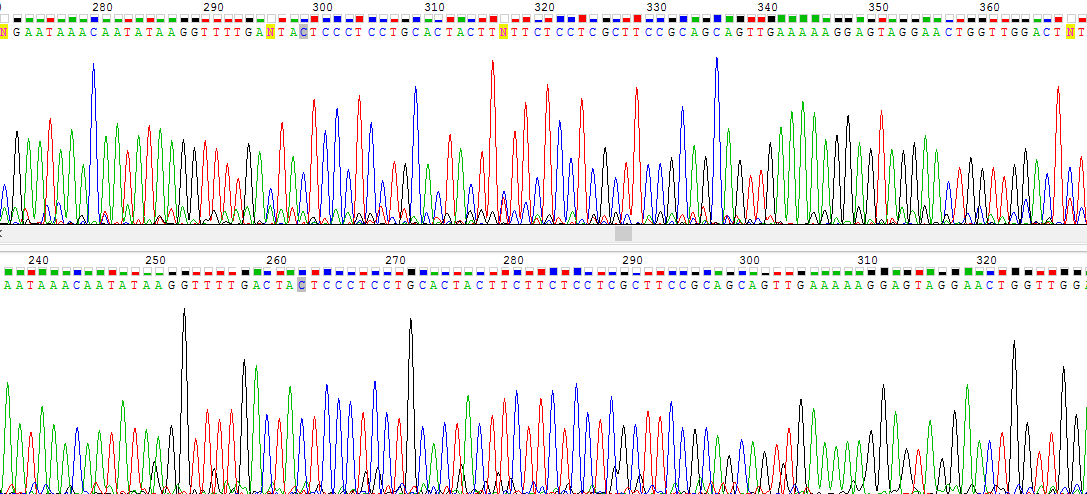
- В последовательности присутствует СТ-богатый участок (262-294 позиции по прямой цепи), который сильно бросается в глаза на комплементарной к обратной цепи, так как там пики этих нуклеотидов самые высокие (Рис. 3).
- На мой взгляд, обе хроматограммы довольно хорошие.
Далее требовалось проанализировать и отредактировать все "сложные места" обеих цепей, к которым относятся:
- шум выше среднего уровня шума и почти как сигнал (возможна замена нуклеотида)
- пик на нетипичном расстоянии от соседей - вклинился лишний или соседние пики нетипично удалены (возможны удаление или вставка буквы)
- вместо двух или более пиков - один широкий (аналогично)
Сложные места рекомендовалось проверять по второй цепочке. Все изменения обозначались маленькими буквами, а позиции, между которыми был удален нуклеотид, отмечены * в нижней строке выравнивания в проекте JalView.
В результате были получены fasta-файлы с отредактированными последовательностями (прямая цепь, комплементарная обратной), а также я получила "чистую" последовательность и сделала выравнивание этих последовательностей в программе JalView (проект JalView).
Для иллюстрации "сложных мест" на прямой цепи приведу несколько примеров:
- На Рис. 4 продемонстрировано несколько сложностей: во-первых, С в 17 позиции на прямой цепи (снизу) имеет очень низкий сигнал, близкий к фону. В комплементарной к
обратной цепи в соответствующей позиции вообще стоит N, так как там сигнал С тоже низкий, а сигнал Т максимален для фонового. Несмотря на то, что эта позиция довольно сомнительна,
я оставила в ней С, так как сигнал все-таки немного выше фонового и нет доказательств того, что это ошибочная вставка.
Также на Рис. 4 на прямой цепи (позиции 24-29) присутствуют нестандартные расстояния между пиками, но не наблюдается ни удаление нуклеотидов, ни объединение пиков, так что было принято решение оставить все как есть. А вот после 35 А вклинился лишний пик G, который на самом деле просто представляет собой один размытый пик (на второй цепи видны 2 пика G вместо 3 на прямой цепи), поэтому вклинившийся пик был удален.
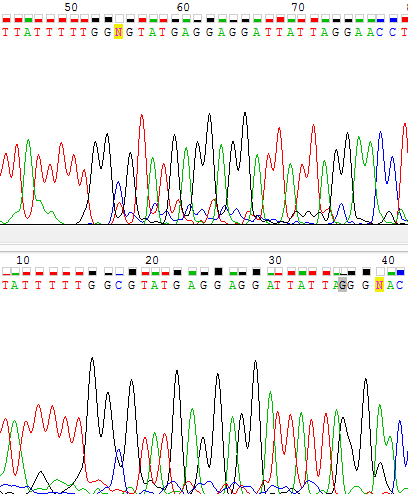
Рис. 4. Изображение проблемного участка прямой цепи (снизу) и соответствующего участка комплементарной к обратной цепи (сверху). G после 35 А на прямой цепи будет удален. С 17 останется на своем месте.
- На Рис. 5 приведено 2 примера конкуренции слабого сигнала с сильным шумом (позиции N 38 и 47). В обоих случаях благодаря второй цепи можно довольно достоверно
сказать, что в этих позициях должен быть А.
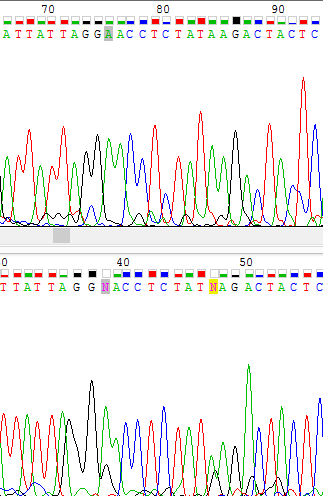
Рис. 5. Изображение проблемного участка прямой цепи (снизу) и соответствующего участка комплементарной к обратной цепи (сверху). N в позициях 38 и 47 должен быть заменен на А.
- Очень увлекательная проблема: в 340 позиции на прямой цепи стоит N, хотя под ним прекрасный пик С, а уровень фона там в пределах среднего. Видимо, это ошибка программы,
вторая цепб подтверждает наличие С в этой позиции (Рис. 6).
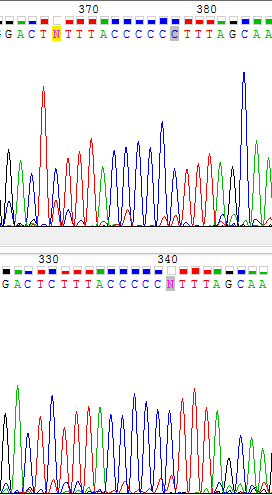
Рис. 6. Изображение проблемного участка прямой цепи (снизу) и соответствующего участка комплементарной к обратной цепи (сверху). N в позиции 340 должен быть заменен на С.
- Для сравнения привожу пример участка, где последовательность определяется однозначно (Рис. 7). Кстати сказать, сложных мест было совсем немного, хроматограмма прямой цепи очень
хорошая.
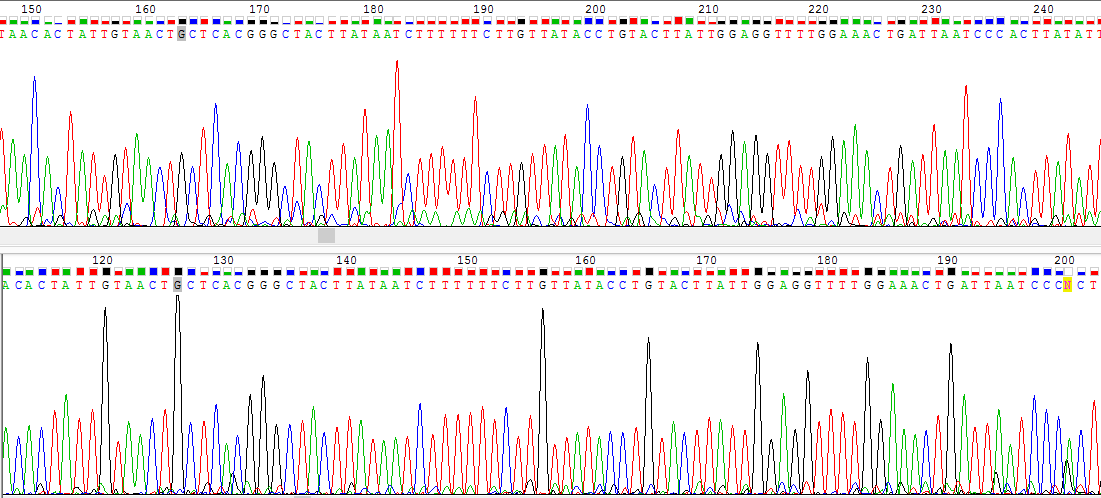
Рис. 7. Изображение хорошо читаемого участка прямой цепи (снизу) и соответствующего участка комплементарной к обратной цепи (сверху).
- Самыми проблемными местами этой цепи можно считать те, которые нельзя проверить ввиду их отсутствия на прямой цепи. На Рис. 8 показаны 3 позиции (18, 24, 27 по обратной цепи),
в которых сигнал слабый, а шум довольно сильный (обозначены N). Первые 2 позиции довольно сомнительны, поэтому я решила оставить там N, тем более, что этого участка нет в прямой
цепи, значит, высока вероятность того, что он не так важен для выполнения задачи секвенирования. В третьей же позиции сигнал С намного выше шумового сигнала Т, поэтому я
изменила N на С.
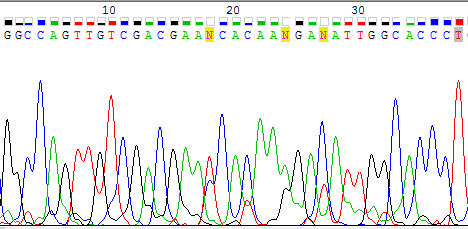
Рис. 8. Изображение проблемного участка комплементарной к обратной цепи; N 27 будет заменен на С.
- Наиболее характерный пример путаницы между сигналом и фоном: в позиции 279 (по прямой цепи) на комплементарной обратной цепи сигнал полностью совпадает с фоном! Однако прямая
цепь позволяет понять, что на месте N должен быть С (Рис. 9).
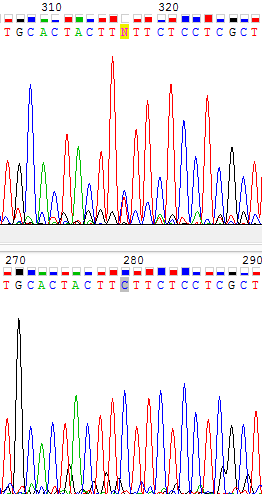
Рис. 9. Изображение проблемного участка комплементарной к обратной цепи (сверху) и соответствующего участка прямой цепи (снизу). N в позиции 279 должен быть заменен на С.
- Остальные трудности сводились к замене N на нуклеотиды, которые можно было однозначно определить по прямой цепи.
Полученные fasta-файлы были использованы для того, чтобы сделать выравнивание отредактированных последовательностей в программе JalView. Выравнивание было легко получено путем сдвига прямой последовательности до момента совпадения с комплементарной обратной, остальные позиции совпали сами. С результатом можно ознакомиться на Рис. 10. Измененные нуклеотиды обозначены строчными буквами; нуклеотиды, между которыми в прямой цепи был удален остаток, отмечены * в строке под выравниванием. Выравнивание раскрашено по нуклеотидам.

Задание 2. Пример нечитаемой хроматограммы.
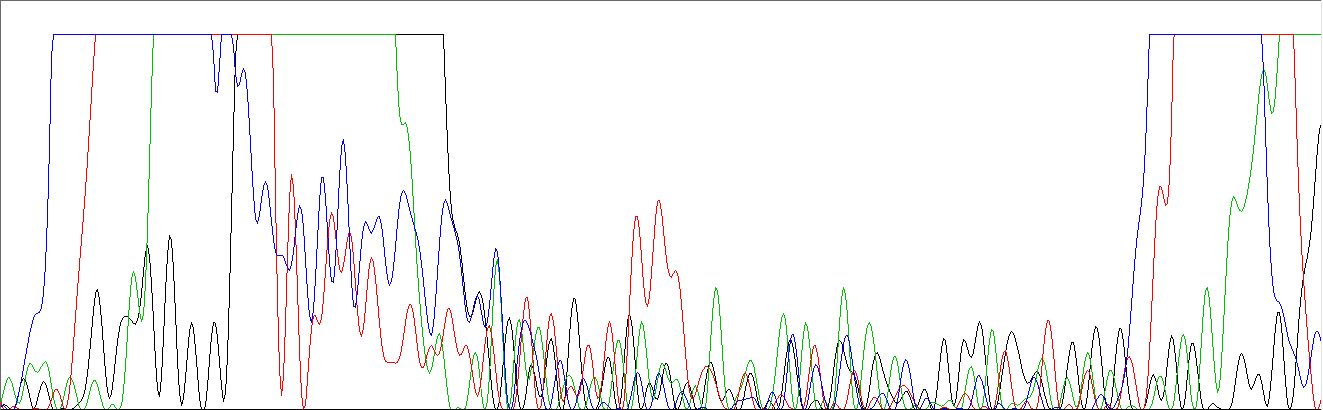
Для иллюстрации нечитаемой хроматограммы я выбрала файл NN_G10.ab1. На Рис. 11 можно видеть участок хроматограммы, на котором присутствуют пятна краски, которые связаны либо с ошибкой секвенатора, либо с загрязнением препарата, а остальная часть последовательности выглядит так, как будто она загрязнена другой ДНК, так как там слишком много пиков на позицию и они наслаиваются друг на друга. Загрязнение может происходить, если праймер для секвенирования отжегся на два разных участка, либо при ПЦР поднялись два фрагмента из исходной ДНК.