EMBOSS: пакет программ для анализа последовательностей
Упражнения
- seqret: собирает несколько файлов в формате fasta в единый файл
Я собрала в файл fasta все записи UniProt, AC которых начинается с Q11 (Рис. 1).
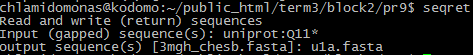
Рис. 1. Команда seqret.
- seqretsplit: разделяет один файл в формате fasta с несколькими последовательностями на отдельные fasta файлы
Я разделила свой файл h4.fasta (c последовательностями белков из практикума 8) на отдельные fasta-файлы с названием последовательностей, в них содержащихся. Команда и результат на Рис. 2.
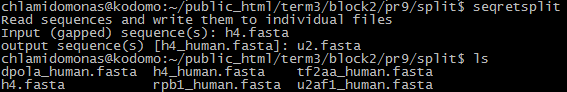
Рис. 2. Команда seqretsplit и ее результат.
- (6) seqret: также позволяет перевести выравнивание из fasta-формата в формат .msf
Для этого задания я взяла файл 1.fasta из практикума 7, который содержит выравнивание лучшей находки с исходной.
Команда на Рис. 3. Результат в файле 1.msf.

Рис. 3. Команда seqret для смены формата выравнивания.
- transeq: позволяет транслировать кодирующие последовательности, лежащие в одном fasta файле, в аминокислотные,
используя указанную таблицу генетического кода.
Я взяла файл, содержащий последовательности гена субъединицы 1 цитохромоксидазы и лучших находок (по результатам blast).
Результат - в одном fasta-файле. Использованная команда на Рис. 4.
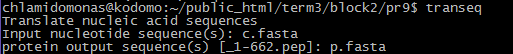
Рис. 4. Команда transeq.
- transeq: транслирует данную нуклеотидную последовательность в шести рамках.
Использовался файл, содержащий консенсусную последовательность из практикума 6 (ген субъединицы 1 цитохромоксидазы у Polycirrus medusa).
Результат трансляции в 6 рамках: fasta-файл. С командой можно ознакомиться на Рис. 5.

Рис. 5. Команда transeq - трансляция последовательности в 6 рамках.
- (11)cusp: находит частоты кодонов в данных кодирующих последовательностях.
Входной файл: ген субъединицы 1 цитохромоксидазы. Результат работы программы: файл. Команда на Рис. 6.

Рис. 6. Команда cusp.
- (12)compseq: находит частоты динуклеотидов (опция -word 2) в данной нуклеотидной последовательности и может сравнить их с ожидаемыми (опция -calcfreq).
Входной файл: ген субъединицы 1 цитохромоксидазы. Результат работы программы: файл. Команда на Рис. 7.

Рис. 7. Команда compseq.
- infoalign: выводит число совпадающих букв между второй последовательностью выравнивания (опция -refseq 2) и всеми остальными
(на выходе только имя последовательности и число: опции -only -name -idcount).
Входной файл: выравнивание 4 последовательностей, кодирующих ген субъединицы 1 цитохромоксидазы. Результат работы программы: файл. Команда на Рис. 8.

Рис. 8. Команда infoalign.
- featcopy: позволяет перевести аннотации особенностей в записи формата .gb в табличный формат .gff.
Входной файл: seq.gb. Результат работы программы: файл. Команда на Рис. 9.

Рис. 9. Команда featcopy.
- shuffle: перемешивает буквы в данной нуклеотидной последовательности.
Входной файл: ген субъединицы 1 цитохромоксидазы. Результат работы программыи команда на Рис. 10.
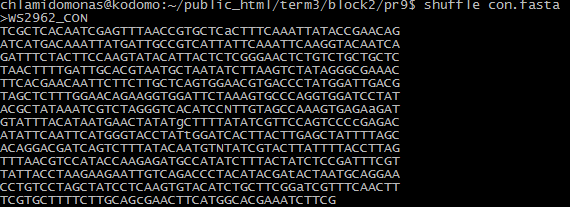
Рис. 10. Команда shuffle и результат ее работы.
Сравнение аннотации генов белков в одной хромосоме бактерии или археи с трансляциями длинных открытых рамок считывания
Для выполнения заданий данного практикума я выбрала свою бактерию из 1-го семестра: Aquifex aeolicus VF5 (Рис. 11).
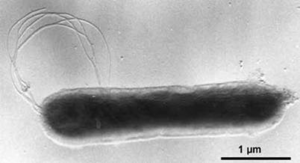
Генетический материал этой бактерии представлен двумя кольцевыми молекулами ДНК: хромосомой (длина 1551335 п.н.) и плазмидой (длина 39456 п.н.). Ссылки на записи NCBI: NC_000918.1 (хромосома), NC_001880.1 (плазмида).
Задания я выполняла для хромосомы Aquifex aeolicus VF5: запись в GenBank (ссылка на файл). Но так как нуклеотидная последовательность хромосомы слишком длинная, в файле .gb содержится только аннотация, для выполнения последующих команд я использовала файл в формате fasta.
Задание 1. Получить список трансляций открытых рамок.
В данном задании требовалось получить трансляции открытых рамок с помощью команды getorf пакета EMBOSS. Данная команда ищет открытые рамки и транслирует последовательности открытых рамок. Требовалось выполнить команду со следующими опциями:
- Минимальная длина открытой рамки - 180 п.н.: -minsize 180
- Таблица генетического кода для данного генома (для бактерии): -table 11
- Кольцевая хромосома: -circular
- Выходные последовательности - трансляции открытых рамок от стоп кодона до стоп кодона: -find 0
Команда представлена на Рис. 12. Результатом является fasta-файл с трансляциями открытых рамок: ссылка.
Далее я получила список координат и ориентаций найденных открытых рамок с помощью infoseq - программы, которая позволяет работать с данными о полученных последовательностях и выбирать нужные. Я запустила команду (Рис. 13) со следующими опциями:
- -name (получим ID открытой рамки)
- -description (в числе прочего - координаты в геноме)
- -sprotein1 - length (длина трансляции в остатках)
Однако результат работы команды дает некоторые избыточные данные, так что потребовалось отфильтровать нужные данные и привести их в читаемый вид в Excel. Таким образом, ID, координаты в геноме и длина трансляции (в остатках) для каждой открытой рамки приведены в файле Excel.
Задание 2. Получить список аннотированных генов белков.
Для выполнения данного задания я скачала файлы NC_000918.faa (аминокислотные последовательности всех белков в формате fasta) и NC_000918.ptt (хромосомная таблица со списком генов белков). Примечание: с последним файлом неудобно работать, поэтому я скачала с сайта NCBI файл со списком генов белков в текстовом формате: ссылка.
Далее потребовалось преобразовать таблицу со списком генов белков в таблицу Excel, оставив только нужные колонки и отсортировав по положению гена на хромосоме (по возрастанию числа в столбце from). Результат: файл Excel.
Задание 3. Сравнить две таблицы.
Для сравнения таблиц я объединила две таблицы в одну, результат - таблица.
При поверхностном взгляде можно заключить:
- Аннотированных белков примерно в 5 раз меньше, чем открытых рамок (1497 против 8110). Это можно объяснить тем, что наличие открытой рамки не гарантирует присутствие гена, кодирующего белок.
- Можно было ожидать, что среди аннотированных белков найдутся те, которые по размеру меньше 60 остатков (то есть их гены короче 180 п.н.) и поэтому не встречаются в открытых рамках (где мы ограничили наименьшую длину рамки), но я не нашла подтверждения своей гипотезы.
- Что касается конкретных различий, то первым делом я заметила, что во всех записях, относящихся к одному гену и совпадающих по длине, аннотированный ген заканчивается
и начинается на 3 нуклеотида позже (на обратной цепи, соответственно, раньше по номеру), чем ген ORF (на Рис. 14а пример для прямой цепи, на Рис. 14b - для обратной).

Рис. 14a. Фрагмент таблицы сравнения: различие в определении границ гена Annotation и ORF (смещение на 3 нуклеотида) (прямая цепь).

Рис. 14b. Фрагмент таблицы сравнения: различие в определении границ гена Annotation и ORF (смещение на 3 нуклеотида) (обратная цепь).
На самом деле, создавшаяся ситуация совсем не очевидна (по сравнению со следующим случаем). Полагаю, что это связано с различием в определении понятия "ген": еще раз повторюсь, что в ORF ген - последовательность от "стопа" до "стопа", а в Annotation - от старта до стопа; и, возможно, в комбинации с альтернативным стартом.
- Следующее различие более понятно: аннотированный ген длиннее на 1 кодон, чем открытая рамка (на Рис. 15а - пример на прямой цепи, на Рис. 15b - на обратной).
Это можно объяснить тем, что в открытую рамку не входит стоп-кодон.

Рис. 15a. Фрагмент таблицы сравнения: различие в определении конца гена Annotation и ORF (ORF короче на 3 нуклеотида) (прямая цепь).

Рис. 15b. Фрагмент таблицы сравнения: различие в определении конца гена Annotation и ORF (ORF короче на 3 нуклеотида) (обратная цепь).
- Большое количество отличий в длине аннотированного гена и открытой рамки связано с наличием у прокариот альтернативных старт-кодонов: GTG, CTG, TTG, ATT.
Из-за того, что реальный ген начинается с нестандартного старт-кодона, открытая рамка принимает за старт ближайший AUG и оказывается длиннее аннотированного гена.
Примеры на прямой и обратной цепях на Рис. 16а-b.

Рис. 16a. Фрагмент таблицы сравнения: различие в определении старта Annotation и ORF (ORF длиннее) (прямая цепь).

Рис. 16b. Фрагмент таблицы сравнения: различие в определении старта Annotation и ORF (ORF длиннее) (обратная цепь).
- Один из самых интересных и самых странных примеров связан с самым началом хромосомы: гену огромного первого белка (700 остатков) не соответствует ни одна из рамок
на прямой цепи, зато с ним перекрываются 4 практически последовательные рамки на обратной цепи (Рис. 17a). К слову, первая рамка на прямой цепи начинается только с
позиции 2105! По всей видимости, getorf не узнал первый старт, а AUG ему либо не попадался, либо нельзя было так разбить последовательность, чтобы все рамки были длиннее
180 нуклеотидов.
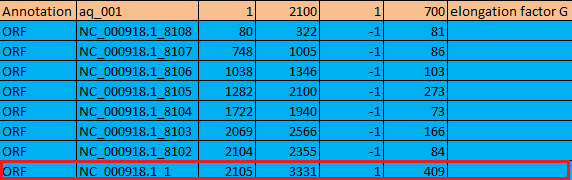
Рис. 17а. Фрагмент таблицы сравнения: гену не соответствует ни одна ORF на прямой цепи, но с ним перекрываются 4 рамки на обратной цепи.
На Рис. 17b представлен пример следующего явления: аннотированному гену соответствуют не только наиболее похожая открытая рамка, но и еще одна рамка, перекрывающаяся с первой (точнее, полностью входящая в нее). Причиной этого может быть распознавание программой дополнительных старта и стопа внутри гена.

Рис. 17b. Фрагмент таблицы сравнения: гену соответствует не только более длинная ORF, но и более короткая рамка на прямой цепи.
- Еще одно интересное различие: записи Annotation и ORF, относящиеся к одному гену, "разделены" перекрывающейся
рамкой с обратной цепи (Рис. 18). Оно могло произойти просто в результате того, что задача программы getorf - разбить геном на рамки, не учитывая наличия у прокариот
таких "отклонений", как перекрывание генов или, к примеру, присутствия интронов (хотя и редко). Видимо, в данном промежутке обратной цепи просто встретились кодоны, распознанные
как старт и стоп.

Рис. 18. Фрагмент таблицы сравнения: гену соответствует не только более длинная ORF, но и некая рамка на обратной цепи.
- Следующий пример аналогичен предыдущему, но там присутствует перекрывание между антипараллельными рамками и между рамкой на обратной цепи и геном, длина которого более 150
п. н. Предполагаемая причина, как и выше, - неточности в работе программы (Рис. 19а).

Рис. 19a. Фрагмент таблицы сравнения: гену соответствует не только более длинная ORF, но и некая рамка на обратной цепи (пример перекрывания).
И еще один пример из этой серии (Рис. 19b), но в данном случае с открытой рамкой на прямой цепи перекрываются 2 рамки на обратной цепи, которые, в свою очередь, перекрываются между собой, причем размер перкрытия около 500 нуклеотидов.
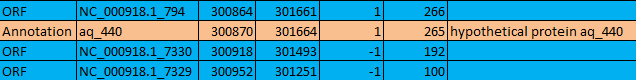
Рис. 19b. Фрагмент таблицы сравнения: гену соответствует не только более длинная ORF, но и некая рамка на обратной цепи (пример перекрывания).