Работа с PyMOL
Познакомимся с возможностями PyMOL как пакета моделирования на примере работы с молекулой лизоцима (PDB ID: 1LMP).
import __main__
__main__.pymol_argv = [ 'pymol', '-x' ]
import pymol
pymol.finish_launching()
from pymol import cmd,stored
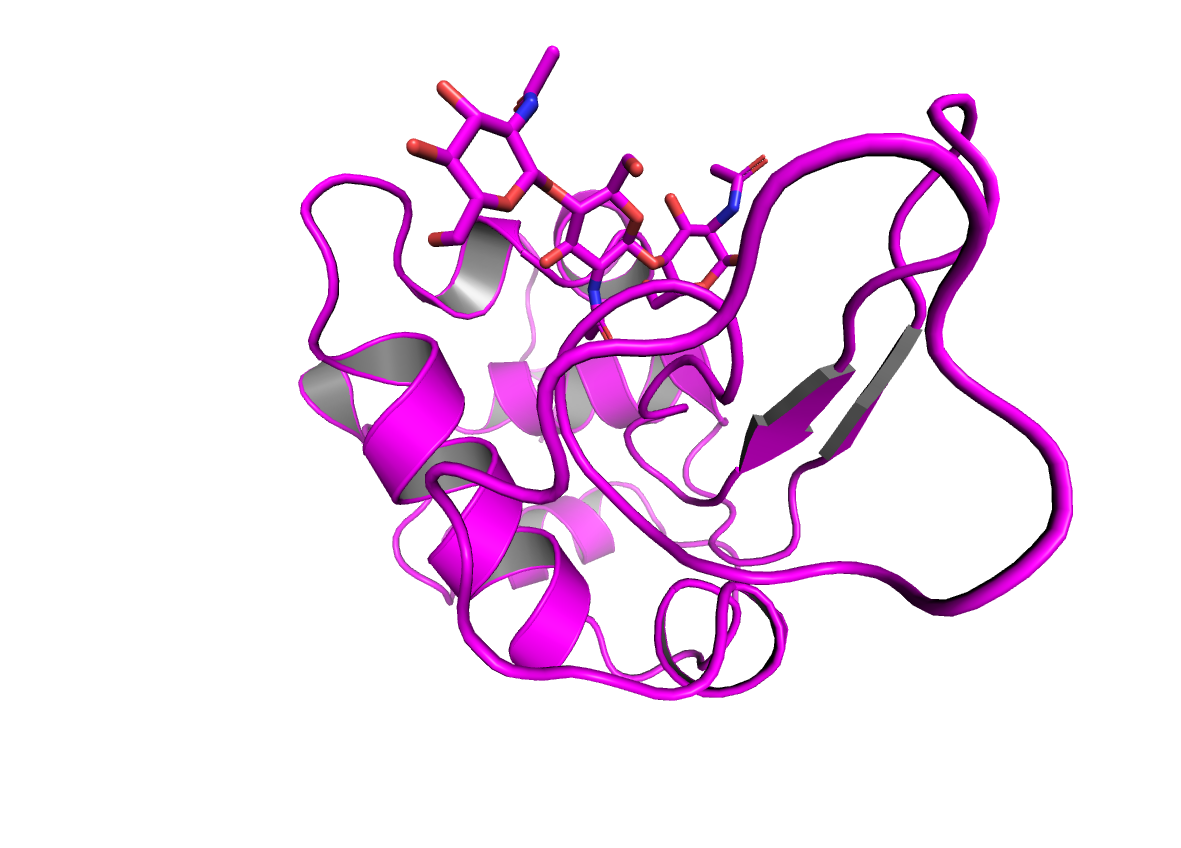
Изобразим структуру белка с лигандом (Рис. 1а).
cmd.fetch("1lmp")
cmd.remove("solvent")
cmd.bg_color("white")
cmd.color("magenta")
cmd.do('''
util.cnc hetatm
set ray_shadows, off
set cartoon_highlight_color, grey50
set field_of_view, 70''')
cmd.cartoon("dumbbell", "ss h")
cmd.zoom()
cmd.png(filename='no_sculpt_1.png',width='10cm',dpi=300,ray=3)
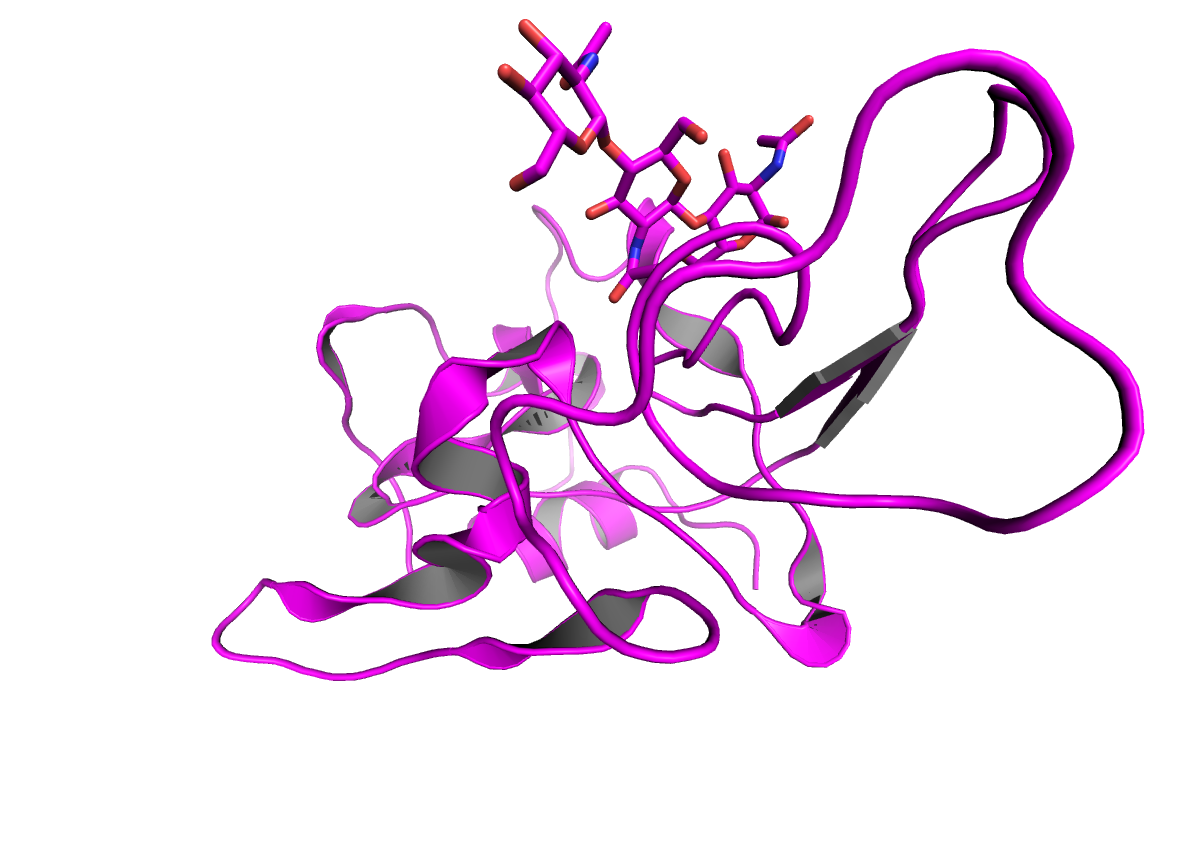
А затем изменим ее с помощью Wizard->Demo->Sculpting (Рис. 1b).
cmd.zoom()
cmd.png(filename='sculpt_2.png',width='10cm',dpi=300,ray=3)
 |
 |
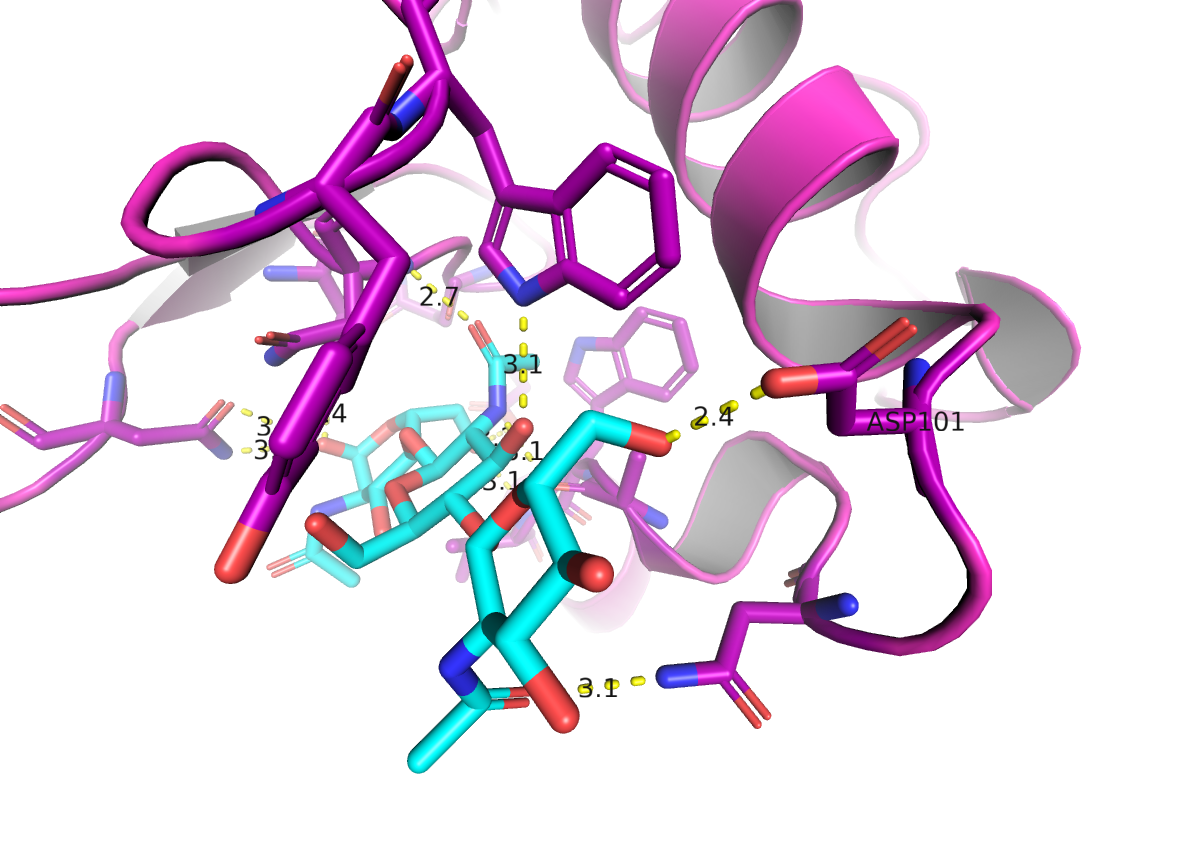
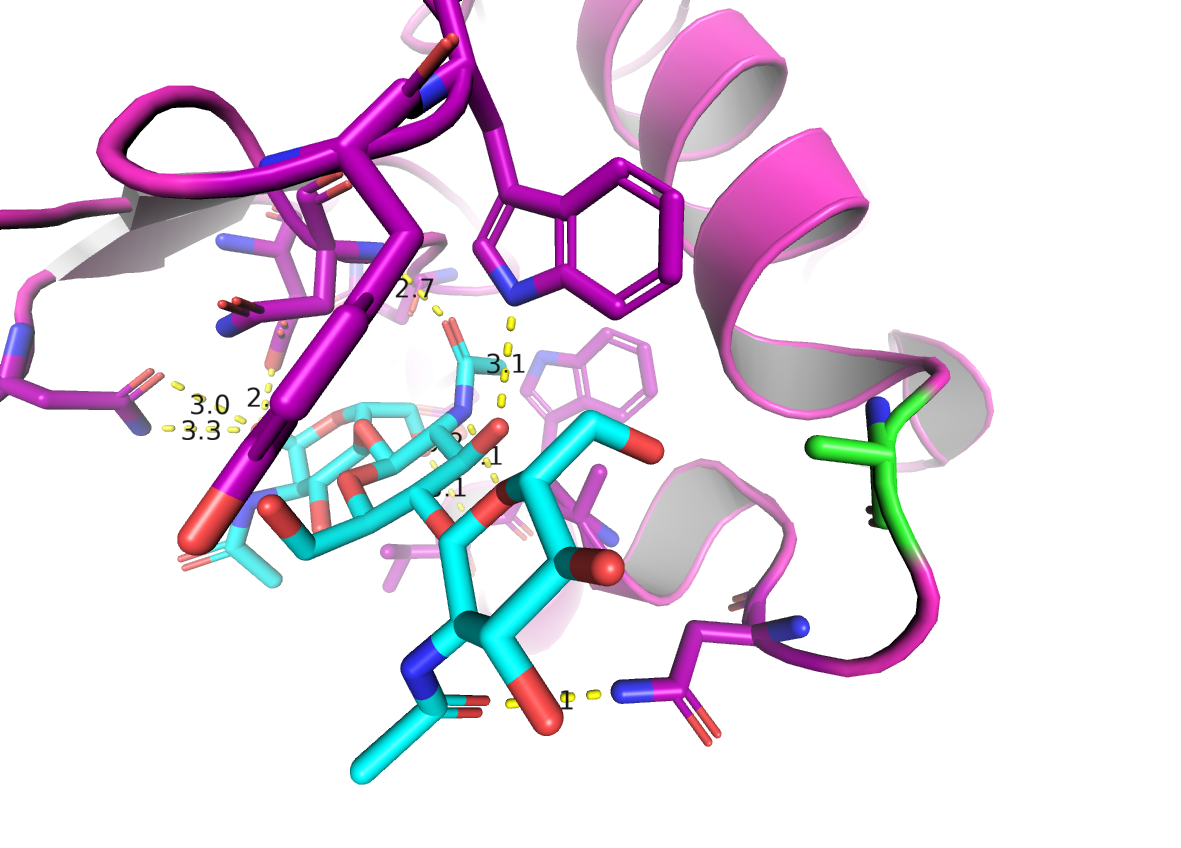
Теперь найдем подходящий остаток для мутагенеза. Для этого покажем остатки, которые взаимодействуют с лигандами водородными связями.
cmd.reinitialize()
cmd.fetch("1lmp")
cmd.remove("solvent")
cmd.bg_color("white")
cmd.color("lightmagenta")
cmd.do('''
util.cbac hetatm
set ray_shadows, off
set cartoon_highlight_color, grey50
set field_of_view, 70
select bindsite, byres m. 1lmp and (hetatm around 3.5)
show sticks, bindsite
util.cbap bindsite
''')
cmd.cartoon("dumbbell", "ss h")
cmd.orient("hetatm")
cmd.zoom("hetatm", "10")
cmd.dist("hydrbonds", "m. 1lmp and (not hetatm)", "hetatm", "3.5", "2")
cmd.do('''
label bindsite and n. ca and resi 101, "%s%s" % (resn, resi)
set label_color, black
''')
view = cmd.get_view()
cmd.set_view(view)
cmd.png(filename='bsite1.png',width='10cm',dpi=300,ray=3)
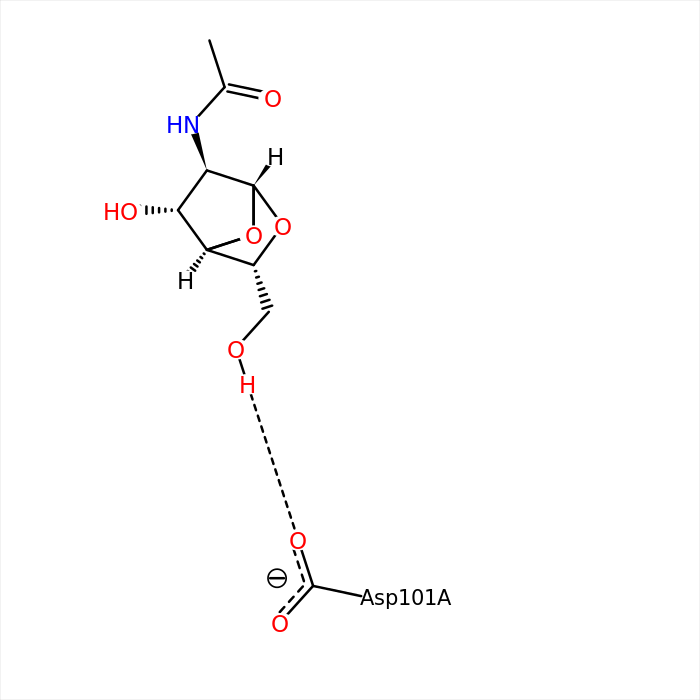
Думаю, что замена остатка ASP101 на аланин приведет к потере водородной связи. На сайте PDB тоже говорится, что этот остаток участвует в связывании лиганда.
 |
 |
Проведем мутацию.
cmd.hide("labels")
cmd.delete("hydrbonds")
from pymol import wizard
cmd.wizard("mutagenesis")
cmd.do("refresh_wizard")
cmd.get_wizard().set_mode("ALA")
cmd.get_wizard().do_select("resi 101")
# Select the first rotamer, which is most probable
cmd.frame(1)
# Apply the mutation
cmd.get_wizard().apply()
# Close wizard
cmd.set_wizard("done")
cmd.do('''
util.cbag resi 101
label n. ca and resi 101, "%s%s" % (resn, resi)
''')
cmd.dist("hydrbonds2", "m. 1lmp and (not hetatm)", "hetatm", "3.5", "2")
view2 = cmd.get_view()
cmd.set_view(view2)
cmd.png(filename='mutsite1.png',width='10cm',dpi=300,ray=3)
Водородная связь пропала.
 |
 |
Теперь совместим это со структурой исходного белка без мутации и сделаем про это фильм.
cmd.delete("hydrbonds2")
cmd.hide("sticks", "not resi 101")
cmd.fetch("1lmp", "nomut")
cmd.remove("solvent")
cmd.cartoon("dumbbell", "ss h")
cmd.color("silver", "nomut")
cmd.do('''
util.cbac hetatm
show sticks, resi 101
util.cnc nomut and resi 101
''')
cmd.do('''
label resi 101, "%s%s" % (resn, resi)
''')
cmd.hide("labels")
cmd.do('''
set matrix_mode, 1
set movie_panel, 1
set cache_frames, 1
mset 1-200
frame 1
zoom nomut, -1
translate [10,0,0], object=1lmp
mview store
mview store, object=nomut
mview store, object=1lmp
frame 20
zoom nomut, -1
translate [-5,0,0], object=1lmp
mview store
mview store, object=nomut
mview store, object=1lmp
frame 40
super 1lmp, nomut
mview store
mview store, object=nomut
mview store, object=1lmp
mview interpolate, object=1lmp
frame 80
orient hetatm
mview store
mview store, object=nomut
mview store, object=1lmp
frame 100
orient hetatm
mview store
mview store, object=nomut
mview store, object=1lmp
frame 130
orient resi 101
mview store
mview store, object=nomut
mview store, object=1lmp
frame 140
orient resi 101
label resi 101 and 1lmp and n. ca, "%s%s" % (resn, resi)
mview store
mview store, object=nomut
mview store, object=1lmp
frame 160
orient resi 101
label resi 101 and nomut and n. cb, "%s%s" % (resn, resi)
mview store
mview store, object=nomut
mview store, object=1lmp
frame 165
orient resi 101
mview store
mview store, object=nomut
mview store, object=1lmp
frame 190
mview reinterpolate, object=nomut
mview reinterpolate, object=1lmp
frame 1
mplay
''')
import io
import base64
from IPython.display import HTML
video = io.open('ugly_movie1.mp4', 'r+b').read()
encoded = base64.b64encode(video)
HTML(data='''<video alt="test" controls>
<source src="data:video/mp4;base64,{0}" type="video/mp4" />
</video>'''.format(encoded.decode('ascii')))
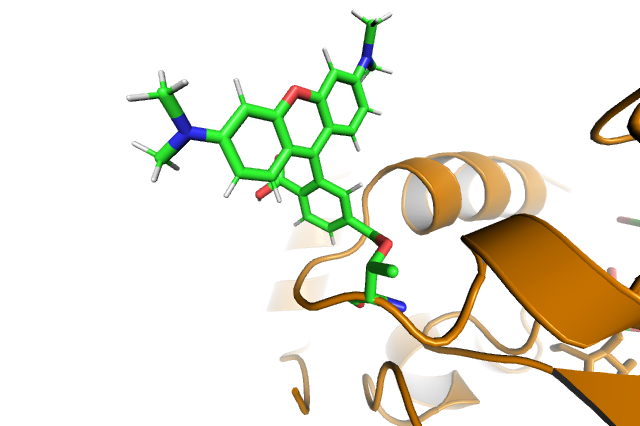
Теперь присоединим к -ОН группе белка через сложноэфирную связь метку TAMRA. PyMOL RIP у меня на виртуальной машине, поэтому ниже буду записывать скрипты и визуализировать их в PyMOL локально.
%%writefile tamra.pml
cmd.load("tamra.sdf")
cmd.remove('tamra and id 4')
cmd.select('o', 'id 32')
cmd.do('''
util.cbag tamra
''')
cmd.fetch("1lmp")
cmd.remove("solvent")
cmd.color("tellurium", "1lmp")
cmd.do('''
set ray_shadows, off
set cartoon_highlight_color, grey50
set field_of_view, 70
''')
cmd.cartoon("dumbbell", "ss h")
cmd.zoom()
cmd.do('''
select thr, resn thr and resi 85
show sticks, thr
util.cbag thr
select og1, thr and name OG1
''')
cmd.fuse('o', 'og1', 1)
cmd.torsion(-180.0)
set_view (\
-0.123622522, -0.752091646, 0.647359014,\
0.778710246, -0.477876723, -0.406487346,\
0.615074396, 0.453855574, 0.644740224,\
0.000000000, 0.000000000, -16.995183945,\
15.268630981, 36.908187866, 20.766571045,\
5.474930286, 28.515300751, -70.000000000 )
cmd.unpick()
Построим полиаланиновую альфа-спираль длиной 100 аминокислот. Средние значения торсионных углов для полиаланиновой альфа-спирали нашла здесь.
with open("polyala.pml", 'w') as polyala:
phi = -63.1
psi = -41.2
polyala.write("cmd.fragment('ala')\n")
for i in range (2,101):
polyala.write("cmd.edit(\"i. " + str(i) + " & n. C\")\n")
polyala.write("cmd.do('editor.attach_amino_acid(\"pk1\", \"ala\")')\n")
polyala.write("cmd.set_dihedral(\"i. "+str(i)+" & n. N\", \"i. "+str(i)+" & n. CA\", \"i. "+str(i)+" & n. C\", \"i. "+str(i+1)+" & n. N\", "+str(phi)+")\n")
polyala.write("cmd.set_dihedral(\"i. "+str(i)+" & n. C\", \"i. "+str(i+1)+" & n. N\", \"i. "+str(i+1)+" & n. CA\", \"i. "+str(i+1)+" & n. C\", "+str(psi)+")\n")
polyala.write("cmd.zoom()\n")
polyala.write("cmd.unpick()\n")
polyala.write("cmd.png(filename='polyala.png',width='10cm',dpi=300,ray=3)")
