Докинг низкомолекулярных лигандов в структуру белка
В этом задании будем осуществлять докинг в структуру, модель которой делали в прошлом практикуме.
In [3]:
import numpy as np
import copy
# Отображение структур
import IPython.display
import ipywidgets
from IPython.display import display,display_svg,SVG,Image
# Open Drug Discovery Toolkit
import oddt
import oddt.docking
import oddt.interactions
# Органика
from rdkit.Chem import Draw
from rdkit.Chem.Draw import IPythonConsole
import pmx # Модуль для манипулирования pdb
Для работы возьмем из прошлого практикума по гомологичному моделированию структуру с наибольшим скором.
In [4]:
pdb=pmx.Model('MERLU.B99990002.pdb')
for r in pdb.residues[120:]:
print r #посмотрим остатки, чтобы найти лиганд
In [5]:
# разделяем на отдельные объекты белок и лиганд
newpdb = pdb.copy()
for r in newpdb.residues[-3:]:
newpdb.remove_residue(r)
lig = pdb.copy()
del lig.residues[:-3]
In [6]:
# ищем геометрический центр лиганда
x = []
y = []
z = []
for a in lig.atoms:
coord = a.x
x.append(coord[0])
y.append(coord[1])
z.append(coord[2])
geom_center = [np.mean(x), np.mean(y), np.mean(z)]
geom_center
Out[6]:
In [7]:
newpdb.writePDB("myprot.pdb")
lig.writePDB("lig.pdb")
Подготовка белка для докинга
In [10]:
prot = oddt.toolkit.readfile('pdb','myprot.pdb').next()
prot.OBMol.AddPolarHydrogens()
prot.OBMol.AutomaticPartialCharge()
print 'is it the first mol in 1lmp is protein?',prot.protein,':) and MW of this mol is:', prot.molwt
Странно, но белок не распознается как белок.
Лиганды для докинга
In [11]:
smiles = ['[NH3+]C(=O)NC1C(C(C(OC1O)CO)O)O', 'C(=O)NC1C(C(C(OC1O)CO)O)O',
'[OH]C(=O)NC1C(C(C(OC1O)CO)O)O', 'C1=CC=C(C=C1)C(=O)NC1C(C(C(OC1O)CO)O)O',
'[O-]C(=O)C(=O)NC1C(C(C(OC1O)CO)O)O']
mols= []
images =[]
for s in smiles:
m = oddt.toolkit.readstring('smi', s)
if not m.OBMol.Has3D():
m.make3D(forcefield='mmff94', steps=150)
m.removeh()
m.OBMol.AddPolarHydrogens()
mols.append(m)
###with print m.OBMol.Has3D() was found that:
### deep copy needed to keep 3D , write svg make mols flat
images.append((SVG(copy.deepcopy(m).write('svg'))))
display_svg(*images)
Докинг
In [12]:
#create docking object
#в качестве центра укажем геометрический центр лиганда"
dock_obj= oddt.docking.AutodockVina.autodock_vina(
protein=prot,size=(20,20,20),center=geom_center,
executable='/usr/bin/vina',autocleanup=True, num_modes=20)
print dock_obj.tmp_dir
print " ".join(dock_obj.params)
В выдаче указаны координаты геометрического центра, размер объекта и технические характеристики.
In [13]:
# do it
res = dock_obj.dock(mols,prot)
Результаты докинга
In [16]:
import pandas as pd
In [35]:
hbs_total = []
hbs_strict = []
stack = []
phob = []
formulas = []
aff = []
rmsd = []
for i,r in enumerate(res):
hbs_total.append(len(oddt.interactions.hbonds(prot,r)[0]))
hbs_strict.append(oddt.interactions.hbonds(prot,r)[2].sum())
stack.append(len(oddt.interactions.pi_stacking(prot,r)[0]))
phob.append(len(oddt.interactions.hydrophobic_contacts(prot,r)[0]))
formulas.append(r.formula)
aff.append(r.data['vina_affinity'])
rmsd.append(r.data['vina_rmsd_ub'])
restable = pd.DataFrame({"Ligand_formulae": formulas, "Affinity": aff, "RMSD": rmsd, "H-bonds (total)": hbs_total,
"H-bonds (strict)": hbs_strict, "Stacking": stack, "Hydrophobic_contacts": phob})
In [39]:
restable.sort_values(by = ['Affinity'], ascending=False)
Out[39]:
Топ-5 находок принадлежат лиганду с фенильным радикалом. Помимо него в десятку лучших попало карбокси-производное NAG. Интересно, что две лучшие находки добиваются хорошей аффинности разными способами: первая - в большей степени за счет гидрофобных взаимодействий, а вторая - водородных связей и стекинга. Посмотрим на них поближе.
In [40]:
for i in (27, 28):
res[i].write(filename='%s.pdb' % str(i), format='pdb')
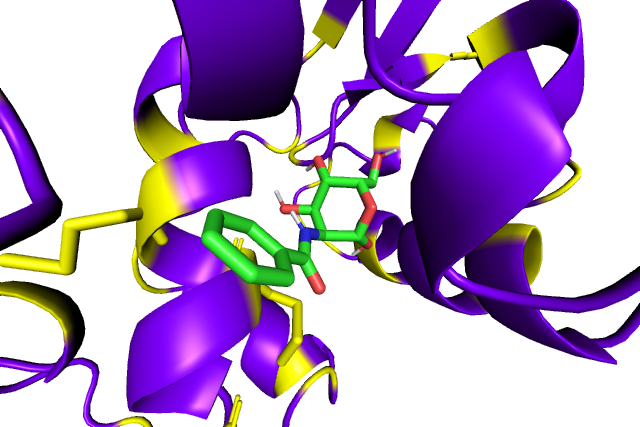 |
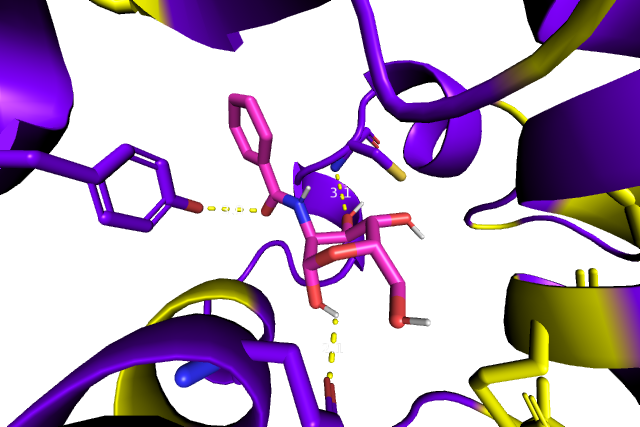 |
| Рис. 1a. В этом взаимодействии не удалось найти ни одной из 6 заявленных водородных связей. Гидрофобные остатки (желтые) слишком далеко от лиганда, чтобы образовывать гидрофобные контакты. | Рис. 1b. Нашлось 3 из 9 водородных связей, одна из них с тирозином, который и, может быть, образует стэкинг с лигандом. |