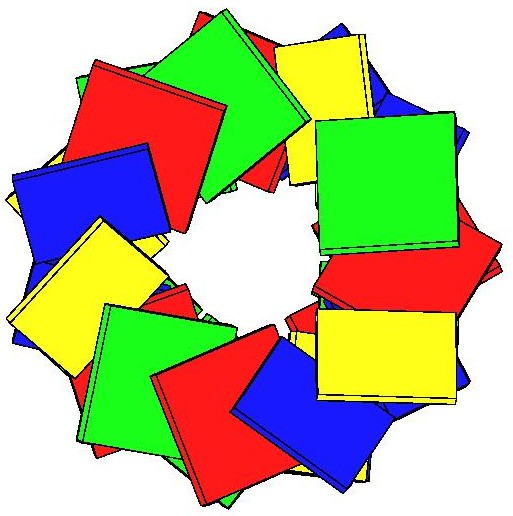
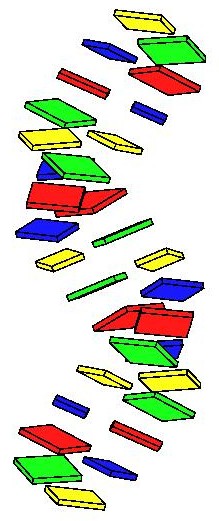
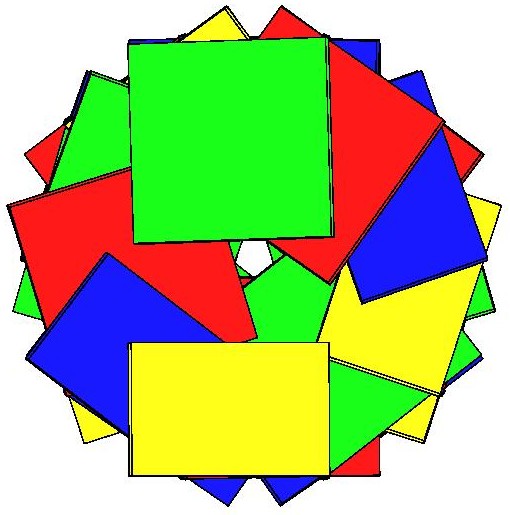
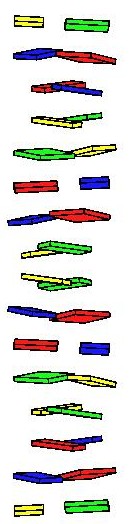
На главную страницу третьего семестра A and B DNA
В первом задании нужно было пострить 2-цепочечную молекулу A и В
ДНК, одна из цепей которой представляет собой четырежды повторяющеюся последовательность gatc. Это нужно было сделать
с помощью команды fiber пакета 3DNA:
fiber -a gatc_a.pdb для А формы ДНК
fiber -a gatc_b.pdb соответсвенно для В формы.
Далее при помощи программы RasMol были исследованы полученные 2 файла, а также файл dna36.pdb.
| A-форма | B-форма | Файл dna36.pdb | |
| Тип спирали (правая или левая) | правая | левая | правая |
| Шаг спирали (Å) | 28.02 | 33,74 | 36,43 |
| Число оснований на виток | 11 | 10 | 9 |
| Ширина большой бороздки (Å) | 7,98(С24) | 17,20(G21) | 11,15(T8) |
| Ширина малой бороздки (Å) | 16,80(T23) | 11,69(T11) | 16,36(G6) |
Эти 3 файла были анализированы другими программами из пакета 3DNA:
find_pair -t gatc_a.pdb stdout | analyze
В результате были получены 3 файла: gatc_a.out, gatc_b.out,
dna36.out. В них, помимо всего прочего, содержится информация о результатах сравнения
торсионных углов.
| Альфа | Бета | Гамма | Дельта | Эпсилон | Зета | Чи | |
| gatc_a.pdb (для каждого нуклеотида каждой цепи почти одиноакого) | - 51.7 | 174.8 | 41.7 | 79.1(0) | - 147.8(-147.7) | - 75.1(-75.0) | -157.2 |
| gatc_b.pdb (для каждого нуклеотида каждой цепи почти одиноакого) | - 29.9 | 136.4(136.3) | 31.1(31.2) | 143.3(143.4) | -140.8 | -160.5 | -98.0(-97.9) |
Strand I base alpha beta gamma delta epsilon zeta chi 1 C --- --- 170.0 98.1 -164.1 -77.2 -152.7 2 C -70.0 177.0 56.9 148.5 -99.9 165.2 -83.7 3 A -74.7 145.9 52.1 143.0 173.4 -87.1 -82.3 4 A -70.1 177.7 47.5 123.7 173.6 -96.9 -96.8 5 C -65.2 -179.7 49.2 144.8 -114.2 -179.1 -95.6 6 G -75.5 153.4 43.4 130.4 168.8 -88.7 -105.3 7 T -64.4 170.5 65.3 125.6 -163.6 -85.3 -129.6 8 T -81.9 -173.3 45.5 134.3 -100.3 167.3 -88.1 9 G -75.6 152.2 48.3 145.6 -172.8 -87.9 -76.3 10 G -69.4 159.1 52.6 96.2 --- --- -107.4 Strand II base alpha beta gamma delta epsilon zeta chi 1 G -69.4 159.1 52.6 96.2 --- --- -107.4 2 G -75.6 152.2 48.3 145.6 -172.8 -87.9 -76.3 3 T -81.9 -173.3 45.5 134.3 -100.3 167.3 -88.1 4 T -64.4 170.5 65.3 125.6 -163.6 -85.3 -129.6 5 G -75.5 153.4 43.4 130.4 168.8 -88.7 -105.3 6 C -65.2 -179.7 49.2 144.8 -114.2 -179.1 -95.6 7 A -70.1 177.7 47.5 123.7 173.6 -96.9 -96.8 8 A -74.7 145.9 52.1 143.0 173.4 -87.1 -82.3 9 C -70.0 177.0 56.9 148.5 -99.9 165.2 -83.7 10 C --- --- 170.0 98.1 -164.1 -77.2 -152.7
Далее эти ДНК были представленны в виде стопочных моделей при помощи следующих комманд:
rotate_mol -b gatc_a.pdb gatc_a_side.pdb
rotate_mol -b gatc_b.pdb gatc-b_side.pdb
rotate_mol -b dna36.pdb dna36_side.pdb
pdb2img -c -i gatc_a.pdb gatc_a.ps
pdb2img -c -i gatc_a_side.pdb gatc_a_side.ps
pdb2img -c -i gatc_b.pdb gatc_b.ps
pdb2img -c -i gatc_b_side.pdb gatc_b_side.ps
pdb2img -c -i dna36.pdb dna36.ps
pdb2img -c -i dna36_side.pdb dna36_side.ps
Полученные результаты:
| А-ДНК |  |
 |
| В-ДНК |  |
 |
| DNA36 |  |
 |