На главную страницу третьего семестра
tRNA structure
Структура тРНК.
В PDB банке, на сайте http://www.rcsb.org был найден
файл 1ml5.pdb, содержащий информацию о структуре рибосомальной терминационном
комплексе с освобождающим фактором 2 в организме E. coli. В нем приведены
структры рибосомальны белков и РНК а ткаже фенилаланиновая тРНК (цепь: В, ID: 4)
Последовательность фенилаланиновой тРНК:
G C G G A U U U A 2MG C U C
A G H2U H2U G G G A G A G C M2G
C C A G A OMC U OMG A A YG A PSU
5MC U G G A G 7MG U C 5MC U G U
G 5MU PSU C G 1MA U C C A C A G
A A U U C G C A C C A
Имеется только одно модифицированное основание: YG - вибутозин (wybotosine). Нумерация
атомов идет с 1884 по 3535, нет ни пропусков, ни вставок.
В данной структре всего 3 спирали. Первая состоит из нуклеотидов (в скобках
комплементарные позиции) с 2 по 7 (71-66) и с 49 по 55 (65-61, 58, 18)
позиции, соответсвенно имеетдлину 13 нуклеотидов. Вторая спираль - с 38 по 44 (32-26)
и с 10 по 15 (25-22, 8, 48) позиции, следовательноее длина также 13 нуклеотдов.
Далее 19 атом (56) возможно не входит в состав спирали. Последняя спираль
состоит всего из 3 нуклеотидов с позциями 34-36 (13-11 цепи С).
Разметка спиралей цветом (красным - первая спираль, зеленым- вторая спираль, голубым - третья спираль):
G C G G A U U U A 2MG C U C
A G H2U H2U G G G A G A G C M2G
C C A G A OMC U OMG A A YG A PSU
5MC U G G A G 7MG U C 5MC U G U
G 5MU PSU C G 1MA U C C A C A G
A A U U C G C A C C A
Затем, при помощи программы RasMol была получена 3D модель этой тРНК. На рисунке отмечены цветами (аналогичными предыдущему заданию) спирали. Ниже приводятся использованные команды.

select all
backbone 60
wireframe off
restrict *B
define helix1 2-7:B, 66-71:B, 49-55:B 58:B, 18:B, 61-65:B
select helix1
color red
define helix2 8:B, 10-15:B, 22-32:B, 38-44:B, 48:B
select helix2
color green
define helix3 34-36:B
select helix3
color blue
select atomno=3514
label 76
select atomno=1885
label 1
Для проверки была использованна программа einverted. Эта программа ищет потенциальнно двуспиральные участки, делает вырвнивание. Некоторые результаты были получены при устоновлении
минимального порогового веса равным 24:
rna: Score 24: 8/8 (100%) matches, 0 gaps
2 atcccccg 9
||||||||
123 tagggggc 116
Далее следовало получить предсказание вторичной структуры этой тРНК при помощи программы mfold:
mfold SEQ=1ml5.fasta P=10
Я указала параметр P равный 10 - процент, того на сколько процентов может отличаться выдаваемое предсказание структуры по своей вычисленной энергии от оптимального, поскольку при этом
значении в результатах появилось нечто отдаленно напоминающее реальность:
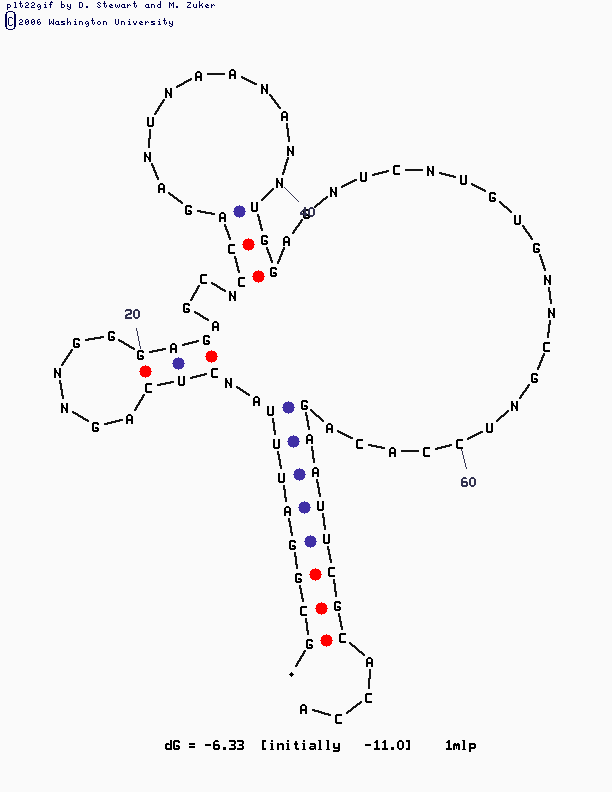
Основной минус - отсуствие 4ой шпильки, но лучщих результатов не было. Такая ошибка возникла из-за того что все модифицированные основания
в исходном FASTA файле были заменены на N и, как следствие, многие связи пропали.
©Попенко Анна
