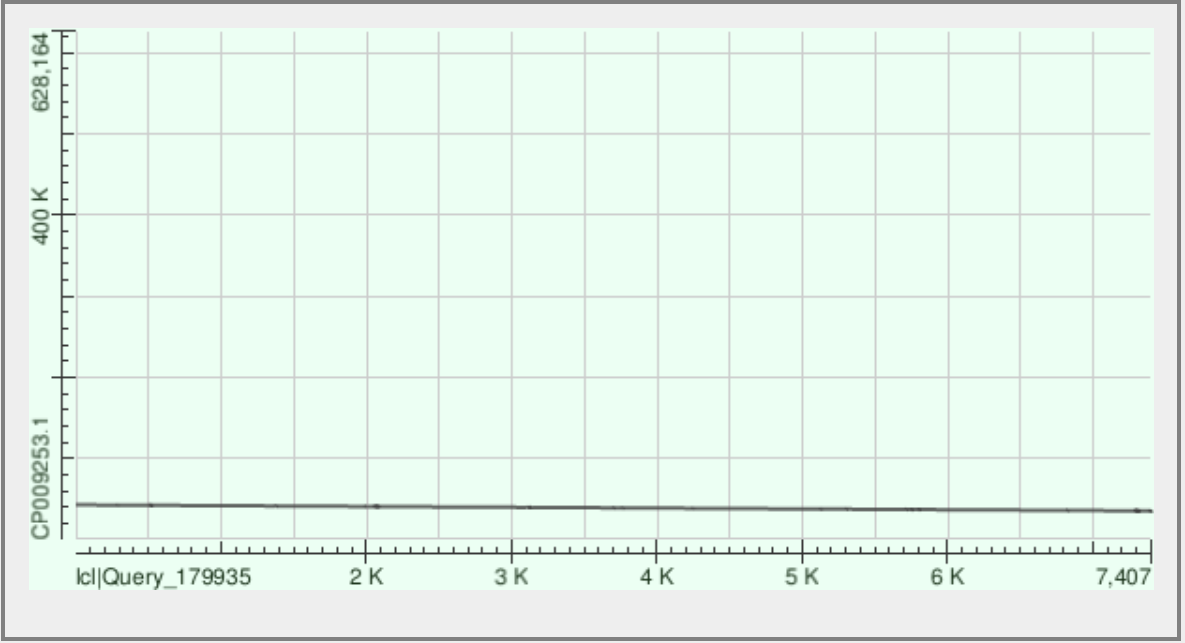
Рис.1. Контиг ID 78 |
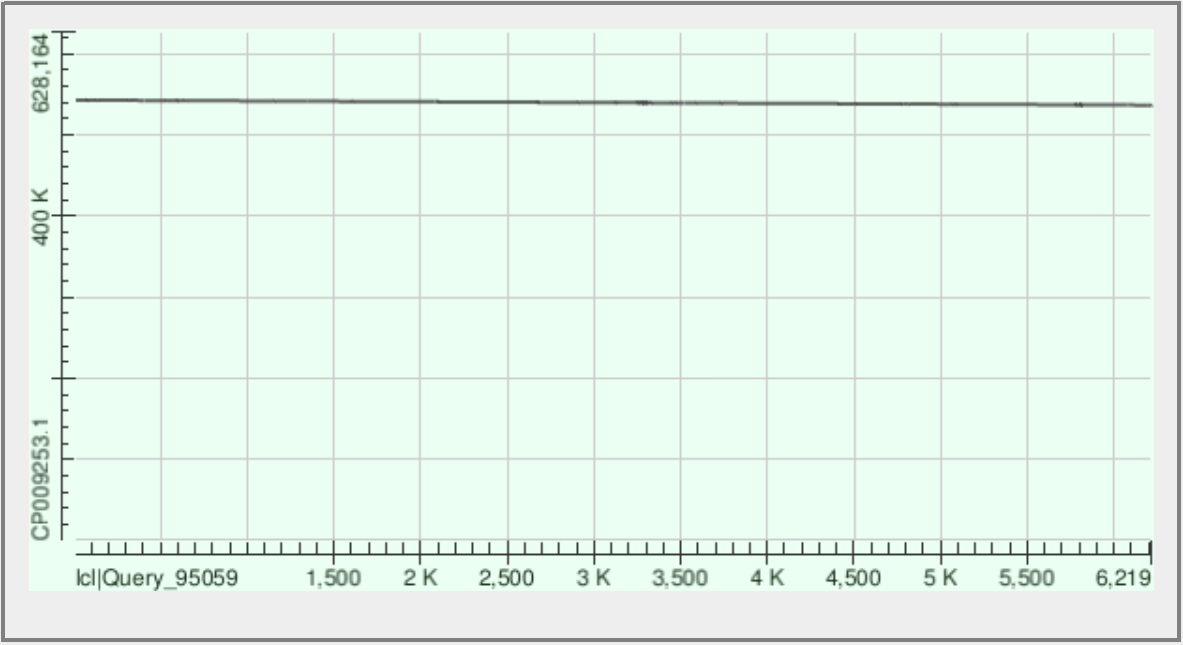
Рис.2. Контиг ID 26 |
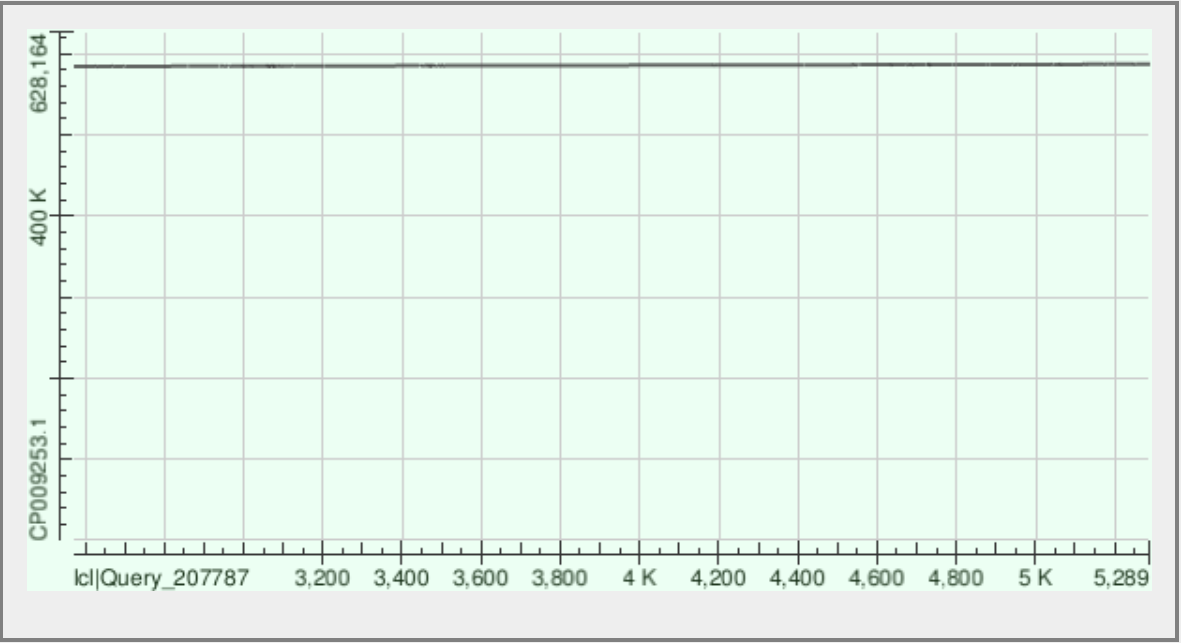
Рис.3. Контиг ID 21 |
Для начала мною был создан файл, в котором содержатся все адаптеры:
cat *.fa > adapters.fasta
Адаптеры удалялись с помощью команды:
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240387.fastq 2.fastq ILLUMINACLIP:adapters.fasta:2:7:7
| До чистки | После чистки | |
| Размер файла | 1613 M | 1612 M |
| Число чтений | 15032810 | 15029657 |
Далее были удалены чтения длины менее 30 и нуклеотиды плохого качества с конца чтений:
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 2.fastq 3.fastq TRAILING:20 MINLEN:30
| До чистки | После чистки | |
| Размер файла | 1612 M | 444 M |
| Число чтений | 15029657 | 4437416 |
29-mers были получены командой:
velveth 29mers 29 -fastq -short 3.fastq
Контиги были собраны командой:
velvetg 29mers/
| K-mers 29 | |
| N50 | 1374 |
| Длина | 633551 |
| Количество контигов | 1152 |
| Самые длинные контиги | 1)ID 78, lgth 7399, cov 33.421679 2)ID 26, lgth 6230, cov 25.778652 3)ID 21, lgth 5261, cov 23.969397 |
Встречаются контиги с аномальным покрытием, например, контиг ID 918 c покрытием 328.390909 и длиной 110.
Или также контиги ID 974 c покрытием 195.039474 и длиной 76 и ID 387 c покрытием 344.965517 и длиной 29.

Рис.1. Контиг ID 78 |

Рис.2. Контиг ID 26 |

Рис.3. Контиг ID 21 |
| Контиг | Mismatches | Gaps | Chromosome | Query coverage | Identity |
| 78 | 1219 | 66 | 35162-42578 | 99% | 84% |
| 26 | 1248 | 111 | 536550-542787 | 99% | 80% |
| 21 | 677 | 108 | 584329-587054 | 51% | 76% |
Все три контига ложатся на геном без существенных разрывов и в одном месте.
© Макиевская Кьяра, 2018