Задание 1.
В этом задании с помощью программы fiber пакета 3DNA необходимо было получить 3 структуры - A-, B- и Z- формы ДНК.
Ссылки на файлы: А-форма, B-форма, Z-форма.
Задание 2.
Упр.1.
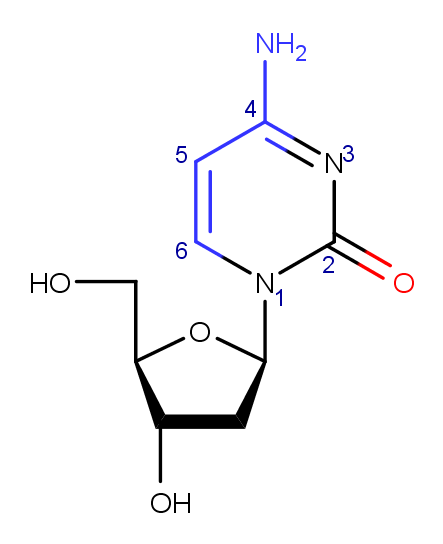 Рисунок 1. Цитозин |
Для выполнения последующих заданий мною была выбрана полученная в 1 задании A-форма ДНК. Для выбранного мной цитозина необходимо было определить, какие атомы ориентированы в сторону большой бороздки, а какие - в сторону малой. На рисунке слева представлена структура цитозина с покрашенными в красный атомами, смотрящими в сторону большой бороздки и покращенными в синий атомами, смотрящими в сторону малой бороздки. Cписок атомов по номерам:
В сторону большой бороздки обращен атом: O2.
В сторону малой бороздки обращены атомы: N4, C4, C5, C6.
|
Упр.2.
Ниже представлена таблица сравнения параметров разных форм ДНК.
| А-форма | B-форма | Z-форма |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (А) | 28,03 | 33,75 | 43,5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 7,98[7:A.P-28:B.P] | 17,21[6:A.P-32:B.P] | 16,08[12:A.P-28:B.P] |
| Ширина малой бороздки | 16,81[17:A.P-26:B.P] | 11,69 [40:B.P-5:A.P] | 9,87[20:A.P-25:B.P] |
Ниже представлены изображения описанных форм ДНК
 | 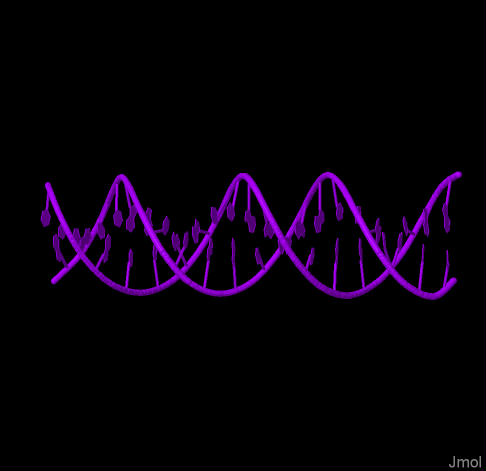 | 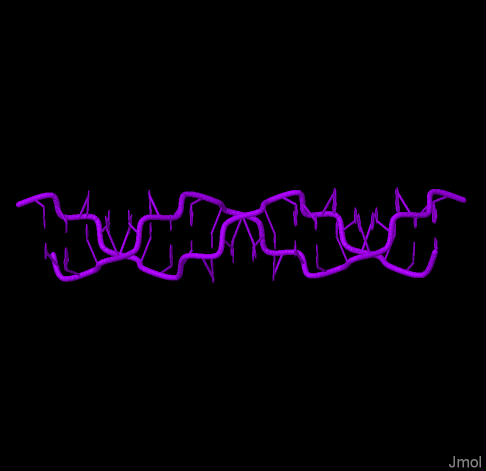 |
| Рисунок 2. А-форма | Рисунок 3. B-форма | Рисунок 4. Z-форма |
Задание 3.
Упр.1.
В приведенной ниже таблице представлены значения торсионных углов для данной тРНК (PDB ID: 1QRT)
| Strand I |
|
|
|
|
|
|
|
|
|
base |
alpha |
beta |
gamma |
delta |
epsilon |
zeta |
chi |
| 1 |
G |
--- |
107.8 |
54.4 |
78.2 |
-141.0 |
-78.5 |
-173.0 |
| 2 |
G |
-64.8 |
162.9 |
63.3 |
78.1 |
-151.7 |
-78.5 |
-168.4 |
| 3 |
G |
-47.0 |
168.7 |
42.3 |
76.6 |
-168.4 |
-55.4 |
-158.7 |
| 4 |
G |
169.4 |
175.5 |
169.7 |
85.2 |
-135.9 |
-70.9 |
-173.6 |
| 5 |
U |
-54.6 |
173.4 |
48.0 |
79.4 |
178.9 |
-76.4 |
-157.5 |
| 6 |
A |
-173.5 |
-137.5 |
159.0 |
118.9 |
-65.4 |
-77.6 |
-158.2 |
| 7 |
C |
51.8 |
177.0 |
32.4 |
88.0 |
-150.8 |
-64.8 |
-167.4 |
| 8 |
G |
-65.5 |
-172.2 |
50.1 |
81.5 |
-160.7 |
-69.3 |
-157.2 |
| 9 |
A |
-59.1 |
180.0 |
41.4 |
82.5 |
-173.6 |
-80.0 |
-146.8 |
| 10 |
G |
147.9 |
-146.9 |
175.0 |
83.8 |
-144.9 |
-54.2 |
178.5 |
| 11 |
G |
-173.6 |
158.3 |
158.3 |
84.5 |
-117.4 |
-86.9 |
176.6 |
| 12 |
U |
-54.2 |
154.5 |
51.3 |
79.7 |
-129.2 |
-72.9 |
-169.2 |
| 13 |
U |
-60.6 |
171.8 |
48.0 |
77.3 |
-136.6 |
-89.5 |
-150.8 |
| 14 |
A |
-39.1 |
176.9 |
59.6 |
82.7 |
-151.8 |
-53.7 |
179.1 |
| 15 |
U |
-69.8 |
-174.0 |
50.0 |
85.5 |
-154.9 |
-55.5 |
-161.9 |
| 16 |
U |
-76.8 |
-170.8 |
49.8 |
87.6 |
-162.7 |
-59.0 |
-165.4 |
| 17 |
C |
-83.0 |
-163.0 |
57.4 |
88.7 |
-165.6 |
-63.9 |
-159.8 |
| 18 |
C |
-64.9 |
-168.2 |
33.1 |
83.6 |
-142.1 |
-89.8 |
-159.4 |
| 19 |
G |
-50.9 |
146.4 |
53.6 |
78.8 |
-164.1 |
-65.8 |
-173.4 |
| 20 |
G |
-56.3 |
-166.5 |
44.1 |
82.7 |
-149.1 |
-82.5 |
-151.7 |
| 21 |
C |
121.7 |
-147.8 |
-164.1 |
86.9 |
-82.3 |
-152.1 |
-163.1 |
| 22 |
G |
-127.6 |
138.0 |
34.0 |
83.8 |
-152.1 |
-68.2 |
-177.4 |
| 23 |
C |
-76.7 |
-168.2 |
52.0 |
85.2 |
-155.1 |
-78.4 |
-161.2 |
| 24 |
C |
137.7 |
-166.5 |
-170.5 |
85.3 |
-129.8 |
-60.8 |
-168.2 |
| 25 |
A |
-56.5 |
171.1 |
54.9 |
85.1 |
-167.0 |
-77.9 |
-161.5 |
| 26 |
A |
-76.8 |
173.6 |
69.6 |
80.5 |
-129.0 |
-77.4 |
-166.6 |
| 27 |
G |
-50.7 |
154.8 |
56.5 |
79.5 |
140.7 |
42.6 |
-139.6 |
| 28 |
G |
-142.5 |
-96.7 |
120.3 |
111.9 |
-69.5 |
113.7 |
-104.7 |
| 29 |
U |
28,20 |
-124.9 |
-62.5 |
78.5 |
-120.9 |
-177.7 |
-112.3 |
|
|
|
|
|
|
|
|
|
| Strand II |
|
|
|
|
|
|
|
|
|
base |
alpha |
beta |
gamma |
delta |
epsilon |
zeta |
chi |
| 1 |
C |
-58.6 |
179.1 |
42.2 |
87.9 |
-146.6 |
-78.7 |
-144.4 |
| 2 |
C |
-69.8 |
-174.4 |
53.5 |
85.2 |
-150.2 |
-76.1 |
-159.3 |
| 3 |
C |
-50.6 |
171.4 |
39.8 |
83.4 |
-162.2 |
-66.0 |
-162.3 |
| 4 |
C |
-70.2 |
165.9 |
62.3 |
81.9 |
-149.0 |
-79.6 |
-158.4 |
| 5 |
A |
-66.5 |
-179.6 |
49.4 |
83.4 |
-155.6 |
-71.8 |
-150.7 |
| 6 |
U |
-61.3 |
174.7 |
49.2 |
80.9 |
-156.9 |
-71.8 |
-166.5 |
| 7 |
G |
152.7 |
-153.2 |
179.4 |
87.4 |
-138.5 |
-67.8 |
173.7 |
| 8 |
C |
-1.9 |
127.6 |
14,3 |
77.5 |
-170.3 |
-86.7 |
-155.3 |
| 9 |
U |
-144.5 |
126.1 |
141.6 |
86.6 |
-113.2 |
-105.7 |
-177.3 |
| 10 |
C |
-60.1 |
-173.2 |
48.3 |
88.9 |
-154.8 |
-46.9 |
-160.1 |
| 11 |
C |
-124.2 |
-144.6 |
65.0 |
85.0 |
-157.8 |
-70.6 |
-168.2 |
| 12 |
A |
-85.7 |
-116.8 |
60.1 |
99.1 |
7,5 |
103.0 |
-83.4 |
| 13 |
G |
168.9 |
154.2 |
-55.2 |
90.9 |
135.2 |
-84.4 |
-107.1 |
| 14 |
U |
-66.8 |
173.6 |
57.4 |
87.2 |
-120.4 |
-114.0 |
-155.4 |
| 15 |
U |
-67.3 |
-172.4 |
52.9 |
80.2 |
-154.1 |
-62.6 |
-152.9 |
| 16 |
A |
-77.0 |
-167.1 |
56.2 |
82.1 |
-154.5 |
-64.0 |
-176.5 |
| 17 |
G |
20,8 |
113.0 |
9,5 |
79.9 |
-169.4 |
-69.0 |
-169.1 |
| 18 |
G |
-145.2 |
81.9 |
152.6 |
92.0 |
-114.4 |
-128.1 |
172.7 |
| 19 |
C |
-61.9 |
176.3 |
58.0 |
85.0 |
-151.5 |
-18.6 |
-151.4 |
| 20 |
C |
-74.0 |
-174.9 |
50.7 |
80.1 |
-155.7 |
-73.9 |
-154.0 |
| 21 |
A |
-71.0 |
173.3 |
57.6 |
80.7 |
-152.5 |
-57.9 |
-165.3 |
| 22 |
C |
-80.9 |
178.2 |
65.0 |
82.8 |
-147.8 |
-58.5 |
-164.5 |
| 23 |
G |
-51.6 |
160.9 |
54.4 |
80.0 |
-150.5 |
-71.5 |
-171.8 |
| 24 |
G |
-53.6 |
158.4 |
52.3 |
78.8 |
-145.1 |
-83.8 |
-173.2 |
| 25 |
A |
-72.1 |
-157.5 |
69.4 |
86.1 |
47.7 |
153.1 |
-94.1 |
| 26 |
A |
-115.0 |
-162.8 |
-178.4 |
87.3 |
-165.4 |
-109.4 |
-151.7 |
| 27 |
C |
-31.2 |
139.1 |
62.5 |
105.3 |
-152.6 |
110.9 |
-145.6 |
| 28 |
C |
151.0 |
-172.3 |
66.9 |
82.1 |
-134.7 |
-74.6 |
179.0 |
| 29 |
U |
-86.9 |
104.0 |
47.6 |
83.3 |
-164.3 |
-132.5 |
138.2 |
С помощью программ find_pair и analyze пакета 3DNA ананлогичным способом я получила значения торсионных углов для A-, B- и Z-форм ДНК.
Сравнив разницы по модулю полученных средних значений торсионных углов с вышеуказанными значениями для структуры тРНК, я сделала вывод о том, что тяжи этой структуры наиболее похожи на Z-форму ДНК.
В таблице ниже приведены средние значения торсионных углов для рассматриваемой структуры тРНК, A-, B- и Z-форм ДНК (I, II - номер цепи, a,b,g,d,e,z,c - торсионные углы)
| tRNA |
|
|
|
|
|
|
|
|
a |
b |
g |
d |
e |
z |
c |
| I |
-38,13571429 |
20,25862069 |
49,34482759 |
84,82758621 |
-119,0344828 |
-67,63103448 |
-123,2 |
| II |
-46,70689655 |
20,99655172 |
51,18965517 |
85,20689655 |
-127,5034483 |
-57,15517241 |
-108,7896552 |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
| A |
|
|
|
|
|
|
|
|
a |
b |
g |
d |
e |
z |
c |
| I |
-51,7 |
174,8 |
41,7 |
79,1 |
-147,8 |
-75,1 |
-157,2 |
| II |
-51,7 |
174,8 |
41,7 |
79,1 |
-147,8 |
-75,1 |
-157,2 |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
| B |
|
|
|
|
|
|
|
|
a |
b |
g |
d |
e |
z |
c |
| I |
-29,9 |
136,3 |
31,1 |
143,3 |
-140,8 |
-160,5 |
-98 |
| II |
-29,9 |
136,3 |
31,1 |
143,3 |
-140,8 |
-160,5 |
-98 |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
| Z |
|
|
|
|
|
|
|
|
a |
b |
g |
d |
e |
z |
c |
| I |
-48,82105263 |
21,12 |
-61,465 |
116,25 |
-100,2368421 |
4,721052632 |
-47,8 |
| II |
-48,82105263 |
21,12 |
-61,465 |
116,25 |
-100,2368421 |
4,721052632 |
-47,8 |
Упр.3.
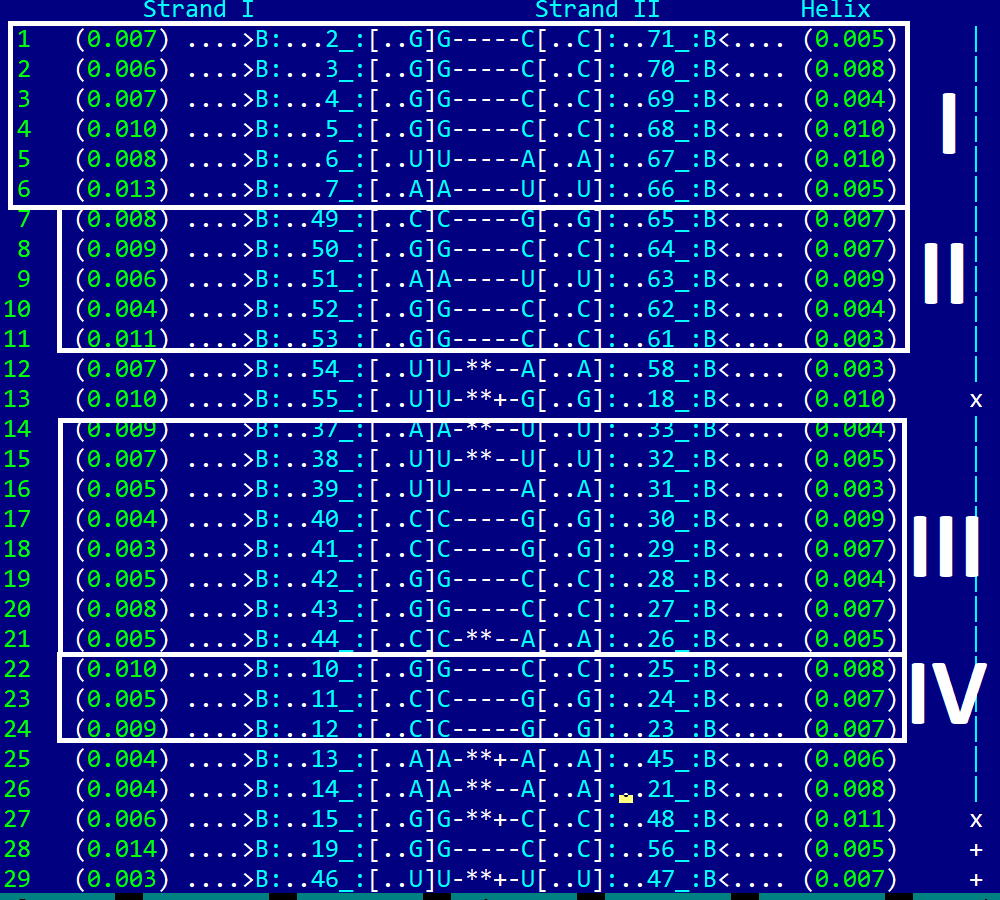
Рисунок 5. Stems structure |
В данной структуре тРНК (PDB ID: 1QRT) было обнаружено 4 стебля:
I) 2 - 7 нуклеотиды (Strand I) / 66 - 71 нуклеотиды (Strand II)
II) 49 - 53 нуклеотиды (Strand I) / 61 - 65 нуклеотиды (Strand II)
III) 37 - 44 нуклеотиды (Strand I) / 26 - 33 нуклеотиды (Strand II)
IV) 10 - 12 нуклеотиды (Strand I) / 23 - 25 нуклеотиды (Strand II)
Неканонические пары оснований:
[..U]U-**--A[..A]
[..U]U-**+-G[..G]
[..A]A-**--U[..U]
[..U]U-**--U[..U]
[..C]C-**--A[..A]
[..A]A-**+-A[..A]
[..A]A-**--A[..A]
[..G]G-**+-C[..C]
[..U]U-**+-U[..U]
В cтруктуре тРНК присутствуют участки, стабилизирующие третичную структуру посредством
дополнительных водородных связей в молекуле. В данном случае такое взаимодействие показано в строчках 28-29 на рисунке слева.
|




