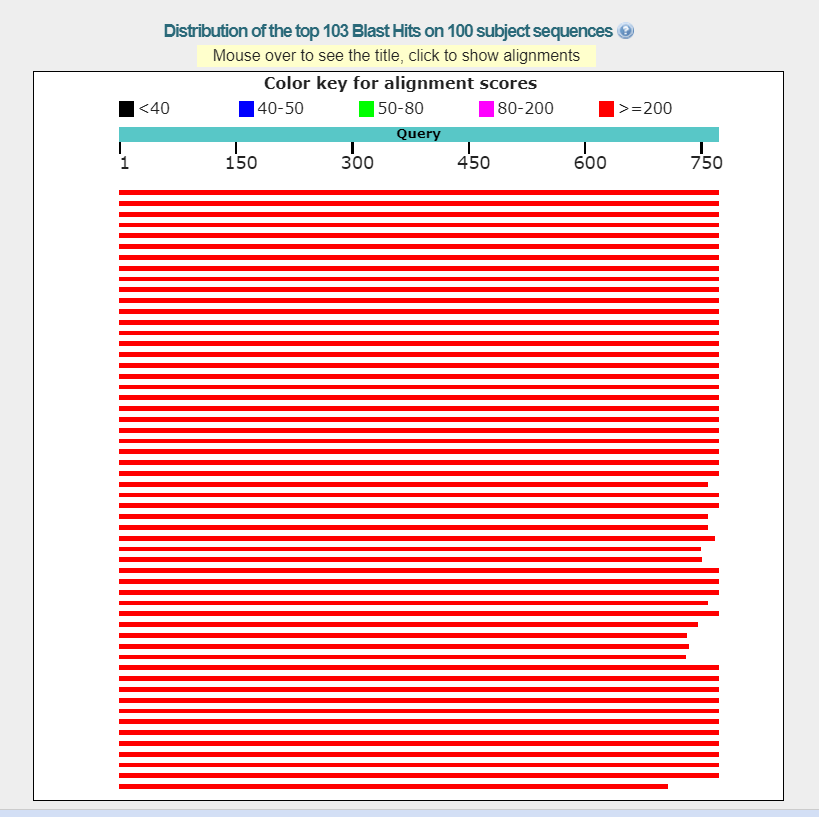
С помощью программы consambig пакета EMBOSS была получена консенсусная последовательность из выравнивания прямой и обратной последовательностей. BlastN этой последовательности выдал 100 лучших совпадений. Для определения аннотации взяты 2 лучших совпадения с Ident 98%. Выбранные записи соответствуют гену Pyramicocephalus phocarum voucher PBI-604 18S ribosomal RNA gene.
Пример верхней записи
Рис.1. Распределение гомологов на выходе BLAST
Ниже приведено лучшее выравнивание BLAST. Можно заметить, что все 16 несовпадений вызваны неопределенностью нуклеотида в прочтении хроматограммы.
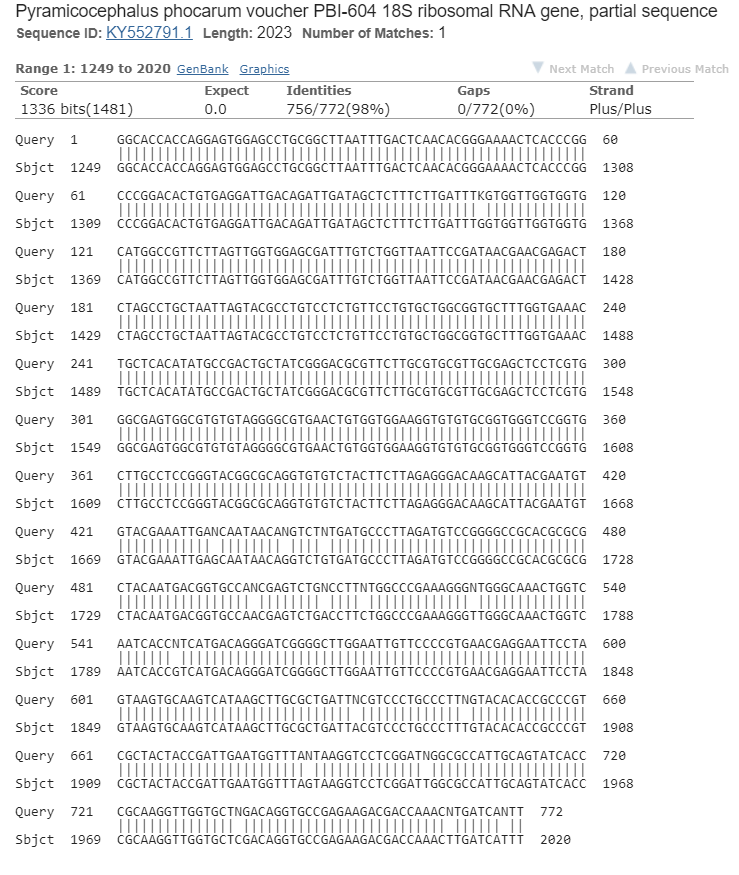
Рис.2. Лучшее выравнивание BLAST
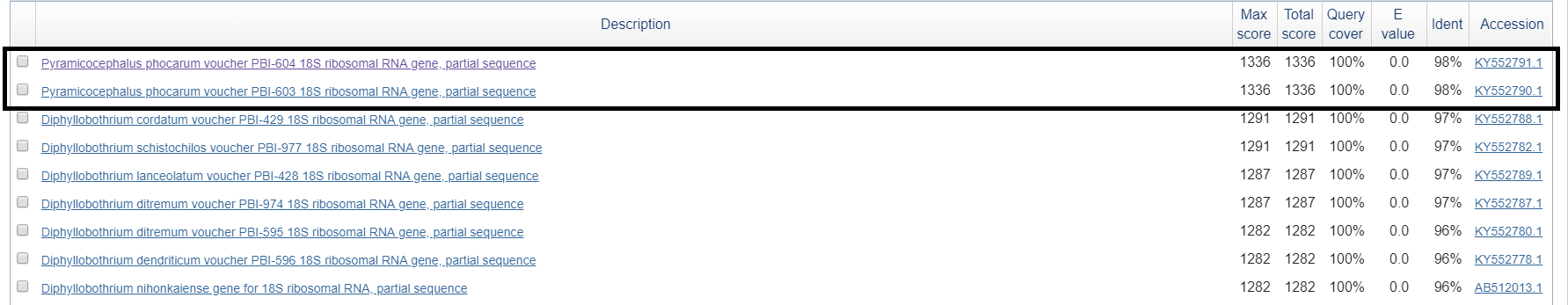
Рис.3. Фрагмент таблицы находок
Можно сказать, что нуклеотидная последовательность относится к гену, кодирующему 18S рибосомальной РНК у организма рода Pyramicocephalus, относящегося к семейству Diphyllobothriidae порядка Diphyllobothriidea.
| Алгоритм | Параметры алгоритма | Число находок | Комментарии |
| megablast | Стандартные; длина слова = 28; M/M Score 1,-2 | 2 | Поиск ограничен семейством; последняя находка с E-value = 0.0; первая = 0.0; минимальный Ident = 98%; |
| blastn | Стандартные; длина слова=11; M/M Score 2,-3 | 4 | Поиск ограничен семейством; последняя находка с E-value = 1.5; первая = 0.0; минимальный Ident = 98% |
| blastn sensitive | Длина слова=7; Score 1,-4 | 7 | Поиск ограничен семейством; последняя находка с E-value = 2.4; первая = 0.0; минимальный Ident = 98% |
Табл.1. Разница в работе вариантов blast
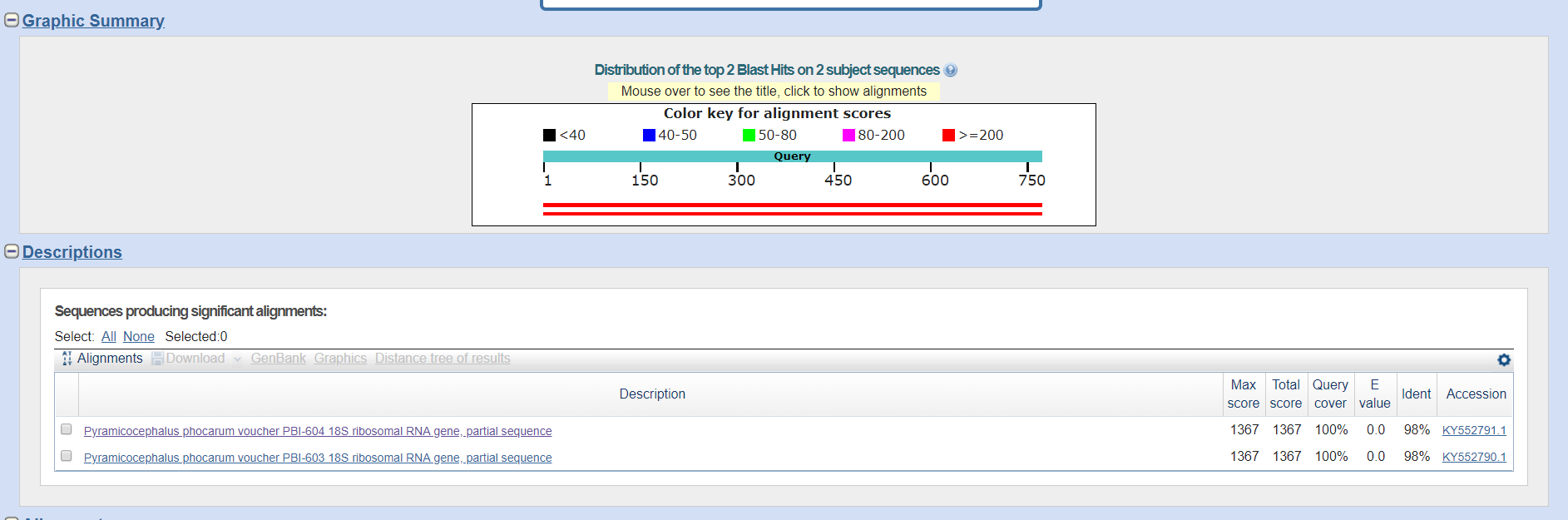
Рис.4. Результаты megablast
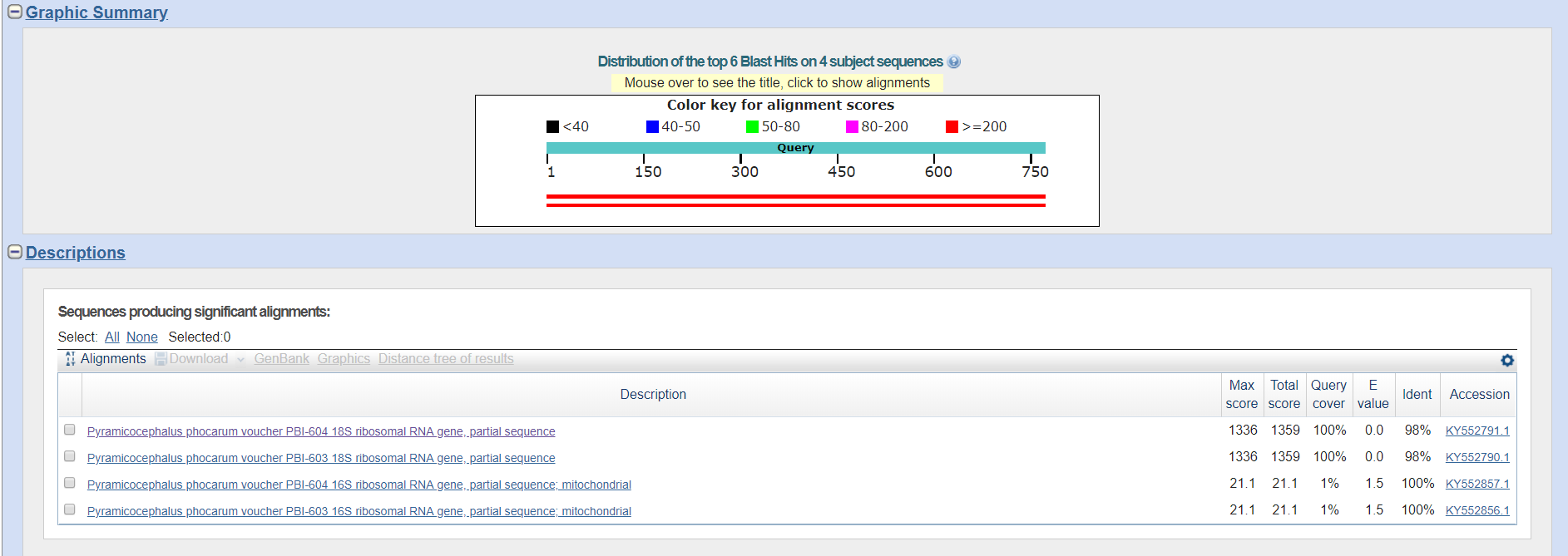
Рис.5. Результаты blastn
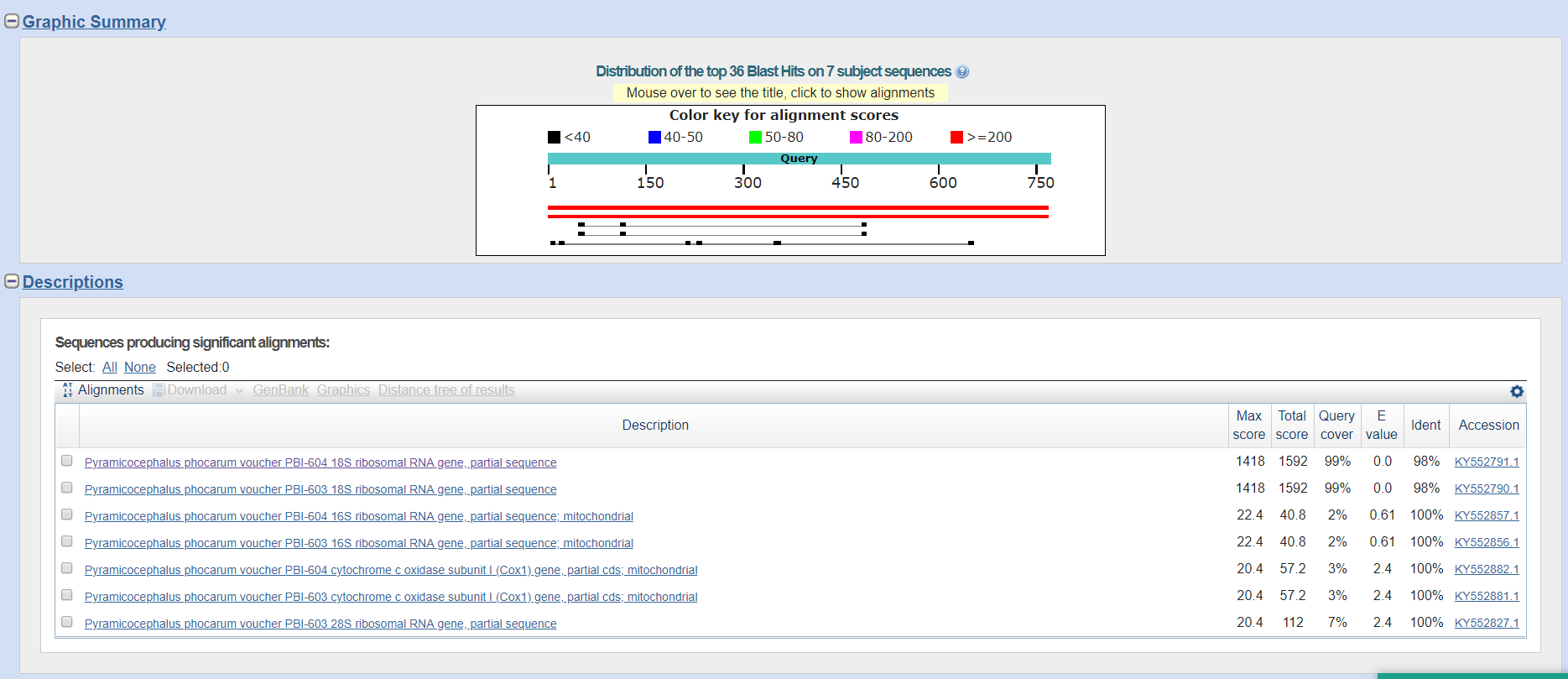
Рис.6. Результаты blastn sensitive
На основании полученных результатов можно сделать вывод о том, что Megablast работает гораздо более строго, пересекая большее количество находок и, следовательно, выдавая только последовательности, которые ближе всего к исходной. Он подходит для поиска близкородственных последовательностей, работает довольно быстро. Blastn sensitive хорошо подходит для точного выравнивания высококонсервативных участков ДНК, а не целых генов. Исходя из этого, неудивительно, чтот выдача megablast наименьшая.
Далее необходимо было провести запуски BLAST для последовательности какой-нибудь некодирующей РНК из прошлого практикума. Для выполнения этого задания я выбрала тРНК complement(237..315). Для начала я применила программы megablast, blastn (default) и blastn (sensetive) с теми же параметрами алгоритма, что и в прошлый раз, однако без ограничений по таксону. Оба варианта blastn выдали чрезмерное количество находок, в то время как megablast выдал лишь 5, 4 из которых относились к организму из семейства Parameciidae.
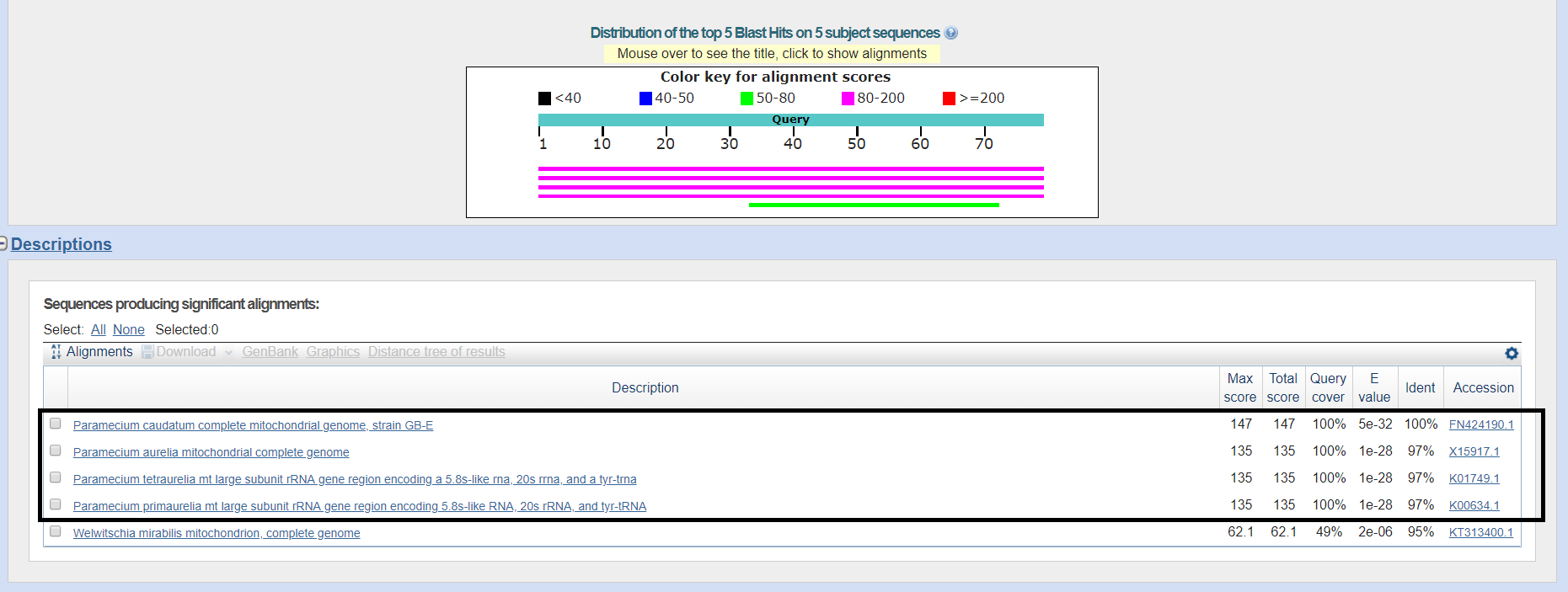
Рис.7. Результаты megablast для тРНК (1)
Тогда я решила произвести ограничение по семейству Parameciidae.
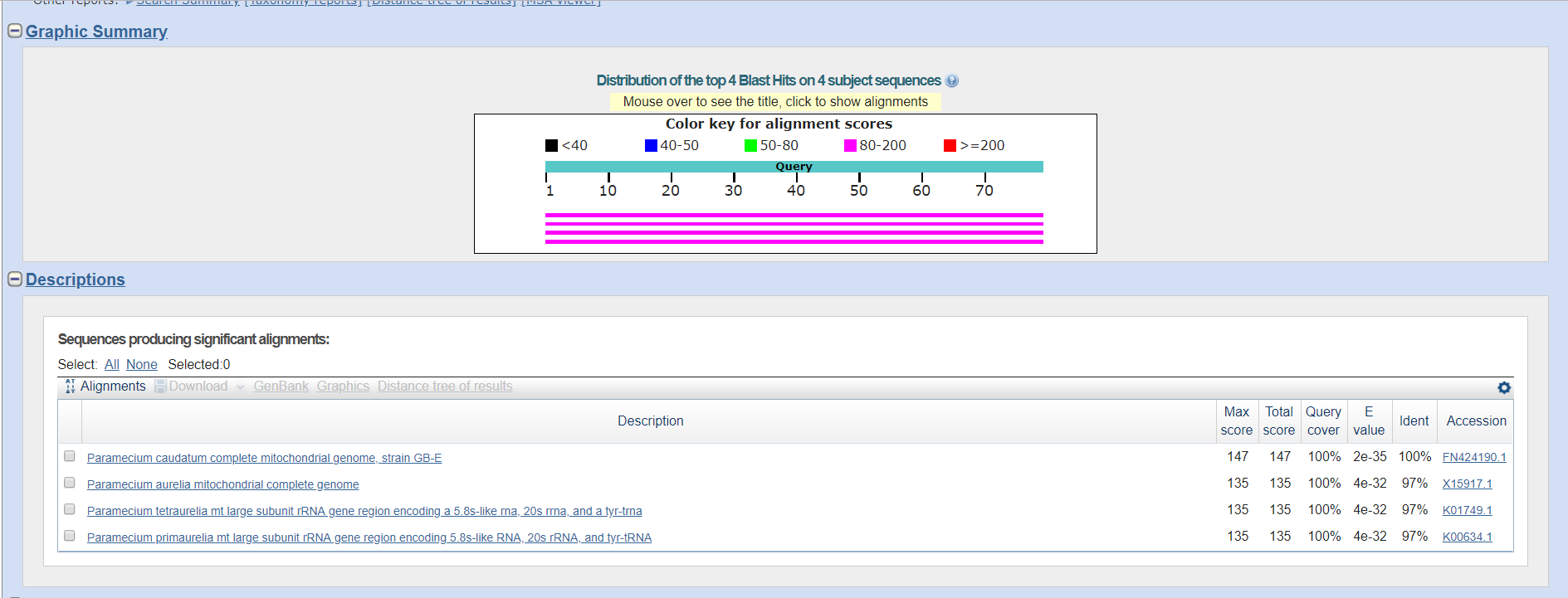
Рис.8. Результаты megablast для тРНК (2)
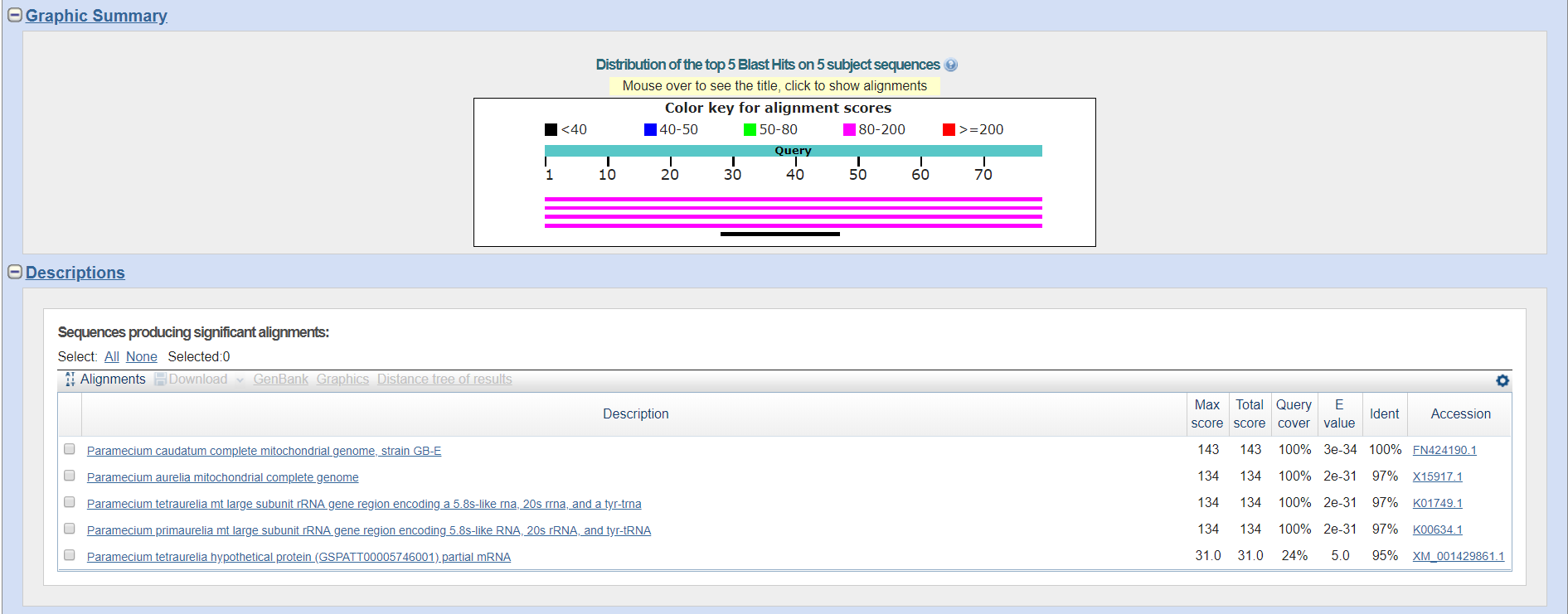
Рис.9. Результаты blastn для тРНК
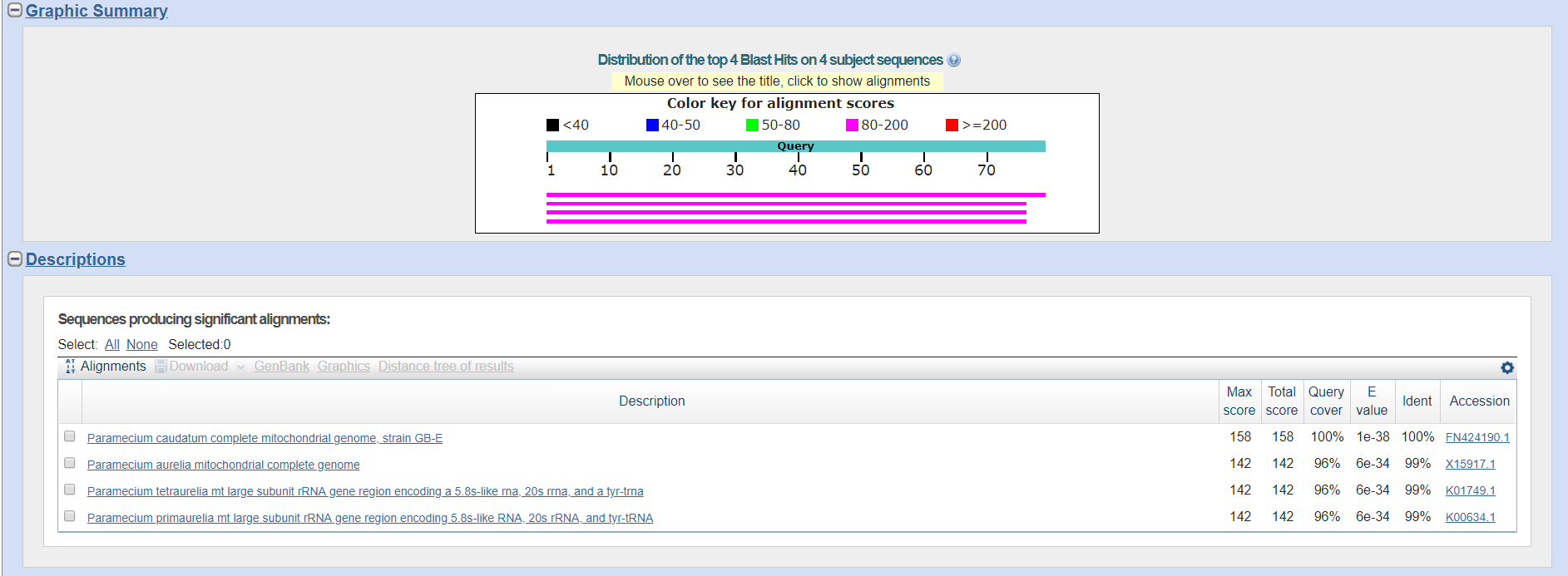
Рис.10. Результаты blastn sensitive для тРНК
Нетрудно заметить, что находки всех 3 программ совпадают, за исключением одной находки в стандартном Blastn, однако данная находка имеет E-value больше 0.
Задание выполнялось для 3 белков: HSP71_YEAST, PRPC_EMENI и TERT_SCHPO при помощи BLAST+.
В первую очередь создавалась локальная база данных:
makeblastdb -in X5.fasta -dbtype nucl
Поскольку представленные последовательности белковые, то использовался tblastn.
Запрос для HSP71_YEAST:
tblastn -query hsp71.fasta -db X5.fasta > hsp71.out
Вывод:

Рис.11. Вывод запроса для HSP71_YEAST
HSP71_YEAST кодирует Heat shock protein SSA1 у пекарских дрожжей Saccharomyces cerevisiae (strain ATCC 204508 / S288c). Белок выполняет функцию транспорта полипептидов через митохондриальную мембрану и в ЭПР. SSA1 может участвовать в ATP-зависимой разборке в везикулах, покрытых клатрином.
Выравнивание со scaffold-199 имеет E-value = 0.0 и высокий вес (920), исходя из этого, эти 2 последовательности гомологичны и, вероятно, схожи функционально.
Аналогичный бласт сделан для PRPC_EMENI и TERT_SCHPO. Ниже представлена соответствующая сводная таблица:
| TERT_SCHPO | PRPC_EMENI | |
| Кодируемый белок | trt1 | mcsA |
| Функция | Теломераза необходима для репликации хромосомных концов у большинства эукариот. Она отвечает за удлинение теломер. Это обратная транскриптаза, которая добавляет простые повторения последовательности к концам хромосом с помощью копирования последовательности шаблонов в РНК-компоненте фермента. | Митохондриальный ген. Катализирует синтез (2S, 3S) -2-метилцитрата из пропионил-СоА и оксалоацетата, а также из ацетил-СоА и оксалоацетата с большей эффективностью. Также имеет активность цитратсинтазы и может заменить недостаток активности citА. |
| Лучшее совпадение | scaffold-17, Score: 108, E-value: 1e-23 | scaffold-693, Score: 393, E-value: 6e-121 |
| Результат | Имеются короткие идентичные участки без гэпов. О чистой гомологии речи идти не может, так как низкие значения Identity (25%) и Positives (47%). | Находку можно считать условно положительной. E-value достаточно низок, чтобы утверждать о гомологии 2 белков, но вес недостаточно велик, чтобы однозначно утверждать о сходной функции. Имеются консервативные участки в 10-30 пар оснований. |
Табл.2. Гомология PRPC_EMENI и TERT_SCHPO
Поиск проводился с помощью blastx по базе данных Reference proteins, ограничив поиск таксоном Fungi. В контиге unplaced-30 длиной 45453 пар оснований был найден ген, кодирующий T-complex protein 1 subunit epsilon [Lobosporangium transversale] с E-value = 0.0.
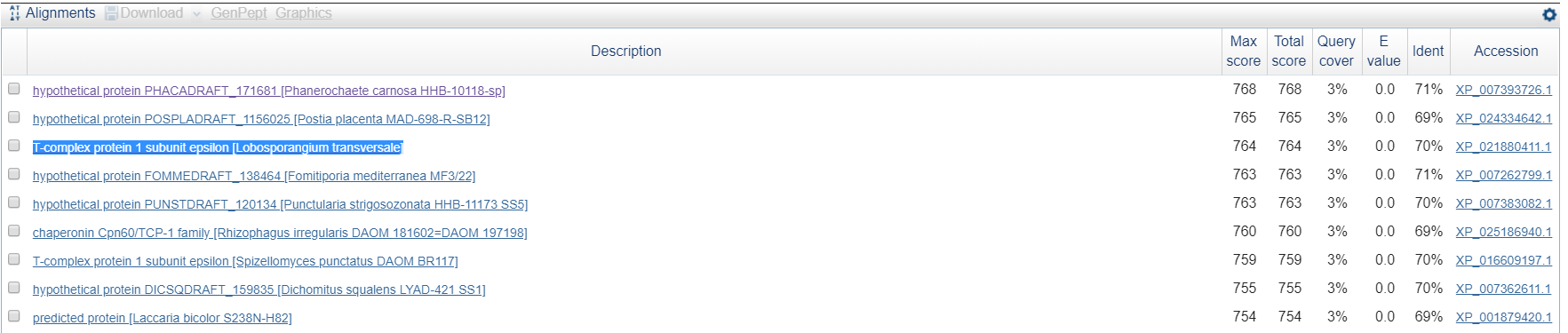
Рис.12. BlastX
Для выравнивания я взялa геномы из RefSeq 2 бактерий: Chlamydia abortus и Chlamydia avium (NC_007461.1 и NZ_CP006571.1 соответсвенно), и выравняла их с помощью Multiple Sequence Alignment blastn. Перестройки генома прокариот можно увидеть ниже; идентичные участки помечены одной буквой.
Принимая во внимание то, что геномы кольцевые, данная карта локального сходства означает довольно сильное сходство между ними, однако всё равно на карте мы можем наблюдать небольшие участки, в которых произошла делеция.
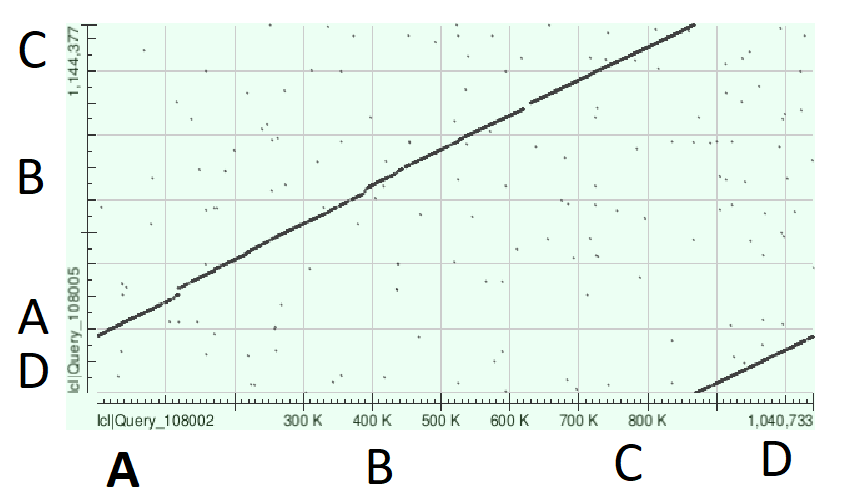
Рис.13. BlastX
© Макиевская Кьяра, 2018