BLAST
Выявление гомологии
Для поиска гомологии было взято 9 белков, которые выдавались на запрос в BLAST. По результатам их сравнения была составлена нижележащая таблица. Также, здесь есть ссылка на JalView-проект с выравниванием, наиболее содержательная часть которого лежит ниже.
Рисунок с блоками выравнивания
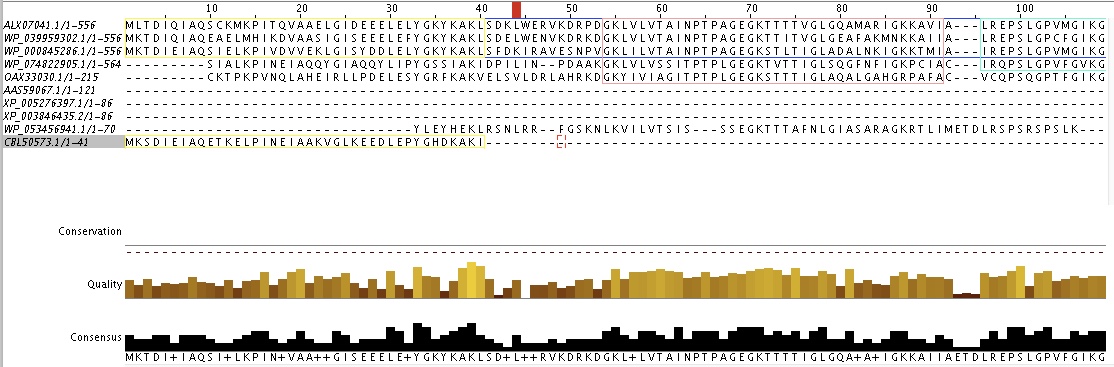
Таблица с признаками гомологии
| ID | Name | Identity, % | Coverage, % | E-value | Hom. |
| WP_039959302.1 | Formate--tetrahydrofolate ligase | 67.986 | 100 | 0.0 | + |
| WP_000845286.1 | Formate--tetrahydrofolate ligase | 62.410 | 100 | 0.0 | + |
| WP_074822905.1 | Formate--tetrahydrofolate ligase | 47.895 | 98 | 3.74e-148 | + |
| AAS59067.1 | Methylenetetrahydrofolate dehydrogenase, partial | 46.281 | 21 | 5.08e-25 | + |
| OAX33030.1 | P-loop containing nucleoside triphosphate hydrolase protein, partial | 34.419 | 29 | 1.13e-21 | + |
| XP_005276397.1 | C1-tetrahydrofolate synthase, mitochondrial-like isoform X1 | 38.372 | 15 | 1.50e-10 | + |
| XP_003846435.2 | C1-tetrahydrofolate synthase, mitochondrial-like isoform X4 | 38.372 | 15 | 1.58e-10 | + |
| CBL50573.1 | Formyltetrahydrofolate synthetase | 51.220 | 7 | 0.007 | + |
| WP_053456941.1 | Lipopolysaccharide biosynthesis protein | 40.000 | 12 | 2.7 | - |
Краткое обсуждение
В связи с нечётким заданием "гомологичности" я определял как гомологичные последовательности, которые имеют значимые блоки достоверного выравнивания, в которые входит последовательность моего белка. Так во все последовательности, кроме последней, такие блоки входили (и их сила/количество соотносилась с E-value). Блоки обозначены на рисунке рамками. Для некоторых последовательностей на рисунке их не видно (смотреть в проект).
Итак, последовательности WP_039959302.1, WP_000845286.1, WP_074822905.1 и OAX33030.1 объединены с ALX07041.1 блоком, на рисунке обозначенным розовым цветом. AAS59067.1, XP_005276397.1 и XP_003846435.2 объединены с ALX07041.1 блоком зелёного цвета, с 505 по 588 амк. Блок 1-40 также показывает гомологичностьCBL50573.1. А вот для WP_053456941.1 блоков не найдено.
Подтверждает мой вывод о негомологичности последней последовательности её высокий E-value.
Карта локального сходства
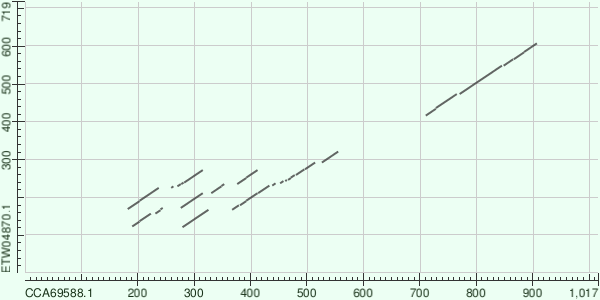
Мной были использованы последовательности с ID ETW04870.1 и CCA69588.1.
Они не являются гомологами моего белка. Даже общих доменов не имеют, так как мой белок отличается отсутствием каких-либо доменов, не принадлежащих данному семейству. При том, что все белки семейства имеют практически одинаковое строение и отличаются разве что инделами разных участков.
Так что мной был взят ZZ-домен, принадлежащий белками с цинковыми пальцами. И, соответственно, вёлся поиск белков, содержащих данный домен.
Первый белок имеет в своём составе три ZZ-домена и ещё один домен, схожий со вторым белком
Второй белок имеет в своём составе четыре ZZ-домена и вышеописанный домен.
Карта выравнивания
Интерпретиованная версия
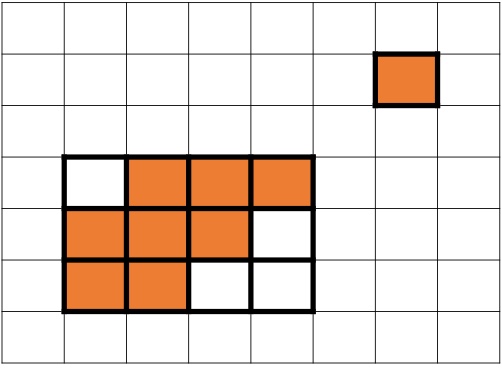
BLAST выявил сходства между белками, описанные в Pfam. Так имеется матрица 3*4, заполненная нужными нам ZZ-фрагментами, доказывающая то, что они возникли в результате дупликации и имеют сходную структуру. Ну и контрольный домен N_BRCA1_IG также имеется на карте. Однако, у белка CCA69588.1 4 домен уже имеет слабое сходство с первыми двумя доменами белка ETW04870.1, так что BLAST не нашёл сходств в тех метах. Принимая во внимание близкородствненность белков, можно предложить такую модель дупликаций: 1<-0(2)->3->4. То есть нулевой домен дуплицировался два раза, дав первый и третий (став вторым). А потом третий дал четвёртый. Но, несмотря на это, домены всё равно являются гомологичными. На интерпретации оранжевым обозначены гомологичные участки.
Для работы были использованы рекомендумемые параметры: длина слова 2 и E-value 1.0E-5. Однако, поднятие его до 0.1 ничего не изменило. Лишь при значении E-value 10 стал появляться "мусор".
