A- и В- формы ДНК. Структура РНК
Создание структур ДНК с необходимой последовательностью
На первой стадии было нужно создать три PDB файла со структурами A-, B- и Z-форм ДНК с заданной последовательностью. Здесь приложен архив с ними.
Сравнение структур 3-х форм ДНК
Далее было необходимо оценить, какие атомы тиминов смотрят в сторону малой бороздки, а какие в сторону большой. Для этого я использовал средства JMol. На рисунке ниже представлена структура основания, на которой красным цветом обозначены атомы, смотрящие в сторону большой бороздки, а синим - в сторону малой. Хочется отметить, что атомы, окрашеные в чёрный цвет, не смотрят, очевидно, в сторону бороздок. Для A-формы ДНК окраска точно такая же, а в Z-форма моё основание не встречается.
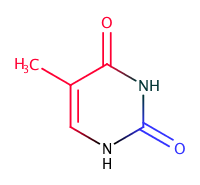
- В сторону большой бороздки обращены атомы с32.c4, c32.o4, c32.c5, c32.c7, c32.c6
- В сторону малой бороздки обращены атомы c32.c2, c32.o2
Далее представлена таблица, содержащая сравнительную характеристику данных молекул ДНК.
| A-ДНК | B-ДНК B | Z-ДНК Z | |
| Тип | Право | Право | Лево |
| Шаг | 28 A | 33,8 A | 43,5 |
| bp/виток | 11 | 10 | 12 |
| Ширина бол. бороздки | 8 A (29:B.P - 6:A.P) | 17,2 A (30:B.P - 8:A.P) | 9,9 A (11:A.P - 34:B.P) |
| Ширина мал. бороздки | 16,8 A (29:B.P - 14:A.P) | 11,7 A (29:B.P - 16:A.P) | 18,3 A (11:A.P - 28:B.P) |
Определение параметров структур нуклеиновых кислот
Определение торсионных углов
Для начала дам рисунок, показывающий расположение этих углов (рисунок взят с сайта kodomo.fbb.msu.ru ).
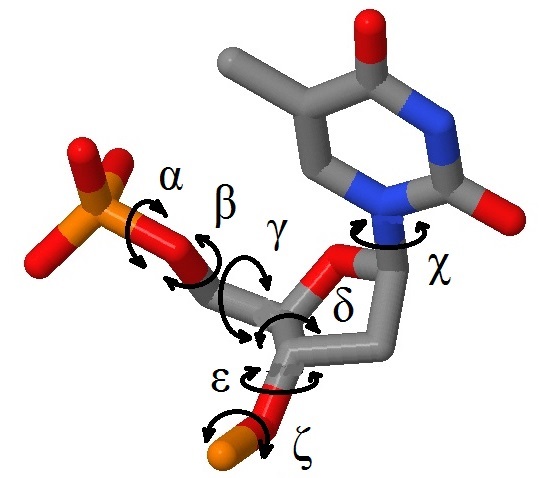
В результате использования программы analyze были получены значения торсионных углов для всех структур. Далее было произведено сравнение углов тРНК и ДНК, которое выявило наибольшее сходство между А-ДНК и тРНК (5 из 7 углов в пределах 20 градусов).
Далее представлена таблица со средними значениями торсионных углов в различных структурах. Основываясь на этих данных для tRNA мы моем найти наиболее "деформированный" нуклеотид. В данном случае это аденин в позиции 22:B. Значения его торсионных углов приведены в таблице. Он имеет наибольшие значения отклонения по двум из семи углов. Суммарное его отклонение также максимально.
| alpha | beta | gamma | delta | epsilon | zeta | chi | |
| A-DNA | -51,70 | 174,80 | 41,70 | 79,09 | -147,79 | -75,10 | -157,20 |
| B-DNA | -29,90 | 136,34 | 31,14 | 143,34 | -140,80 | -160,50 | -97,99 |
| Z-DNA | -48,82 | 21,12 | -61,47 | 116,25 | -100,24 | 4,72 | -47,80 |
| t-RNA | -36,93 | 57,02 | 59,18 | 87,08 | -110,65 | -65,58 | -133,09 |
| Adenin | -161,3 | -115,1 | 72,1 | 81,4 | 42,6 | 137,3 | -89,9 |
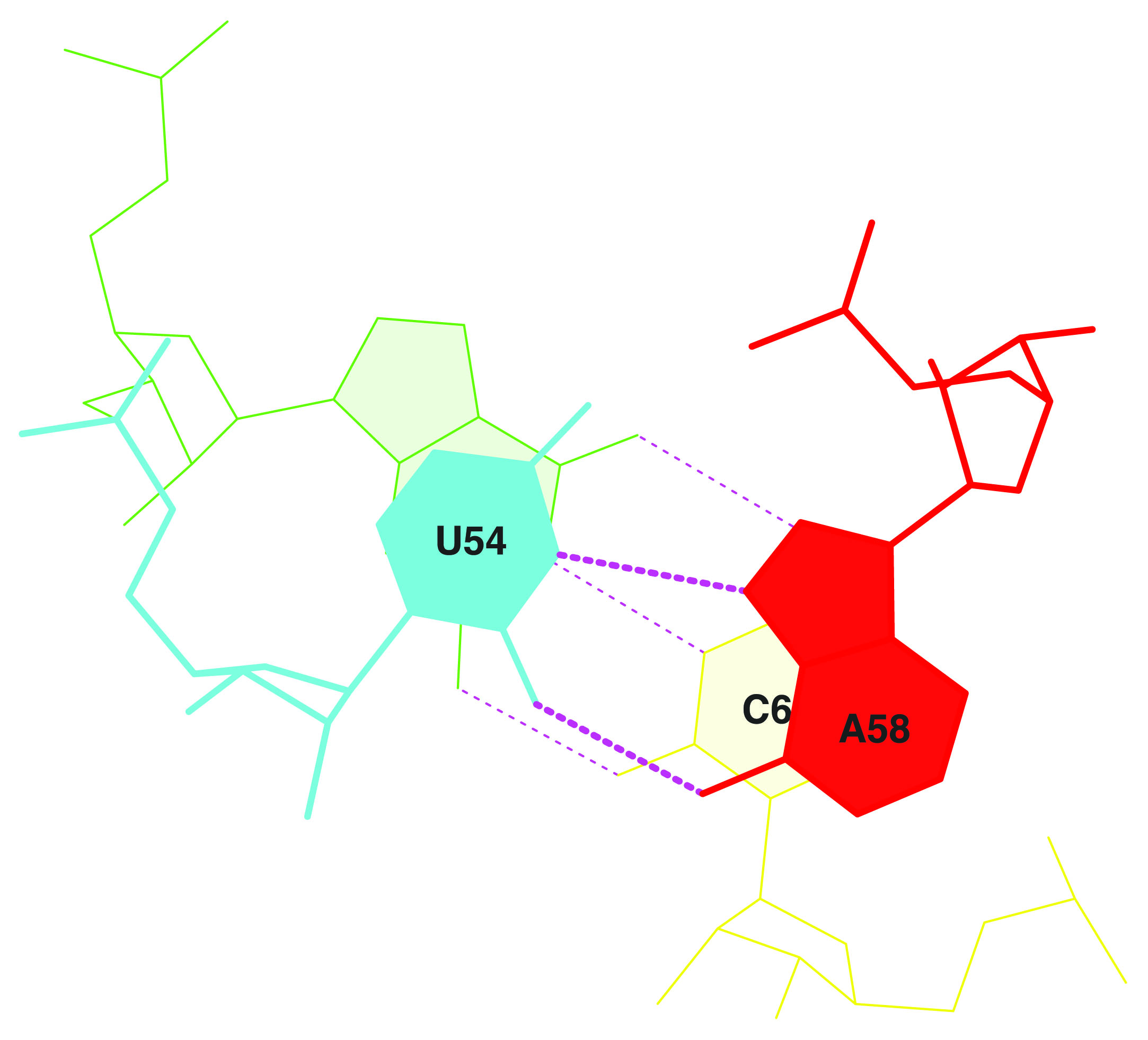
Далее было необходимо выполнить нахождение стеблей и неканонических пар оснований в структуре РНК. Эти данные также находятся в файле ".out". По ним была составлена таблица.
| Стебли | Дополнительные связи | Неканонические пары | |
| 1 | C2[G]---[C]C71 … C7[A]---[U]C66 | C54[U]---[A]C58 C55[U]---[G]C18 C13[A]---[A]C45 C14[A]---[U]C8 C15[G]---[C]C48 C19[G]---[C]C56 | C54[U]---[A]C58 C55[U]---[G]C18 C13[A]---[A]C45 C14[A]---[U]C8 C15[G]---[C]C48 C37[A]---[U]C33 C38[U]---[U]C32 C44[C]---[A]C26 |
| 2 | C49[C]---[G]C65 … C53[G]---[C]C61 | ||
| 3 | C37[A]---[U]C33 … C44[C]---[A]C26 | ||
| 4 | C10[G]---[C]C25 … C12[C]---[G]C23 |
Дополнительно был произведён анализ стекинг взаимодействий между парами соседних нуклеотидов. Наибольшую площадь перекрывания показало пересечение 11 GU/AC (11.38 A^2). Рисунок, изображающий это пересечение, был получен с помощью программы stack2img. Сравнение его с парами с наименьшей площадью перекрывания решено было не проводить, так как это не имело смысла. Однако, интересно отметить, что данная структура располагаетя в месте перехода стебля шпильки в петлю и включает в себя неканоническое взаимодействие A и U.