Прочтение последовательностей по Сэнгеру
Прочтение последовательности ДНК на основании данных, полученных из капиллярного секвенатора по Сэнгеру
Здесь приложена прямая последовательность, а здесь - обратная.
| По результатам find_pair | С помощью einverted | По алгоритму Зукера | |
| Акцепторный стебель | 6 | 6 | 6 |
| D-стебель | 3 | 0 | 3 |
| T-стебель | 5 | 0 | 5 |
| Антикодоновый стебель | 8 | 8* | 5 |
Видно, что любая из программ с точностью предсказывает акцепторный стебель, так как он наиболее очевиден и не имеет неканонических пар. D- и T-стебли не предсказываются с помощью einverted, так как антикодоновый и акцепторный стебель более существенны, а программа не умеет искать спаривания, расположенные на одной цепи. Для нахождения этих спариваний можно попытаться разделить последовательность на 4 части, но это сильно усложняет работу. Также антикодоновый стебель был найден со сдвигом на одну пару оснований. Не было идентифицировано неканоническое спаривание 44-26, зато было найдено отсутствующее в структуре спаривание 36-34. Для работы программы использовались стандартные параметры, за исключением Match score, которому было придано значение 100. Параметры были подобраны со второго раза после недолгих раздумий, так как первый был пущен со стандартными параметрами. Дальнейшие попытки что-то улучшить не давали изменений в сторону большей точности.
Далее были построены структуры по алгоритму Зукера. Видно, что он нашёл все спаривания, которые были. Однако, антикодоновая шпилька оказалась короче с обеих сторон. Судя по всему, это связано с тем, что с одной из сторон последовательность прерывается неканонической парой, а другая нестабильна без белковой части. Изменение параметров не помогло улучшить структуру, однако, удалось пронаблюдать изменения структуры при изменении температуры с 1 до 100 градусов по Цельсию.
Поиск ДНК-белковых контактов в заданной структуре
Далее было необходимо оценить, какие атомы тиминов смотрят в сторону малой бороздки, а какие в сторону большой. Для этого я использовал средства JMol. На рисунке ниже представлена структура основания, на которой красным цветом обозначены атомы, смотрящие в сторону большой бороздки, а синим - в сторону малой. Хочется отметить, что атомы, окрашеные в чёрный цвет, не смотрят, очевидно, в сторону бороздок. Для A-формы ДНК окраска точно такая же, а в Z-форма моё основание не встречается.
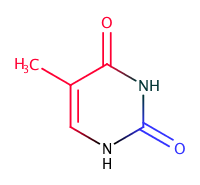
- В сторону большой бороздки обращены атомы с32.c4, c32.o4, c32.c5, c32.c7, c32.c6
- В сторону малой бороздки обращены атомы c32.c2, c32.o2
Далее представлена таблица, содержащая сравнительную характеристику данных молекул ДНК.
| A-ДНК | B-ДНК B | Z-ДНК Z | |
| Тип | Право | Право | Лево |
| Шаг | 28 A | 33,8 A | 43,5 |
| bp/виток | 11 | 10 | 12 |
| Ширина бол. бороздки | 8 A (29:B.P - 6:A.P) | 17,2 A (30:B.P - 8:A.P) | 9,9 A (11:A.P - 34:B.P) |
| Ширина мал. бороздки | 16,8 A (29:B.P - 14:A.P) | 11,7 A (29:B.P - 16:A.P) | 18,3 A (11:A.P - 28:B.P) |
Определение параметров структур нуклеиновых кислот
Определение торсионных углов
Для начала дам рисунок, показывающий расположение этих углов (рисунок взят с сайта kodomo.fbb.msu.ru ).
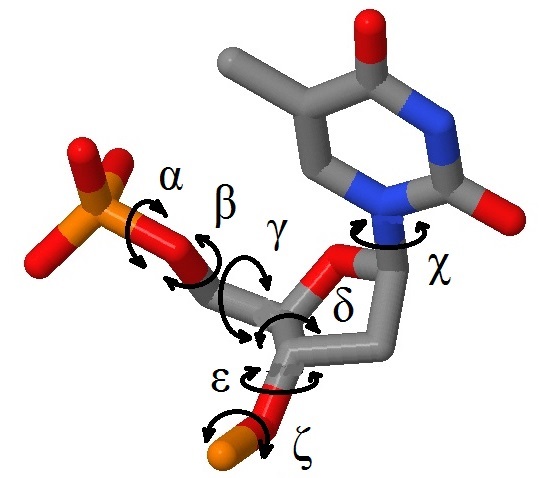
В результате использования программы analyze были получены значения торсионных углов для всех структур. Далее было произведено сравнение углов тРНК и ДНК, которое выявило наибольшее сходство между А-ДНК и тРНК (5 из 7 углов в пределах 20 градусов).
Далее представлена таблица со средними значениями торсионных углов в различных структурах. Основываясь на этих данных для tRNA мы моем найти наиболее "деформированный" нуклеотид. В данном случае это аденин в позиции 22:B. Значения его торсионных углов приведены в таблице. Он имеет наибольшие значения отклонения по двум из семи углов. Суммарное его отклонение также максимально.
| alpha | beta | gamma | delta | epsilon | zeta | chi | |
| A-DNA | -51,70 | 174,80 | 41,70 | 79,09 | -147,79 | -75,10 | -157,20 |
| B-DNA | -29,90 | 136,34 | 31,14 | 143,34 | -140,80 | -160,50 | -97,99 |
| Z-DNA | -48,82 | 21,12 | -61,47 | 116,25 | -100,24 | 4,72 | -47,80 |
| t-RNA | -36,93 | 57,02 | 59,18 | 87,08 | -110,65 | -65,58 | -133,09 |
| Adenin | -161,3 | -115,1 | 72,1 | 81,4 | 42,6 | 137,3 | -89,9 |
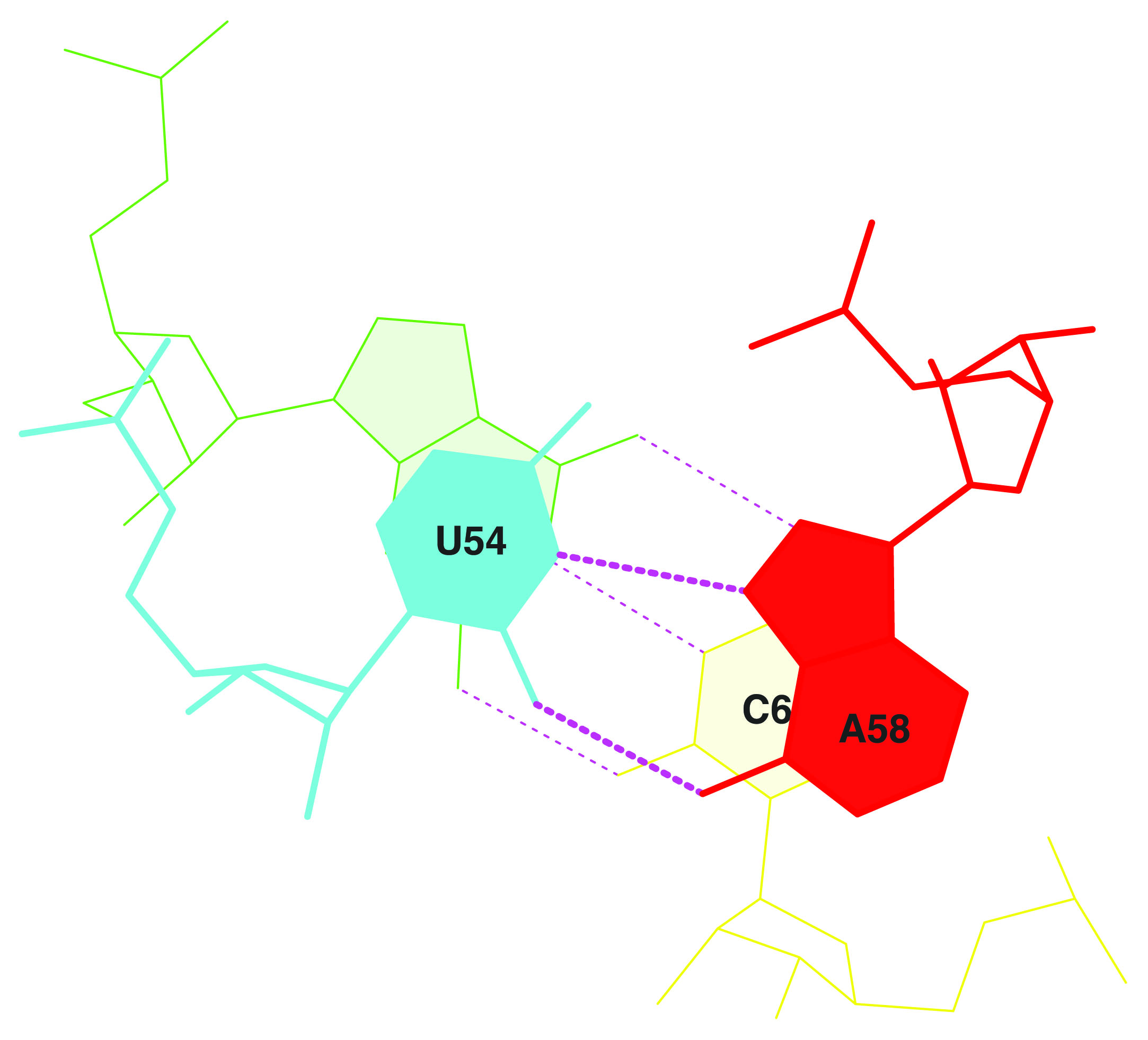
Далее было необходимо выполнить нахождение стеблей и неканонических пар оснований в структуре РНК. Эти данные также находятся в файле ".out". По ним была составлена таблица.
| Стебли | Дополнительные связи | Неканонические пары | |
| 1 | C2[G]---[C]C71 … C7[A]---[U]C66 | C54[U]---[A]C58 C55[U]---[G]C18 C13[A]---[A]C45 C14[A]---[U]C8 C15[G]---[C]C48 C19[G]---[C]C56 | C54[U]---[A]C58 C55[U]---[G]C18 C13[A]---[A]C45 C14[A]---[U]C8 C15[G]---[C]C48 C37[A]---[U]C33 C38[U]---[U]C32 C44[C]---[A]C26 |
| 2 | C49[C]---[G]C65 … C53[G]---[C]C61 | ||
| 3 | C37[A]---[U]C33 … C44[C]---[A]C26 | ||
| 4 | C10[G]---[C]C25 … C12[C]---[G]C23 |
Дополнительно был произведён анализ стекинг взаимодействий между парами соседних нуклеотидов. Наибольшую площадь перекрывания показало пересечение 11 GU/AC (11.38 A^2). Рисунок, изображающий это пересечение, был получен с помощью программы stack2img. Сравнение его с парами с наименьшей площадью перекрывания решено было не проводить, так как это не имело смысла. Однако, интересно отметить, что данная структура располагаетя в месте перехода стебля шпильки в петлю и включает в себя неканоническое взаимодействие A и U.