| Команда | Что делает |
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240361.fastq trimSRR.fastq ILLUMINACLIP:adapters.fa:2:7:7 | Удаление адаптеров, которые могли бы быть в прочтении |
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 trimSRR.fastq finaltrimSRR.fastq TRAILING:20 MINLEN:32 | Удаление плохих букв с концов чтений, остаются только чтения длиной не менее 32 |
| velveth velveth31 31 -fastq -short finaltrimSRR.fastq | Подготовление k-меров длины k=31. -short: короткие непарные чтения.velveth31: название выходной директории. |
| velvetg velveth31 | Используя файлы директории velveth31, полученной предыдущей командой, происходит сборка k-меров. |
| Название файла | Размер файла, мб |
| SRR4240361.fastq (исходные риды) | 734 |
| trimSRR.fastq (очищенные от адаптеров) | 730 |
| finaltrimSRR.fastq | 686 |
Выдача velvetg: "Final graph has 477 nodes and n50 of 25683, max 49238, total 668931, using 0/6834335 reads"
| Контиг | Длина | Покрытие |
| 6 | 49238 | 26.660851 |
| 2 | 45555 | 26.450466 |
| 34 | 43866 | 23.514977 |
Было произведено выравнивание трех самых длинных контигов и хромосомы Buchnera aphidicola с помощью megablast.
Были получены карты локального сходства и проанализированы выравнивания.
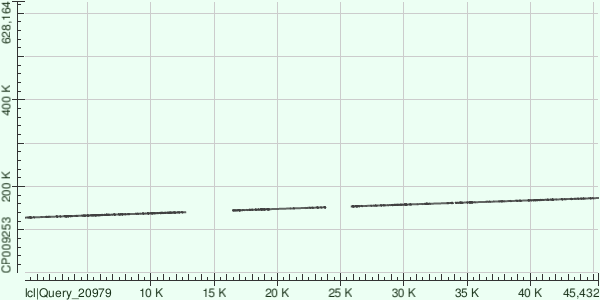
Range: 127825 to 140555 Score Expect Identities Gaps Strand 5465 bits(2959) 0.0 9751/13010(75%) 548/13010(4%) Plus/Plus
Выравнивание не очень хорошее, имеется 548 гэпов и 2711 полиморфизмов, причём выровнен не весь контиг, а его 80%.
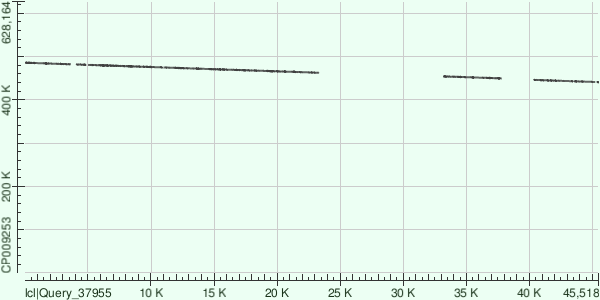
Range: 467412 to 474667 Score Expect Identities Gaps Strand 4047 bits(2191) 0.0 5691/7389(77%) 208/7389(2%) Plus/Minus
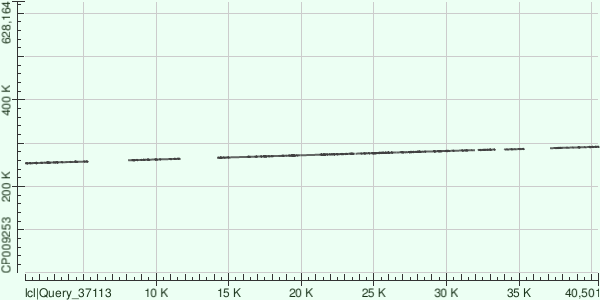
Range: 266073 to 275551 Score Expect Identities Gaps Strand 6154 bits(3332) 0.0 7611/9660(79%) 361/9660(3%) Plus/Plus
Как видно, все выравнивания довольно плохие. Контиг 2, в отличие от двух других, вообще лёг на обратную цепь.
Можно предположить, что контиги соответсвуют не Buchnera aphidicola, а какой-нибудь другой бактерии того же рода.