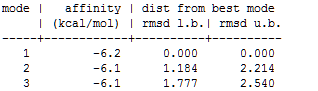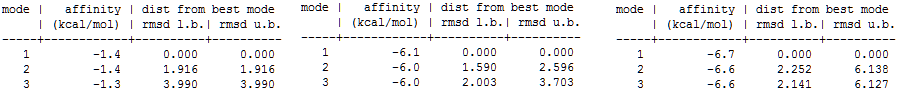Докинг низкомолекулярных лигандов в структуру белка.
1. В PDB банке нашла SMILES нотацию для NAG:
O=C(N[C@@H]1[C@@H](O)[C@H](O)[C@H](O[C@H]1O)CO)C
C помощью программ obgen и babel получила 3D структуру NAG:
obgen nag.smi > nag.mol
babel -imol nag.mol -opdb nag.pdb -xk "PM6"
2. Воспользовавшись скриптом prepare_ligand4.py из пакета Autodock tools, создала pdbqt файл лиганда.
3. Скриптом prepare_receptor4.py из пакета Autodock tools создала pdbqt файл моего белка (LYSC_BPAPS).
4. Это входные файлы для докинга. Теперь надо создать файл с параметрами докинга vina.cfg. Для докинга
необходимо указать область структуры белка в которой будет происходить поиск места для связывания. Удобно его задать как куб с неким центором.
Координаты центра определила из модели комплекса, которую построила на прошлом занятии. Выбрала С8B атом сахара, который, как мне кажется, находится в центре сайта
связывания и из текста pdb файла извлекла его координаты:
center_x=38.311
center_y=38.461
center_z=20.053
size_x = 25
size_y = 25
size_z = 25
num_modes = 20
5. Теперь можно запустить докинг:
vina --config vina.cfg --receptor seq3.pdbqt --ligand nag.pdbqt --out nag_seq3.pdbqt --log nag_seq3.log
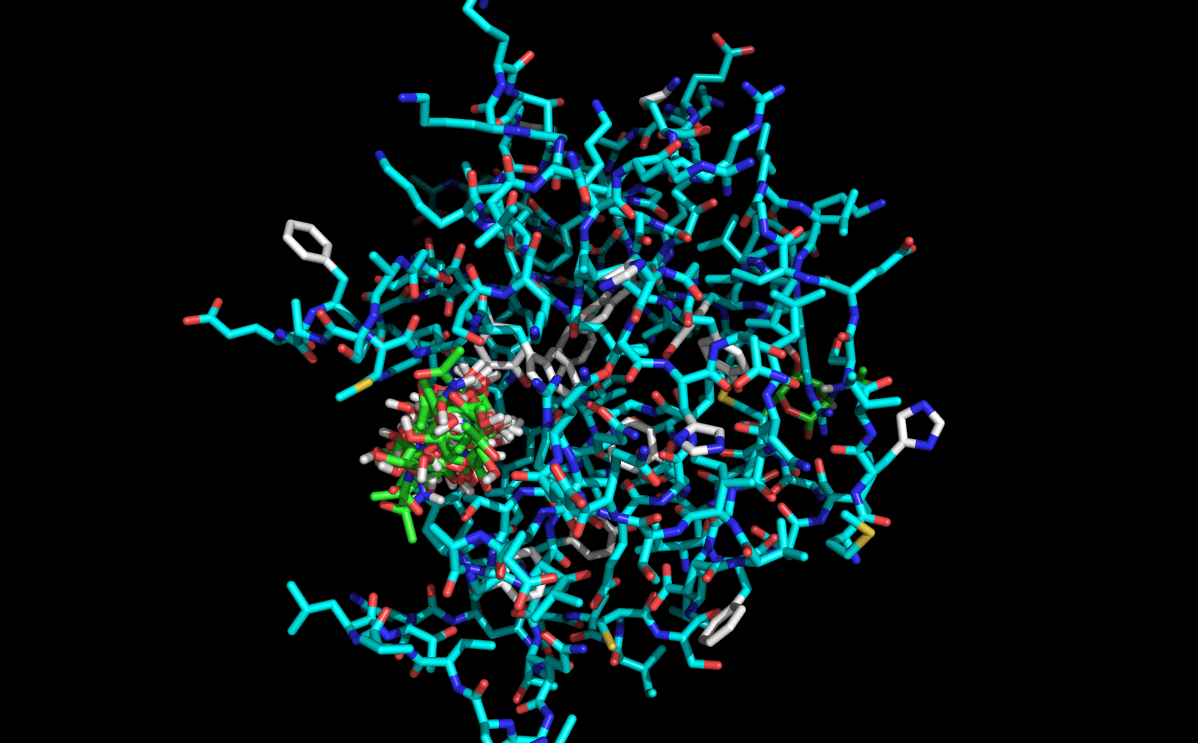
Получили такие состояния лиганда с белком:
5. Из файла выдачи nag_allni.log взяла 3 лучших расположения и геометрическую разницу между ними.
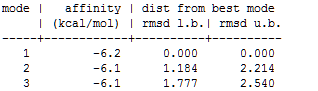
6.Теперь проведём докинг рассматривая подвижность некоторых боковых радикалов белка. Сначала разбила белок
на две части, подвижную и неподвижную. Для подвижной части выберала 3 аминокислоты, которые я использовала в прошлом задании
для позиционирования лиганда. Теперь запустим докинг:
prepare_flexreceptor4.py -r allni.pdbqt -s GLN49_ASN103
vina --config vina.cfg --receptor seq3_rigid.pdbqt --flex seq3_flex.pdbqt --ligand nag.pdbqt --out vina_1_flex.pdbqt --log vina_1_flex.log
Получили такие состояния лиганда с белком:
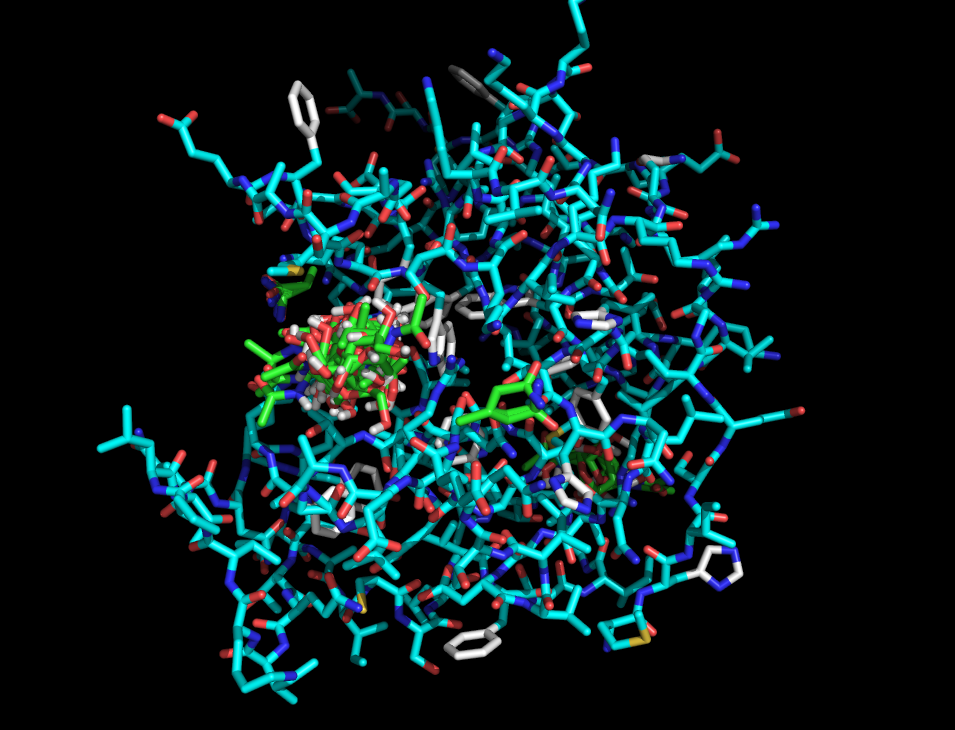
7. 3 лучших расположения и гемоетрическая разница между ними:
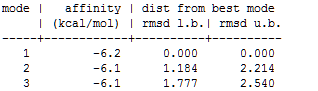
8. NAG содержит в себе СH3C(=O)NH группу. Создала 3 лиганда где метильный радикал этой группы заменён на OH, NH2, H. Для каждого из этих лигандов я провела
обыкновенный докинг. Результаты для каждого лиганда представлены ниже:

Результаты идут по порядку: H, OH, NH2, PH.
Далее был проведен докинг, учитывая подвижность боковых радикалов белка:
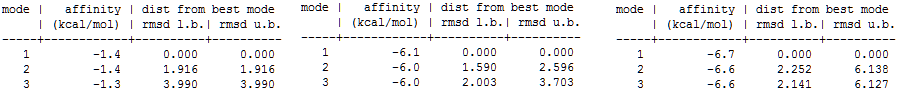
Результаты идут по порядку: OH, NH2, PH.
Если судить по состояниям докинга лигандов в Pymol и по наилучшей афинности, то можно сказать, что связывание лиганда с Ph наилучшее.
© Свистунова Даша