Практикум 3.
Статью про свой белок, ARSC_Bacsu (1JL3), я нашла в открытом доступе - Bacillus subtilis arsenate reductase is structurally and functionally similar to low molecular weight protein tyrosine phosphatases.
После выравнивания с помощью BLAST мною было выбрано 5 белков, 1LJL, 3T38, 4EGS, 2FEP, 2P4U, из разных организмов - Mus musculus, Escherichia coli, Thermoanaerobacter tengcongensis, Staphylococcus aureus, Corynebacterium glutamicum.
Таблица с различными данными об этих белках.
Из статьи о моем белке я извлекла следующие данные -
разрешение структуры 1.6 A
R-фактор = 0.226, R-free фактор = 0.241
Измерено 74177 структурных факторов
Из этих структурных факторов 91.6% (67937) имеют силу сигнала больше 3.
Фазовая проблема решена совмещением методов мультиволнового аномального рассеяния и изоморфного замещения.
Источники:
Статья
RCSB PDB
EDS
Авторы статьи обнаружили, что арсенат редуктаза бактерии структурно очень похожа на бычью фосфатазу, особенно в активном цетре, хотя последовательности белков значительно различаются.
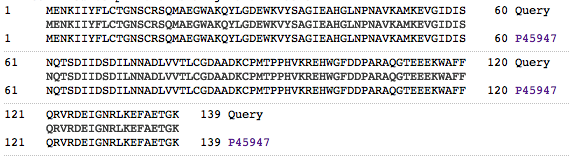
Последовательность белка, взятая из PDB, совпала на 100% с последовательностью белка с Uniprot. К сожалению, программа JalView напрочь отказалась запускаться на моем ноутбуке, поэтому здесь я представляю просто выравнивание с Uniprot. Когда та программа наконец согласится поработать, я поменяю картинку на более соответствующую заданию.
© Свистунова Дарья

