
Выданная мне плазмида CP011283 состоит из 58197 пар нуклеотидов и содержит 3,042 генов (из них белок-кодирующих - 2,950).
Enterococcus faecium — вид бактерий рода энтерококков, входящий в состав нормальной микрофлоры пищеварительного тракта человека, а также некоторых млекопитающих. Энтерококки — молочнокислые грамположительные бактерии, не образующие спор и капсул, факультативные анаэробы (способные использовать энергию брожения и поэтому, жить и при больших и при ничтожных количествах кислорода). Оптимальная температура культивирования энтерококков +35…+37 °С. Энтерококки осуществляют метаболизм бродильного типа, ферментируют разнообразные углеводы с образованием в основном молочной кислоты, но не газа, снижая кислотность среды до 4,2-4,6 pH. Энтерококки высокорезистентны к различным факторам внешней среды и дезинфицирующим средствам, могут длительное время сохранять жизнеспособность на предметах домашнего обихода, выдерживают нагревание до 60 °С в течение 30 минут. Enterococcus faecium заселяет кишечник человека в первые дни жизни. Заселение энтерококками происходит более активно у детей, находящихся на грудном вскармливании.[1]
С помощью EMBOSS (seqret embl:CP014283 CP011283.fasta -feature; seqret embl:CP014283 CP011283.gff -feature) была скачана последовательность в форматах gff и fasta.
Параметр -feature позволяет записать в файлы особенности.
Далее была запущена программа Prodigal и последовательность была переведена в формат sco.

-i (input_file) - последовательность на входе, -o (output_file) - последовательность на выходе, -f - определяет формат результата.
С помощью скриптов (1, 2) на языке python файлы CP011283.gff и CP011283.sco были приведены к более удобному виду: CP011283_new.txt (из xxx.gff), CP011283_new.sco (из xxx.sco) - были выделены ориентация гена, координаты его начала и конца.
Затем для сравнения предсказания Prodigal и данных GenBank был написан скрипт на языке python. Общий ход рассуждений и алгоритм действий можно посмотреть в комментариях к скрипту.
| Совпадающие гены | Несовпадения по N концу | Несовпадения по C концу | Несовпадающие гены | |
| Число | 55 | 4 | 1 | 3 |
| Процент,% | 87.302% | 6.349% | 1.587% | 4.762% |
Prodigal в целом предсказал гены достаточно верно: всего 4 несовпадения.
Рассмотрим несовпадение только по N-концу. В файле gff указано "20815 22713 +", а предсказано программой "20797 22713 +".
Последовательность ДНК с 20815 по 22713:
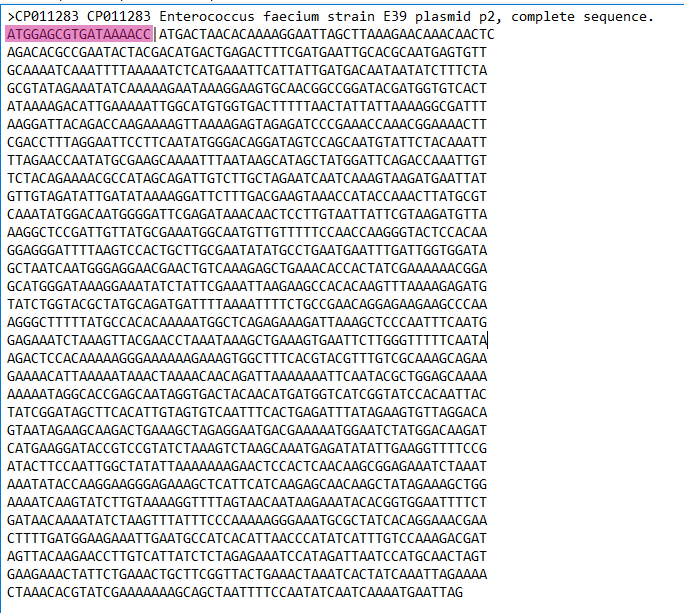
Лишние нуклеотиды, ложно указанные в гене Prodigal, отмечены цветом. Как видно в начале этой последовательности есть стартовый кодон ATG. Думаю, это и полужило причиной ошибки.
Т.к. несовпадений по С-концу нет, рассмотрим еще одно несовпадение только по N-концу. В файле gff указано "22830 23630 +", а предсказано программой "22839 23630 +".
Последовательность ДНК с 22830 по 23630:

Нуклеотиды, не указанные в гене, предсказанном Prodigal, отмечены голубым цветом. При трансляции генов данного генома используется таблица 11, которая предполагает наличие нескольких старт-кодонов. Скорее всего, ошибки предсказания связаны с этим. Как видно в начале последовательности, ошибочно принятой Prodigal за ген, есть стартовый кодон ATG, хотя настоящий стартовый кодон в данном случае TTG.
© Кучеренко Варвара 2015